 |
Bioinformatik-Praktikum
Modul Protein-Strukturvorhersage
|
 |
 |

Ergebnisse
TO257
Im Folgenden werden wir die Ergebnisse unserer Arbeit zum Target T0257 darlegen.
Festzuhalten ist, dass sich die Suche nach homologer Strukturinformation zum Target TO257 als äußerst schwierig erwiesen hat.
Gegen Ende des Praktikums erfuhren wir von weiteren Möglichkeiten der Homologie-Suche. Die Tools Superfamily und ProtoNet brachten erstaunlicherweise doch noch eventuelle Homologien zu Tage (1ugi,1qje). Hiervon verwarfen wir allerdings 1qje wieder, da der Ermittlungsprozess nicht reproduziert werden konnte.
Sogleich versuchten wir eine Tertiärstruktur mit dem Programm Modeller zu berechnen, doch wollte uns dies nicht gelingen. Das Skript konnte nicht zum Laufen gebracht werden und auch professionelle Hilfe durch die Praktikumsbetreuer vermochte nicht bei der Lösung des Problems zu helfen. Selbst wenn es gelungen wäre, so ist das ganze Vorhaben für das Target T0257 auf Grund der vielen negativ-Ergebnisse sowieso als zweifelhaft einzustufen.
Um uns dennoch ein Bild von unserem Protein machen zu können, nutzten wir Stichproben der Strukturinformationen bereits vorhandener CASP6 pdb-Daten verschiedener Server, die auf der CASP6 Seite aufgeführt sind.
Dabei achteten wir auf Vergleichbarkeit der Daten, indem nur möglichst gleichlange Sequenzen betrachtet wurden.
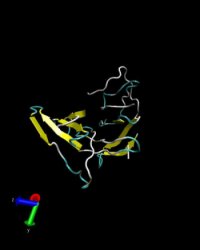
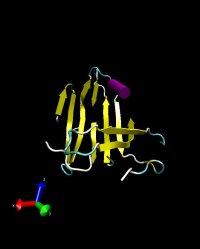
Wie zu sehen ist, gleichen sich die Strukturen kaum. Es sind keine markanten Ähnlichkeiten zu erkennen.
Deshalb schließen wir, dass es momentan nur unzureichend möglich ist, signifikante Ergebnisse durch
homologiebasierte Strukturvorhersage zum Target T0257 zu erhalten.
Festzuhalten ist, dass sich die Suche nach homologer Strukturinformation zum Target TO257 als äußerst schwierig erwiesen hat.
- rpsblast ermittelte duf796, uns war es jedoch nicht möglich, entsprechende pdb-files zu finden.
- FUGUE fand keinen einzigen signifikanten Hit.
- Eine blastp-Suche mit der Einschränkung auf Hits, zu denen auch ein pdb-file vorlag, brachte
auf Grund bescheidener eValues
keine homologe Strukturinformation.
Der manuelle Abgleich der Ergebnisse einer uneingeschränkten Anfrage brachte ebenfalls keine auffälligen, in der Ergebnis-Liste immer wiederkehrenden Proteingruppen, und damit keine Homologien. - Das gleiche Prinzip (die manuelle Analyse der Hits) versuchten wir nochmals mit dem Programm PredictProtein, doch blieben auch hier unsere Versuche, Homologien zu erkennen, erfolglos.
- nnPredict scheidet aus, da Motive wie HHEEH unwahrscheinlich sind.
- Ähnlich schwach präsentiert sich PSIPRED
Gegen Ende des Praktikums erfuhren wir von weiteren Möglichkeiten der Homologie-Suche. Die Tools Superfamily und ProtoNet brachten erstaunlicherweise doch noch eventuelle Homologien zu Tage (1ugi,1qje). Hiervon verwarfen wir allerdings 1qje wieder, da der Ermittlungsprozess nicht reproduziert werden konnte.
Sogleich versuchten wir eine Tertiärstruktur mit dem Programm Modeller zu berechnen, doch wollte uns dies nicht gelingen. Das Skript konnte nicht zum Laufen gebracht werden und auch professionelle Hilfe durch die Praktikumsbetreuer vermochte nicht bei der Lösung des Problems zu helfen. Selbst wenn es gelungen wäre, so ist das ganze Vorhaben für das Target T0257 auf Grund der vielen negativ-Ergebnisse sowieso als zweifelhaft einzustufen.
Um uns dennoch ein Bild von unserem Protein machen zu können, nutzten wir Stichproben der Strukturinformationen bereits vorhandener CASP6 pdb-Daten verschiedener Server, die auf der CASP6 Seite aufgeführt sind.
Dabei achteten wir auf Vergleichbarkeit der Daten, indem nur möglichst gleichlange Sequenzen betrachtet wurden.
| Ergebnisse | |
 |
 |
| 3D-JIGSAW TS1.pdb | ACE TS1.pdb |
 |
 |
| nFOLD TS1.pdb | ACE TS2.pdb |
TO276
Im Folgenden werden wir die Ergebnisse unserer Arbeit zum Target T0276 darlegen.
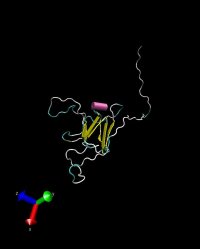
Zu dem von FUGUE gefundenen Protein existieren in der PDB 20 mögliche Tertiärstrukturen. Wir nutzten die ersten vier dieser Modelle für Tertiärstrukturvorhersagen von T0276. Diese vorhergesagten Strukturen finden sich im Folgenden:
Man erkennt Ähnlichkeiten und gemeinsame Motive. Die tatsächliche Tertiärstruktur von T0276 liegt wohl
innerhalb der Grenzen die durch die aufgeführten Strukturen gesetzt werden.
Wir validieren exemplarisch die erste Strukturvorhersage mittels RMS-Wert und des Energieniveaus des vorhergesagten Moleküls.
Die Distanz (RMS-Wert) zwischen homologer und vorhergesagter Struktur ist mit einem Wert kleiner 5 im Toleranzbereich.
Dies spricht dafür, dass die Vorhersage recht wahrscheinlich und ausreichend genau ist.
Im Energieschema ist gut zu erkennen, dass die Vorhersage energetisch stabil ist,
und dass ihre Energiekurve(grün) der Energiekurve des homologen Proteins(blau) ähnelt.
Das Webinterface FUGUE sagte folgende Tertärstruktur voraus:
Es ist gut zu erkennen, dass diese Struktur den oben Aufgeführten ähnelt.
Damit ist auch diese Vorhersage eine mögliche Lösung.
Zu dem von FUGUE gefundenen Protein existieren in der PDB 20 mögliche Tertiärstrukturen. Wir nutzten die ersten vier dieser Modelle für Tertiärstrukturvorhersagen von T0276. Diese vorhergesagten Strukturen finden sich im Folgenden:
| Illustration von T0276 | |
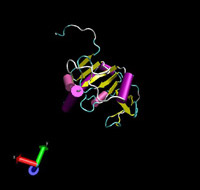 |
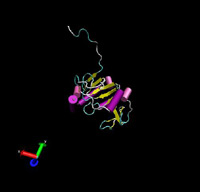 |
| t0276 vorhergesagt mit 1sou Modell 1 | t0276 vorhergesagt mit 1sou Modell 3 |
 |
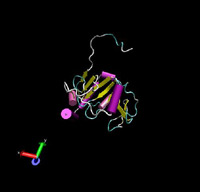 |
| t0276 vorhergesagt mit 1sou Modell 2 | t0276 vorhergesagt mit 1sou_4 |
Wir validieren exemplarisch die erste Strukturvorhersage mittels RMS-Wert und des Energieniveaus des vorhergesagten Moleküls.
| Validierung von T0276 | |
| RMS-Wert: 4.784 |
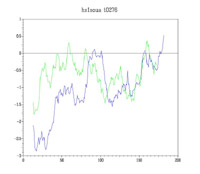 |
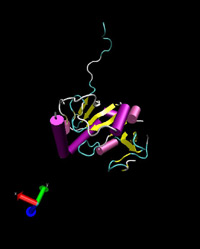
|

