


Next: Pumilio family (PUF)
Up: Bioinformatisches Praktikum
Previous: RNA Recognition Motif (RRM)
Subsections
Die SAM-Domäne findet sich in vielen funktionell unterschiedlichen Proteinen wie etwa Transkriptions- und Translationsregulatoren und Kinasen [3,9,6]. Dabei ist die Domäne selber auch an verschiedenen Funktionen beteiligt, meist an Protein-Protein Interaktionen. Aber auch posttranskriptionelle Regulation durch RNA-Bindung wurde entdeckt [8]. Die Sekundärstruktur der SAM-Domäne faltet sich in vier bis fünf 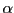 -Helices, welche ein globuläres Bündel mit hydrophobem Kern bilden.
-Helices, welche ein globuläres Bündel mit hydrophobem Kern bilden.
Dateien:
Table 8:
Übersicht der SAM-Domänenverteilung in den Taxa.
| Taxon |
#Gene: min/avg/max |
(annotiert/in superfam) |
| Archaea |
- |
(0/52) |
| Bacteria |
1/1.4/2 |
(5/668) |
| Eukaryota |
1/28.84/415 |
(188/193) |
| Fungi |
1/5.12/14 |
(78/78) |
| Metazoa |
15/72.73/415 |
(64/64) |
| Protostomia |
20/33.05/66 |
(22/22) |
| Deuterostomia |
44/102.59/415 |
(37/37) |
| Sarcopterygii |
44/91.79/189 |
(28/28) |
| Teleostei |
114/184.6/415 |
(5/5) |
|
Die SAM-Domäne ist fast ausschließlich in Eukaryoten vorhanden (siehe Tabelle 8). In diesen ist sie in den Untergruppen (bis auf die Alveolata) vollständig zu finden. Dies läßt ihren Ursprung in der Entstehung der Eukaryoten vermuten. Bei den fünf Funden in den Bakterien gibt es keinen experimentellen Beleg über ihre Funktionalität. Nach [7] könnte die große Sequenzdivergenz der SAM Domäne das Finden von Homologen durch Sequenzähnlichkeit in Bakterien verhindern.
Die Anzahl der Proteine mit SAM in den Organismen (Tabelle 8) im phylogenetischen Baum unterstützt die gängige Ansicht über die Zunahme der Genomgröße durch z.B. Genomduplikationen. Innerhalb der Deuterostomia (sogar der Mammalia) ist im Gegensatz zu dem restlichen Baum eine große Varianz der Genanzahl einzelner Organismen beobachtbar. Auch hier könnten die in 1.2 beschriebenen Probleme der Grund sein. Das Vorkommen von Proteinen mit mehr als einer SAM-Domäne (Tabelle 9) ist spezifisch für Metazoa.
Table 9:
Hier ist die durchschnittliche Anzahl an SAM-Domänen pro Protein für bestimmte Taxa gelistet. Der Durchschnitt pro Organismus berechnet sich durch die Anzahl seiner annotierten SAM-Domänen dividiert mit der Anzahl seiner annotierten SAM-enthaltenden Proteine.
| Taxon |
avg SAM pro Protein |
| Fungi |
1.00 |
| Metazoa |
1.21 |
| Protostomia |
1.27 |
| Deuterostomia |
1.17 |
|



Next: Pumilio family (PUF)
Up: Bioinformatisches Praktikum
Previous: RNA Recognition Motif (RRM)
root
2009-03-18