Herangehensweise und Ergebnisse fuer
"Protein fold Recognition" am Beispiel T0192:
- Da mittels PSI-Blast keine homologen Proteine gefunden wurden ( PSI-Blast ), haben wir den Weg ueber
Protein fold Recognition gewaehlt. Hierzu nutzten wir die Programme nnpredict
sowie 3d-pssm
E :beta strand element
H :helix element
C :no prediction
sequen:
MAKFVIRPATAADCSDILRLIKELAKYEYMEEQVILTEKDLLEDGFGEHPFYHCLVAEVPKEHWTPEGHSIVGFAMYYFTYDPWIGKLLYLEDFFVMSDYRGFGIGSEILKNLSQVAMRCRCSSMHFLVAEWNEPSINFYKRRGASDLSSEEGWRLFKIDKEYLLKMATEE
nnpred:
CCCEEECCCCCCCCHHHHHHHHHHHHHHHHHHHHHHHHHCCCHCCCCCCCCCECCCCCCCCCCCCCCCCCEECEEEEEECCCCCCCCHHEHHHHEECCCCCCCCCCHHHHHHHHHHHEEECCCCCCEEECCCCCCCCCCECCCCCCCCCCHHHHHHHHHHHHHHHHHHCCC
3dpssm:
CCCEEEEECCHHHHHHHHHHHHHHHHHCCCCCCCCCCHHHHHHHHCCCCHHHHHHHCCCCEEEEEEECCCEEEEEEEEECCCCCCCCEEEEEEEEECHHHHCCHHHHHHHHHHHHHHHHCCCCEEEEEECCCCHHHHHHHHHCCCEEEEEECCEEEEEECHHHHHHHHCCC
hompro:.
CCEEEEECCCCHHHHHHHHHHHHHHHHCCCCCHHHHHHHHHHHHCCCCCE.EEEEECCCCCCEECCCCCEEEEEEEEEECCCCCCCCEEHCCCCHHHHHHHHHHHHHHHHCCCCEEEEEECCCHHHHHHHHCCEEECCEEEEEEEECHHEEEEECCCCCCCCCCCCCCCCCCCC
- Das Programm 3d-dpssm suchte als aehnlichstes Protein bezueglich
der Sekundaerstruktur das Protein d1qsma mit einer e-value von 0.104 und
die Familie des Proteins ist alpha/beta.
- Durch ein multiples Alignment mittels FUGUE ( Target
T0192 gegen die Fold library ) bestaetigte sich die Aehnlichkeit
zwischen T0192 sowie d1qsma mit einem ZScore von 17,7 ( Ergebnis ) zudem erstellte FUGUE
die fuer den Modeller notwendige pir-Datei: T0192_d1qsma.pir
- Als naechtes suchten wir aus der PDB-Datenbank die PDB-Datei des
Proteins d1qsma herausgesucht und
erstellten die PDB-Datei unseren Targets: T0192.pdb
- Mittels VMD haben wir die PDB-Dateien grafisch dargestellt und
miteinander verglichen
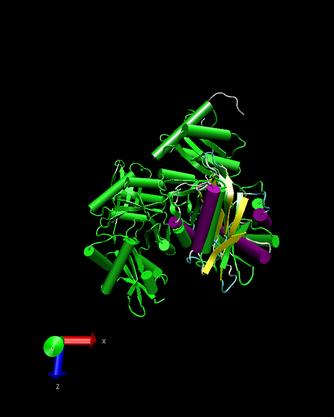
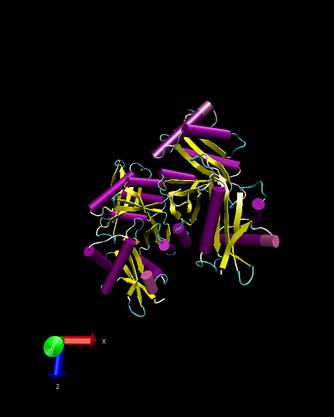
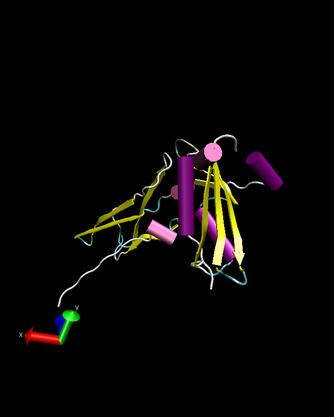
T0192
Ueberlagerung
d1qsma
- An dem Bild kann man erkennen, dass unser Target T0192 mit einer
Domain des Proteins d1qsma teilweise uebereinstimmt. Ein RMS-Wert von
8,619 verdeutlicht, dass es lediglich eine akzeptable Aehnlichkeit
beider Proteine gibt.
- Nun wurde die Aehnlichkeit nochmals mit dem Programm Prosa
untersucht ( prosa.ps ). Man
sieht, dass es eine zwar keine Ueberdeckung gibt, aber der Kurvenverlauf
eine gewisse Aehnlichkeit ( besonders im ersten Drittel ) aufweist. Die
Struktur ist leider nicht sehr glaubhaft, da sich die meisten Werte
unseres Targets oberhalb der Null-Linie bewegt.
Ergebnisse fuer T0146:
- Es wurden keine homologen Proteine gefunden: Blast-Ergebnis
- Sekundaerstrukturanalzse mittels nnpredict sowie 3-dpssm
sequence:
AFTPFPPRQPTASARLPLTLMTLDDWALATITGADSEKYMQGQVTADVSQMAEDQHLLAAHCDAKGKMWSNLRLFRDGDGFAWIERRSVREPQLTELKKYAVFSKVTIAPDDERVLLGVAGFQARAALANLFSELPSKEKQVVKEGATTLLWFEHPAERFLIVTDEATANMLTDKLRGEAELNNSQQWLALNIEAGFPVIDAANSGQFIPQATNLQALGGISFKKGCYTGQEMVARAKFRGANKRALWLLAGSASRLPEAGEDLELKMGENWRRTGTVLAAVKLEDGQVVVQVVMNNDMEPDSIFRVRDDANTLHIEPLPYSLEE
nnpredict:
CCCCCCCCCCCCCCCCHHEHHCHCHHHHHEECCCCCCHHCCCCCCCCHCHHHHHHHHHHHHHHCCCCCHHHHHECCCCCCCEEHHHCCCCCCCHHHHHHHHHCEECCCCCCCHHHEEEHHHHHHHHHHHHHHCCCCCCCHHHEHHCHHHHHHHHCCCCHHEEECCCHCHHHHHHCCCCCHCHHCCHHHHHHHHCCCCCEECCCCCCCCCCCCCCCHHCCCCECCCCCCCCHHHHHHHHHHCCCHHHHHHHHCCCCCCCCCCHHHHHHHCCCCCHCCHHHHHHHCCCCCEEEEEECCCCCCCCCCEEECCCCCCCCCCCCCCCCCC
3-dpssm:
CCCCCCHHHHHHHHHHCCEEEECCCCEEEEEECCHHHHHHHHCHHHCCCCCCCCCEEEEEEECCCCCEEEEEEEEECCCEEEEEECHHHHHHHHHHHHHHHCCCCCEEEECCCEEEEEEECCCHHHHHHHHCCCCCCCCEEECCCCEEEEECCCCCCCEEEEEECHHHHHHHHHHHHHCCCCCCHHHHHHHHHHHCCCCCCCCCCCCCCHHHHHHHHHCCCCCCCCCCCHHHHHHHHHHCCCCCEEEEEEEECCCCCCCCCCEEEECCCCCCCEEEEEEEEEECCCCCEEEEEEECCCCCCCCEEECCCCCCEEEEEECCCCCCC
hompro:
- Das Programm 3d-dpssm suchte als aehnlichstes Protein bezueglich
der Sekundaerstruktur das Protein 1ko3 mit
einer e-value von 0.0473
- Familie(a+b laut 3dpssm)
- mittels FUGUE erstellten wir die fuer den Modeller notwendige
pir-Datei: 1ko3-t0146.pir
- Als naechtes suchten wir aus der PDB-Datenbank die PDB-Datei des
Proteins 1ko3 herausgesucht und
erstellten die PDB-Datei unseren Targets: T0146.pdb
- Mittels VMD haben wir die PDB-Dateien grafisch dargestellt und
miteinander verglichen
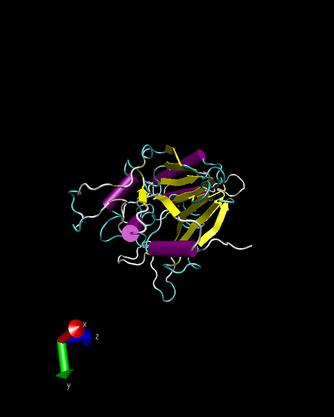
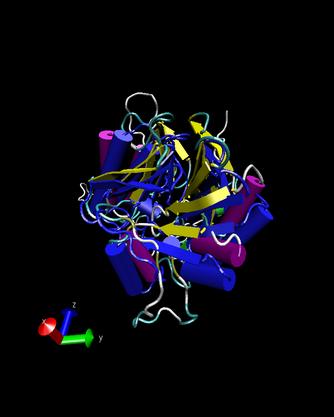
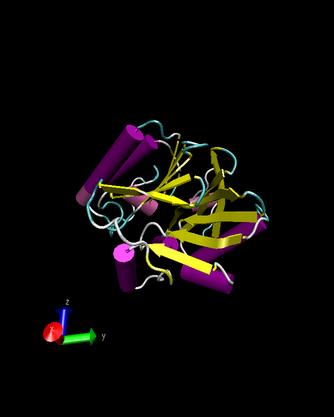
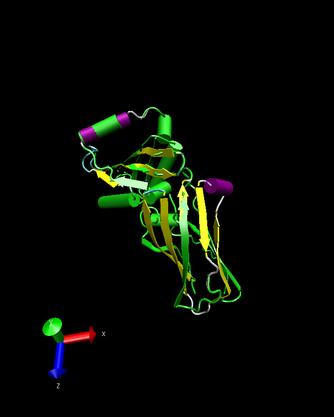
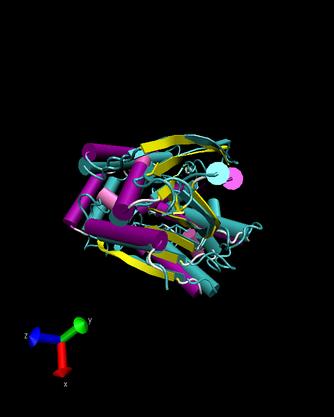
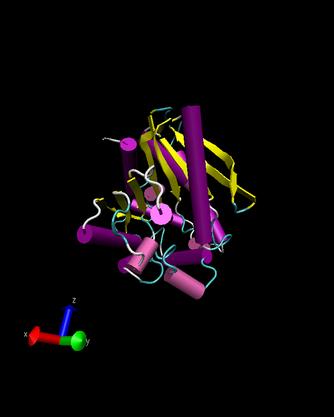
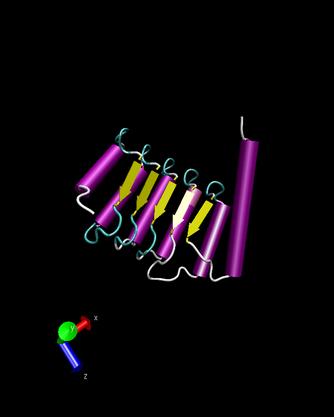
T0146
Ueberlappung(blau 1ko3)
1ko3
- Man kann erkennen, dass die Strukturen nicht sehr gut
uebereinstimmen, lediglich die alpha-helices sind aehnlich angeordnet.
Die Faltblaetter sind bei unserem Target ungeordnet und bei dem dem
Protein 1ko3 sind diese in zwei Reihen parallel zueinander angeordnet.
- Die Untersuchung der Aehnlichkeit mit dem Programm Prosa ( prosa.ps ) ergab, dass beide
Proteine unterschiedlich sind und dass es nur zu einem geringen Maß eine Aehnlichkeit besteht, dies bestaetigt auch
der RMS-Wert von 18,22.
Ergebnisse fuer T0157:
- Es wurden keine homologen Proteine gefunden
- Sekundaerstrukturanalyse mittels nnpredict sowie 3-dpssm (familie
a/b laut 3dpssm )
sequence:
MSGTLLAFDFGTKSIGVAVGQRITGTARPLPAIKAQDGTPDWNIIERLLKEWQPDEIIVGLPLNMDGTEQPLTARARKFANRIHGRFGVEVKLHDERLSTVEARSGLFEQGGYRALNKGKVDSASAVIILESYFEQGY
nnpredict:
CCCCEEEEHCCCCCEEEEECCEECCCCCCCCCCCCCCCCCCCCHHHHHHCCCCCCCEEECCCCCCCCCCCCHHHHHHHHHHHHCCCCCEEECCCCCCCCHHHCCCCCECHCCHHHHCCCCCCCCCEEEEEEHHCCCCC
3-dpssm:
CCEEEEEEECCCCEEEEEEECCCCCCCCCCEEEECCCHHHHHHHHHHHHHHCCCCEEEECCCCCCCCCCHHHHHHHHHHHHHHHHHCCCCEEEECCCCCHHHHHHHHHHCCCCCCCCCCCHHHHHHHHHHHHHHHCCC
- Das Programm 3d-spssm suchte als aehnlichstes Protein bezueglich
der Sekundaerstruktur das Protein d1hjra mit
einer e-value von 0.118
- Durch ein multiples Alignment mittels FUGUE ( Target T0157 gegen
die Fold library ) bestaetigte sich die Aehnlichkeit zwischen T0157
sowie d1hjra mit einem ZScore von 5 (Ergebnis). Dies
entspricht einer confidence von ueber 95%. Zudem erstellte FUGUE die
fuer den Modeller notwendige pir-Datei: T0157_d1hjra.pir
- Als naechtes suchten wir aus der PDB-Datenbank die PDB-Datei des
Proteins d1hjra herausgesucht und
erstellten die PDB-Datei unseren Targets: T0157.pdb
- Mittels VMD haben wir die PDB-Dateien grafisch dargestellt und
miteinander verglichen
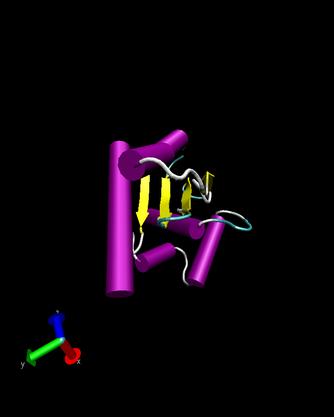

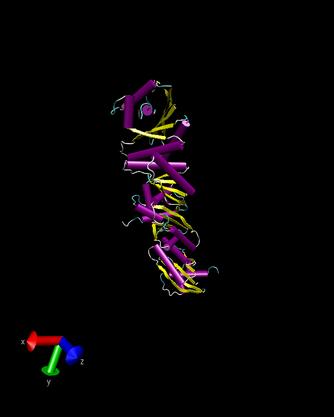
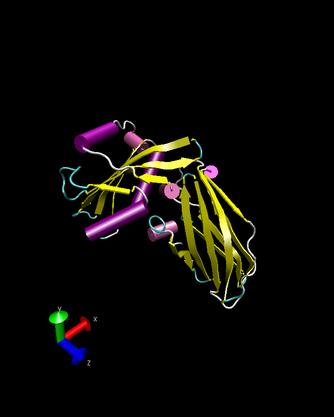
T0157
Ueberlagerung
d1hjra
- Man sieht, dass unser Target mit einer Domain des Proteins d1hjra
verglichen werden kann. Die Strukturen sind sich zwar aehnlich jedoch
gibts eine große Winkelverschiebung,
wordurch der schlechte RMS-Wert von 16,98 zu erklaeren ist.
- Auch die Untersuchung mit dem Programm
Prosa ( prosa.ps ) ergab, dass
es eine Uebereinstimmung der Struktur lediglich in einzelnen Bereichen
zu finden ist, vor allem in der ersten Haelfte.
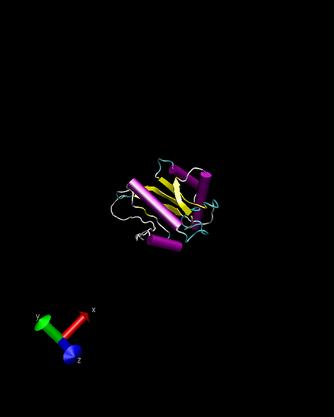
Ergebnisse fuer T0134:
 Franziska Heinze
Franziska Heinze Franziska Heinze
Franziska Heinze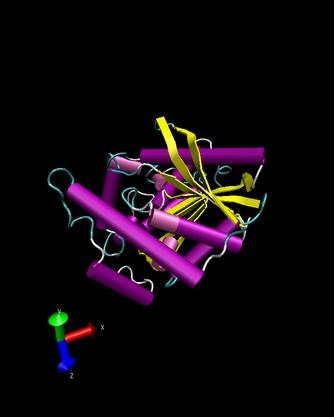





 T0146
Ueberlappung(blau 1ko3)
1ko3
T0146
Ueberlappung(blau 1ko3)
1ko3