|
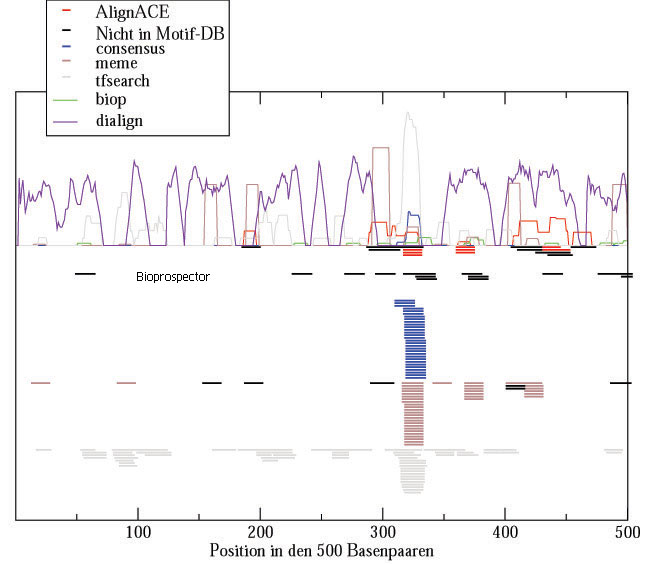
In Abbildung 8 werden Motive wiedergegeben, die durch verschiedenene Programme gefunden wurden. Die Regulatorische Region des Hoxgens Aa11 vom Zebrafisch wurde untersucht. Jedes Programm besitzt eine Farbe und sind vertikal in Banden angeordnet. Die schwarzen Striche sind Motive, die durch die Programme gefunden wurden aber nicht in der TRANSFAC Datenbank in MatComapre. Farblich hervorgehoben sind Motive, die auch in der Datenbank erkannt wurden.
Auffallend ist, dass Meme, Consensus und Tfsearch die gleiche Tendenz bei der Anzahl der gefundenen Motive im Bereich der Basenpaare 325 zeigen. Allerdings hat Consensus nur dort Ergebnisse und diese beschränken sich auch nur auf den Zebrafisch (siehe Abbildung 6). AlignAce hat ebenfalls eine geringe Ansammlung von gefundenen Motiven und Bioprospector hat in diesem, sowie in weiteren Plots keine, bzw. kaum TRANSFAC Datenbank hits. Alle Motive sind deswegen schwarz gekennzeichnet. Meme und AlignAce haben ausserhalb der starken Motivansammlung auch Treffer, die in der Datenbank Analoge zu haben scheinen. Wir fanden die Ergebnisse von Meme und AlignAce von der Trefferverteilung und der Anzahl der in der Datenbank gefundenen Motive am aufschlussreichsten.
Die Programme rVista und YMF wurden nicht weiter benutzt. RVista nimmt zur Motiverkennung lediglich 2 .fasta Dateien entgegen.