



Nächste Seite: Literatur
Aufwärts: Bioinformatik Modul Transkriptionsregulation
Vorherige Seite: Programmvergleich
Inhalt
Bei der Auswertung der Ergebnisse fiel auf, dass die Art und Weise der Durchführung nicht immer optimal war. Ein paar Faktoren könnten starken Einfluss auf die Endresultate genommen haben. Hier eine Aufzählung.
- Die Motivsuche wurde lediglich über die vereinzelten Cluster durchgeführt. Das hat sich direkt auf
die Motivverteilung in den Organismen ausgewirkt. Es haben sich wiederum die gleichen Cluster gebildet.
Eine Motiverkennung über alle Cluster hätte die Menge der möglichen Motive stark beeinflusst. So versucht z.B. AlignAce ein Motiv über alle Sequenzen zu finden, also einen Konsens. Die Ergebnismenge wäre geringer aber dafür die Treffer umso deutlicher. Andere Methoden hätten sicherlich stark abweichende Resultate erbracht.
- Bei den Programmen wurden immer die ersten 10 Motive genommen. Daraus kann sich ein Verlust oder Redundanz von Motiven ergeben. Wir sollten für jede Score abwegen, wie gross
der cut-off ausfallen soll. Zwar wird durch die spätere Suche in den Bindungsmotivdatenbanken der Score bereinigt (
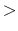 0.9) aber es ist nicht auszuschliessen, dass durch oberflächliche Auswahl der Ausgangmotive, der Datenbestand nachhaltig von einem Optimum abweicht.
0.9) aber es ist nicht auszuschliessen, dass durch oberflächliche Auswahl der Ausgangmotive, der Datenbestand nachhaltig von einem Optimum abweicht.
- Es wurden lediglich 500 Basen upstream untersucht. Es wurde zum Ende festgestellt, dass der Regulationsbereich darüber hinausgeht und 2500 Basen angemessen sind. Wir haben einen Motivplot (Abbildung ...) mit Meme, Ace und TfSearch mit 2500 Basen durchgeführt aber sind wegen Zeitmangels nicht weiter darauf eingegangen.




Nächste Seite: Literatur
Aufwärts: Bioinformatik Modul Transkriptionsregulation
Vorherige Seite: Programmvergleich
Inhalt
Praktikum
2005-11-17