Methoden und Ergebnisse
Konservierung von TNFalpha?
TNFa Protein gegen verschiedene Spezies geblastet (Ensembl)
UCSC
Erkenntnis:
TNFa-aehnliche Proteine sind in Eutheria vorhanden.
In Pro- und Metatheria ist die Uebereinstimmung geringer.
Sammlung von Protein- und mRNA- Sequenzen (inklusive 3' UTR) von TNFa aus unterschiedlichen Spezies
tBlastn: Blasten der Proteinsequenzen gegen bekannte Genome der Metazoen
Selektion der Hits anhand der E-Werte (1,0 * 10-50 (tBlastn_pipe)
Ausschneiden der Bereiche mittels fastacmd (Festlegen der Bereiche anhand der Ergebnisse von Osman et al. 1999)
Laenge: 500 nt (50 nt im Exon 4; 450 nt in 3' UTR)
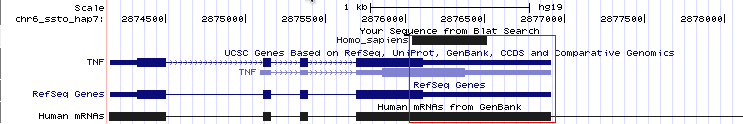
 Qualitaetskontrolle (Auswahl):Ausgeschnittener Sequenzbereich des Menschen gegen humanes TNF
Qualitaetskontrolle (Auswahl):Ausgeschnittener Sequenzbereich des Menschen gegen humanes TNF
 Multiples Sequenzalignment der ausgeschnittenen Sequenzen mittels ClustalW
Multiples Sequenzalignment der ausgeschnittenen Sequenzen mittels ClustalW
In der Abbildung ist der stemloop markiert, der auch in der Arbeit von Osman et al. zur Aktivierung von PKR gefuehrt hat.
Abgegrenzt sind Alignments, die weniger gut zu den anderen Säugetieren passen. Zu diesen zählen Beuteltiere, die Ratte und die Maus.
RNA-Origami
Problem: gefuehlt n verschiedene Faltungsprogramme ergeben n2 verschiedene Sekundaerstrukturen - zum Beispiel an der menschlichen RNA
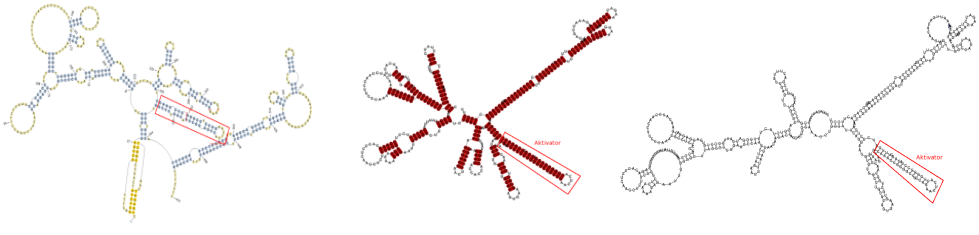 pKnots locaRNA RNAfold
pKnots locaRNA RNAfold
locaRNA zeigt Konservierung der Struktur und des Alignments an. Daher wurde hauptsaechlich mit diesem Programm weiter gearbeitet.
Auch zeigten andere Programme aehnliche Ergebnisse (z.B. rCoffee) oder konnten nur mit kuerzeren Sequenzen arbeiten.
Strukturalignments
Zunaechst wurde ein Alignment verschiedener Mammalia angefertigt, um einen Ueberblick ueber konservierte Strukturen zu erhalten.
Da durch die erhaltene Struktur ersichtlich wird, dass die einzelnen Regionen innerhalb der Bandbreite der Mammalia nicht sonderlich stark konserviert sind,
wurden einzelne (Super-)Ordnungen alignt.
In den angefuegten Links sind die Ergebnisse der Strukturalignments der Pro- und Metatheria, Afrotheria, Insectivora,
Chiroptera, Cetartiodactyla, Carnivora, Rodentia und der Primaten aufgeführt.
Grundsaetzlich sind in allen untersuchten Strukturen entsprechende Stemloops zu finden, die potentielle Aktivatoren für PKR darstellen koennten.
In den Primaten ist die bereits bekannte Aktivatorsequenz (Osman et al. 1999) am staerksten konserviert.
Bei den Chiroptheren gibt es hingegen mehrere moegliche Aktivatoren. Es lassen sich in allen Gruppen stemloops nachweisen. Deren Sequenzen weichen jedoch zum Teil stark von der publizierten Aktivatorsequenz ab. Es wird angenommen, dass die Akvierung von PKR
in erster Linie von einer Stemloop-Struktur abhaengt. Die unterschiedlichen Sequenzen in den Stems sprechen daher nicht gegen eine Aktivierung von PKR.
Die Alignierung mittels ClustalW fuehrte zum Nachweis der Aktivatorsequenz.
Beuteltiere und auch Nagetiere stellten diesbezueglich Ausnahmen dar.
 Mittels foldalign konnte die auch in Oryctolagus nachgeweisen werden. Hier weicht die Struktur des stems jedoch leicht von der postulierten
Form ab.
Strukturmotive
Durch den Einsatz von rmdetect wurden Strukturmotive in den Sequenzen gesucht.
Mittels foldalign konnte die auch in Oryctolagus nachgeweisen werden. Hier weicht die Struktur des stems jedoch leicht von der postulierten
Form ab.
Strukturmotive
Durch den Einsatz von rmdetect wurden Strukturmotive in den Sequenzen gesucht.
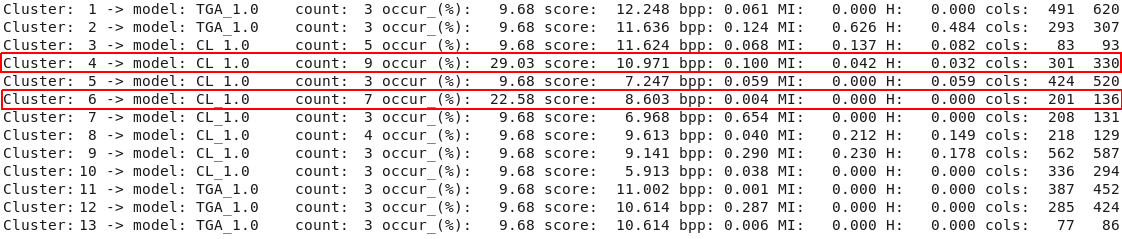 Mittels rmdetect wurden zwei haeufiger auftretende Strukturmotive gefunden. Bei diesen handelte es sich in beiden Faellen um einen C-Loop. Der haeufiger vorkommende
C-Loop befindet sich in der Naehe der postulierten Aktivatorsequenz. Der zweite C-Loop ist ca. 100 nt vor dem Ersten zu finden.
Weitere Strukturmotive
Andere Sekundaerstrukturen wie Kink-Turns oder Pseudoknoten koennten ebenfalls vorkommen. Solche Strukturen wurden jedoch fuer TNFalpha nicht erwaehnt (Osman et al.).
Da Pseudoknoten jedoch beispielsweise bei IFNgamma eine Rolle bei der PKR-Aktivierung spielen, wurde auch bei TNF danach gesucht. Dies wurde vor allem mittels pKnots
realisiert, welches auch laengere Sequenzen bearbeiten kann. Dabei faltete pKnots jedoch nur am Anfang oder Ende der Sequenz Pseudoknoten.
Andere Programme zur Vorhersage von Sekundaerstrukturen konnten nur kuerzere Sequenzen bearbeiten und waren daher fuer die hier verwendeten Fragmente unpassend.
Startseite
Bekanntes und Zielstellung
Zusammenfassung
Mittels rmdetect wurden zwei haeufiger auftretende Strukturmotive gefunden. Bei diesen handelte es sich in beiden Faellen um einen C-Loop. Der haeufiger vorkommende
C-Loop befindet sich in der Naehe der postulierten Aktivatorsequenz. Der zweite C-Loop ist ca. 100 nt vor dem Ersten zu finden.
Weitere Strukturmotive
Andere Sekundaerstrukturen wie Kink-Turns oder Pseudoknoten koennten ebenfalls vorkommen. Solche Strukturen wurden jedoch fuer TNFalpha nicht erwaehnt (Osman et al.).
Da Pseudoknoten jedoch beispielsweise bei IFNgamma eine Rolle bei der PKR-Aktivierung spielen, wurde auch bei TNF danach gesucht. Dies wurde vor allem mittels pKnots
realisiert, welches auch laengere Sequenzen bearbeiten kann. Dabei faltete pKnots jedoch nur am Anfang oder Ende der Sequenz Pseudoknoten.
Andere Programme zur Vorhersage von Sekundaerstrukturen konnten nur kuerzere Sequenzen bearbeiten und waren daher fuer die hier verwendeten Fragmente unpassend.
Startseite
Bekanntes und Zielstellung
Zusammenfassung



 Mittels foldalign konnte die auch in Oryctolagus nachgeweisen werden. Hier weicht die Struktur des stems jedoch leicht von der postulierten
Form ab.
Strukturmotive
Durch den Einsatz von rmdetect wurden Strukturmotive in den Sequenzen gesucht.
Mittels foldalign konnte die auch in Oryctolagus nachgeweisen werden. Hier weicht die Struktur des stems jedoch leicht von der postulierten
Form ab.
Strukturmotive
Durch den Einsatz von rmdetect wurden Strukturmotive in den Sequenzen gesucht.
 Mittels rmdetect wurden zwei haeufiger auftretende Strukturmotive gefunden. Bei diesen handelte es sich in beiden Faellen um einen C-Loop. Der haeufiger vorkommende
C-Loop befindet sich in der Naehe der postulierten Aktivatorsequenz. Der zweite C-Loop ist ca. 100 nt vor dem Ersten zu finden.
Weitere Strukturmotive
Andere Sekundaerstrukturen wie Kink-Turns oder Pseudoknoten koennten ebenfalls vorkommen. Solche Strukturen wurden jedoch fuer TNFalpha nicht erwaehnt (Osman et al.).
Da Pseudoknoten jedoch beispielsweise bei IFNgamma eine Rolle bei der PKR-Aktivierung spielen, wurde auch bei TNF danach gesucht. Dies wurde vor allem mittels pKnots
realisiert, welches auch laengere Sequenzen bearbeiten kann. Dabei faltete pKnots jedoch nur am Anfang oder Ende der Sequenz Pseudoknoten.
Andere Programme zur Vorhersage von Sekundaerstrukturen konnten nur kuerzere Sequenzen bearbeiten und waren daher fuer die hier verwendeten Fragmente unpassend.
Mittels rmdetect wurden zwei haeufiger auftretende Strukturmotive gefunden. Bei diesen handelte es sich in beiden Faellen um einen C-Loop. Der haeufiger vorkommende
C-Loop befindet sich in der Naehe der postulierten Aktivatorsequenz. Der zweite C-Loop ist ca. 100 nt vor dem Ersten zu finden.
Weitere Strukturmotive
Andere Sekundaerstrukturen wie Kink-Turns oder Pseudoknoten koennten ebenfalls vorkommen. Solche Strukturen wurden jedoch fuer TNFalpha nicht erwaehnt (Osman et al.).
Da Pseudoknoten jedoch beispielsweise bei IFNgamma eine Rolle bei der PKR-Aktivierung spielen, wurde auch bei TNF danach gesucht. Dies wurde vor allem mittels pKnots
realisiert, welches auch laengere Sequenzen bearbeiten kann. Dabei faltete pKnots jedoch nur am Anfang oder Ende der Sequenz Pseudoknoten.
Andere Programme zur Vorhersage von Sekundaerstrukturen konnten nur kuerzere Sequenzen bearbeiten und waren daher fuer die hier verwendeten Fragmente unpassend.