Einführung
Einführung
Aufgabenstellung
Aufgabe des Praktikums war es RNA-Sequenzen einer bestimmten Subfamilie der Retroviren zu analysieren und daraus Schlüsse über die Verwandtschaft unter den einzelnen Vertretern zu ziehen und möglicherweise vorhandene konservierte Sekundärstrukturen in diesen aufzuspüren und zu deuten.
Unsere Aufgabe im speziellen bestand in der Analyse von RNA-Sequenzen der Spumaviren.
Wissenswertes
Familie: Retrovidae
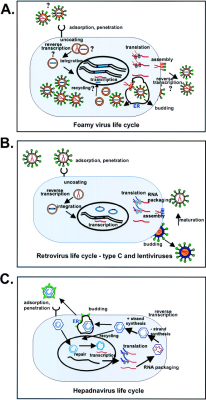
Retroviren sind RNA-Viren, besitzen also nur RNA als genetisches Material. Von allen RNA-Viren besitzen sie die kompliziertesten Reproduktionszyklen. Dabei bezieht sich die Bezeichnung Retro (lat. zurück) auf die umgekehrte Richtung des genetischen Informationsflusses, denn diese Viren können mit Hilfe eines Enzyms, der reversen Transkriptase, RNA in DNA umschreiben. Die dabei gebildete DNA wird dann als Provirus in ein Chromosom einer Wirstszelle eingebaut. Die Wirtszelle transkribiert nun auch immer die virale DNA, entweder zur Synthese von viralen Proteinen oder zur Weitervererbung an die Nachkommen.
Genom:
Die Spumaviren gehören zu dieser Gruppe der Retroviren, sie besitzen wie alle
Viren dieser Familie eine einzelsträngige RNA. Das Genom eines Spumavirus
besteht aus ca. 11 bis 13 tausend Basen und ist als Dimer aufgebaut, d.h. die
Monomere sind über Wasserstoffbrücken verbunden. Wie bei den Retroviren üblich,
besitzen sie 4 Gene, welche für virale Protein kodieren deren prototypische Anordnung
folgendermassen lautet: 5'-gag-pro-pol-env-3'
Die Besonderheit der Spumaretroviren besteht in zusätzlichen Genen, die mit "bel"
und einer Nummer bezeichnet werden.
Morphologie:
Spumaviren sind wie alle Retroviren behüllt und besitzen zusätzlich noch prominente Spikes, die so genannten Envelope-Gene (kurz: env) codieren diese Spikes bzw. Glycoproteine welche in der Virushülle integriert sind. Das Kapsid eines Spumavirus wird vor der Ausschleusung an der Zellmembran zusammengesetzt.
Replikationszyklus
Taxonomie:
Die Spumaviren werden noch weiter in folgende Gruppen unterteilt:
- Bovine foamy virus
- Equine foamy virus
- Feline foamy virus
- Feline syncytial virus
- Foamy retrovirus
- Human foamy virus
- Human spumaretrovirus
- Simian foamy virus
- Simian foamy virus type 1
- Simian foamy virus type 3
- Simian foamy virus type 6
Forschung:
Es gibt sowohl endogene als auch exogene Viren in diesem Genus, aber keines ist mit einem Krankheitsbild verknüpft, was sie sehr interessant für den Einsatz in der Gentherapie macht. Am auffälligsten ist jedoch die Eigenschaft der Spumaviren, dass infizierte Zellen sehr viele Vakuolen bilden, bevor der Zelltod eintritt, weshalb der Spumaretrovirus auch unter der Bezeichnung "Foamy Retro Virus" bekannt ist.