Retrovirusklassen
Alpharetrovirus
Betaretrovirus
Gammaretrovirus
Deltaretrovirus
Epsilonretrovirus
Lentivirus
Spumavirus
Retrovirus = Reverse Transkriptase Onkovirus
Allgemeines
Bei Retroviren liegt das Genom als RNA vor.
Wenn das Virus diese RNA in die zu befallende Zelle eingebracht hat, so muss sie erst in das "Format" der Erbinformation der Wirtszelle (=DNA) umgewandelt werden.
Dies geschieht mit Hilfe des Enzyms Reverse Transkriptase. (hierzu ein kleines Video )
Mutationen die bei diesem Prozess häufig erzeugt werden, ermöglichen eine schnelle Anpassung des Virus an antivirale Medikamente und damit eine Ausbildung von Resistenzen.
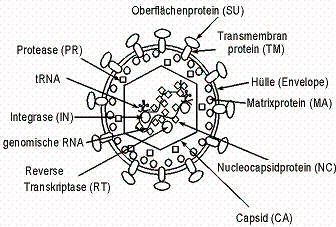
Morphologie
Retroviren sind behüllte Partikel mit ca. 100 nm Durchmesser.
Die Hülle trägt vom Virus codierte "spikes":
- das äußere Hüllen-Glycoprotein (SU), welches das Hauptantigen des Virus darstellt, die Bindung zur Zelle herstellt und über Disulfidbrücken mit
- dem transmembranen Glycoprotein (TM) verbunden ist.
Dieses hält SU in der Hülle und initiiert die Membranfusion.
 Aufbau des Retrovirusgenoms
Aufbau des Retrovirusgenoms
Das Retrovirusgenom besteht aus 2 RNA-Molekülen, einzelsträngig und positivstrangorientiert, die am 5' Ende eine Cap haben und am 3' Ende polyadenyliert sind.
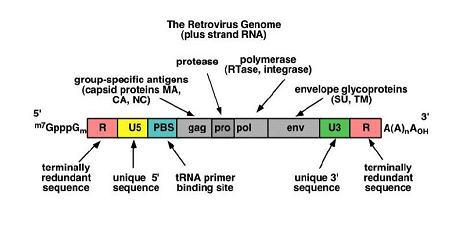
Die Hülle des Genoms entsteht aus der Zellmembran der virusproduzierenden Zelle und enthält zusätzlich das virale Hüllprotein (Env, envelope)
Virusproteine
1. Gruppenspezifische Antigene (Gag-Proteine)
Gag-Vorläuferproteine werden durch virale Proteasen in verschiedenen Proteine gespalten,
die man in freigesetzten infektiösen Viruspartikeln findet.
Die sequenzielle Anordnung der Gag-Proteine im Vorläuferprotein stimmt bei allen Retroviren überein.
Die Gag-Proteine haben mehrere Bedeutungen für das Virus:
- Bildung von Viruspartikel strukturierenden Proteinen.
- Nucleocapsidproteine binden an die virale RNA, kondensieren sie und bereiten sie so auf das packing vor.
- Die Matrixproteine sind auf der Innenseite der Virusmembranhülle lokalisiert.
Sie unterstützen das Einschleusen der doppelsträngigen Virus-DNA in den Zellkern.
Gag-Proteine induzieren die spezifischen Immunantworten auf humoraler wie auch zellulärer Basis, was auf den hohen Gehalt konservierter Aminosäuren zurückzuführen ist.
2. Enzyme
Die Protease, Reverse Transkriptase und die Integrase der Retroviren sind ebenfalls im Gag/Pol-Bereich der Pro-Virus-DNA codiert.
Die Protease spaltet in den ausgeschleusten Viren die Gag- und Gag/Pol-Vorläuferproteine in die Komponenten, die man in infektiösen Viruspartikeln vorfindet.
Die Reverse Transkriptase hat RNA- wie auch DNA-abhängige DNA-Polymerase-Aktivität.
Zusätzlich funktioniert sie noch als Rnase-H, die die RNA von DNA/RNA-Hybriddoppelsträngen abbaut.
Dieses Enzym hat keine Mechanismen zur Kontrolle der Lesegenauigkeit,
was zu einer großen Anzahl falsch eingebauter Basen bei der Neusynthese führt
3. Membranproteine (env)
Die Envelope-Gene codieren Glycoproteine, die in der Cytoplasmamembran infizierter Zellen zu finden sind.
Aus einem Vorläuferprodukt gehen das externe (EP) und das transmembrane (TP) Protein durch Spaltung im ER (und Golgi) hervor.
Der Lebenszyklus von Retroviren
Die An- bzw. Abwesenheit eines geeigneten Rezeptors auf der Zielzelle legt fest, ob die Zelle von einem bestimmten Virus befallen werden kann oder nicht. Die Rezeptoren, die von Retroviren für die Aufnahme in die Zelle verwendet werden, sind normale zelluläre Proteine, die für den normalen Haushalt der Zelle nötig sind. Bisher sind nur wenige dieser Proteine, die eine Bedeutung als virale Rezeptoren haben, identifiziert worden. Das am häufigsten für die Konstruktion retroviraler Vektoren verwendete Retrovirus ist das murine Leukämievirus (MLV).
Einteilung der Viren:
- Ecotrope Viren: Können nur die Spezies infizieren, aus der sie produziert worden sind.
- Amphotrope Viren: Können auch andere Spezies infizieren
- Xenotrope Viren: Können alle Spezies infizieren außer der, aus der sie produziert worden sind.
Zwei verschiedene Aufnahmemechanismen werden vermutet
- entweder eine zelluläre Aufnahme durch Mikropinocytose im Vesikel, gefolgt von einer Fusion
- oder durch direkte Fusion der Virushülle mit der Zellmembran
Die Typisierung in endo-/exogen bezieht sich auf die Tatsache,
dass die mit exogen bezeichneten Retroviren wie andere Viren durch Infektion weitergegeben werden.
Endogene Retroviren sind in der Wirtskeimbahn schon integriert, sind oft inaktiv, können aber bei Bedarf wieder aktiviert werden.
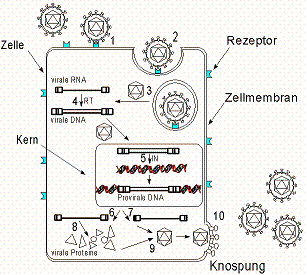 Replikation des Genoms
Replikation des Genoms
- Transkription
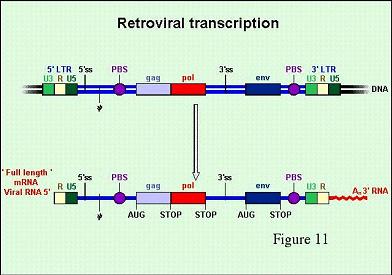
Die doppelsträngige DNA-Kopie, flankiert von den LTRs, wird in den Zellkern eingeschleust.
Der Eintritt in den Zellkern erfordert die Abwesenheit der Kernmembran und ist daher auf aktiv sich teilende Zellen eingeschränkt.
- Integration
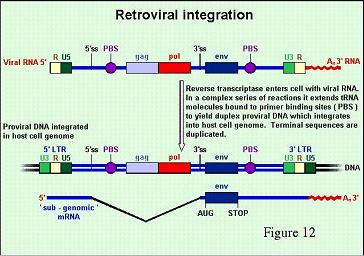
Im Zellkern angelangt, liegt die virale DNA als lineares Molekül vor.
Der Prozeß der Integration beginnt mit der Erkennung der endständigen Nucleotide der LTRs durch IN.
IN ist ein virales Enzym, das vom pol-Bereich codiert wird und als Protein im Viruskern vorhanden ist.
Nach dieser Erkennung werden die beiden endständigen Nucleotide der LTRs auf jeweils einem Strang abgebaut.
Dies führt zu einem einzelsträngigen Überhang von zwei Nucleotiden an beiden Enden des linearen viralen DNA-Moleküls.
Gleichzeitig schneidet IN die zelluläre DNA.
Die überhängenden Enden der viralen und zellulären DNA werden durch Ligation miteinander verbunden,
auch wenn die jeweiligen Basen der viralen und der zellulären DNA nicht komplementär sind.
Nach der Ligation werden solche nicht-komplementären Bereiche als noch bestehende Einzelstrang-Lücken durch zelleigene Reparatursysteme instandgesetzt.
Das Ergebnis ist eine integrierte virale DNA (Provirus),
die um insgesamt vier Basen - zwei von jedem Ende - kürzer ist als die DNA vor der Integration,
flankiert auf beiden Seiten durch identische genomische Sequenzwiederholungen.
- Translation
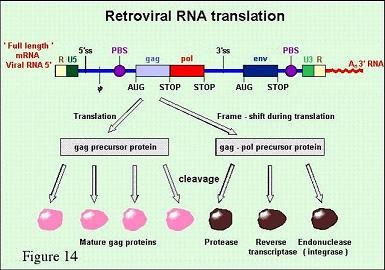
Die Gag- und Pol-Proteine werden in Form eines Polyproteinvorläufers von Transkripten genomischer Länge translatiert.
Diese Vorläuferproteine werden Stück für Stück proteolytisch in die reifen Gag-Proteine gespalten.
Dies ist ein autokatalytischer Prozeß und führt zum Viruszusammenbau und weiter zur Knospung aus der Wirtszelle,
wo die letzten Reifungsschritte ablaufen.
Knospung und Reifung
Die letzten Schritte des Zusammenbaus und der Freisetzung der neugebildeten Virionen erfolgen an der Zellmembran.
Durch Knospung werden die neuen Virionen freigesetzt.
Diese Viruspartikel durchlaufen noch weitere Reifungsstadien,
in denen Proteinspaltung, Konformationsänderungen und eine Reorganisation der viralen Proteine im Viruskern erfolgen,
ehe sie als völlig ausgereifte infektiöse Viren vorliegen.
Während der gesamten Prozesse der retroviralen Infektion und Virusproduktion wird die virusproduzierende Zelle in keiner Weise geschädigt.
Sekundärstruktur
Normalerweise sind RNA-Moleküle einzelsträngig.
Sie sind jedoch in der Lage sich zu falten und so eine Doppel-Helixstruktur ausbilden.
Diese besteht größtenteils aus den bekannten "Watson-Crick-Basenpaaren" (GC und AU), die zur Stabilität der Struktur beitragen,
sowie aus den weniger stabilen GU-Paaren und wird als Sekundärstruktur bezeichnet.
RNA Sekundärstrukturen können zur Interpretation von RNA-Funktionen und Reaktionsfähigkeit benutzt werden.
Sekundärstrukturen mit wichtigen Funktionen werden bei verwandten Arten im Laufe der Evolution konserviert.
Schon kleine Punktmutationen können große Veränderungen in der Sekundärstruktur hervorrufen.
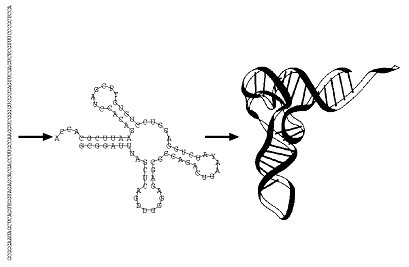 Repräsentation von Sekundärstrukturen
Repräsentation von Sekundärstrukturen
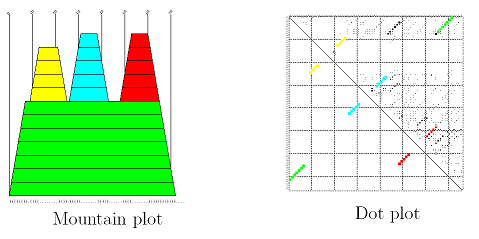
MountainPlot
- Repräsentation als "Berg"
- Die drei Symbole der String-Repräsentation `.´ `(´ und `)´
werden im Plot den 3 Richtungen `horizontal´ `hoch´ und `runter´ zugeteilt
- Die Strukturelemente entsprechen bestimmten Sekundärstrukturmerkmalen:
- Peaks: entsprechen den Hairpins (Haarnadeln).
Die symetrischen Hänge repräsentieren die Stämme, die die unpaarigen Basen in der Haarnadelschleife (Hairpin-Loop)
schliessen, welche als Plateau erscheinen.
- Plateaus: Repräsentieren unpaarige Basen.
Unterbrochene (unterbrechende) Stammregionen zeigen sie an Ausbuchtungen oder inneren Schlingen (Loops halt),
abhängig davon, ob sie allein oder gepaart mit einem anderen Plateu auf der anderen Seite des Berges auf selber Höhe auftreten
- Hügel: Entsprechen den ungepaarten Regionen zwischen Zweigen einer Mehrfachschleife.
DotPlot
- Jedes Quadtrat steht für ein Basenpaar, wobei der Bereich proportional zu der vorhergesagten Basenpaarungswahrscheinlichkeit ist.
- Die Bedeutung der Farben entspricht der Bedeutung der Farben im MountainPlot.









