
Next: Bibliography
Up: Praktikumsbericht RNA
Previous: Analyse mit Nussinov
Wie schon gezeigt, werden fuer lange RNA-Sequenzen (>200nt) lange Basenpaare mit zu grosser Wahrscheinlichkeit vorrausgesagt. Deswegen sollen sogenannte MEA-Strukuturen vorhergesagt werden und lange Basenpaare staerker bestraft werden. MEA (maximum expected accuracy) ist eine Bewertungsfunktion for Sekundaerstrukturen, die sowohl die Wahrscheinlichkeit fuer ein Basenpaar sowie auch die Wahrscheinlichkeit dafuer, dass eine bestimmte Base nicht bindet, in die Bewertung einbezieht. Die Wahrscheinlichkeiten dafuer, dass ein bestimmtes Basenpaar (i,j) eine Sekundaerstruktur formt, werden aus den von RNAfold berechneten Dotplots ausgelesen und in eine Matrix eingetragen. Gleichzeitig werden auch die Wahrscheinlichkeiten, dass eine bestimmte Base nicht bindet, berechnet. Durch dynamische Programmieren wird die Struktur gefunden, die den hoechsten moeglichen Score hat, gegeben die Bewertungsfunktion, die sich mit Hilfe der Betrafungsfunktion und den gegeben Wahrscheinlichkeiten berechnen laesst.
Die Bestrafungsfunktion fuer lange Basenpaare sieht wie folgt aus.
Base pair penalty:
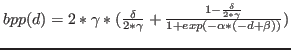
Der Eingabewert fuer die Bestrafungsfunktion ist der Abstand zwischen
den beiden Basen des betrachteten Basenpaares 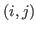 , also,
, also,
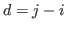 . Durch die anderen Parameter kann die Bestrafung fuer lange
Basenpaare variiert werden. Hierbei gibt
. Durch die anderen Parameter kann die Bestrafung fuer lange
Basenpaare variiert werden. Hierbei gibt 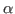 die Steigung der
Funktion an,
die Steigung der
Funktion an, 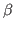 ist die Distanz zwischen den beiden Basen die den
Wendepunkt in der Bestrafungsfunktion angibt,
ist die Distanz zwischen den beiden Basen die den
Wendepunkt in der Bestrafungsfunktion angibt, 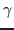 ist der
Gewichtungsfaktor und
ist der
Gewichtungsfaktor und 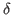 gibt den minimalen Bestrafungsfaktor
fuer Basenpaare an.
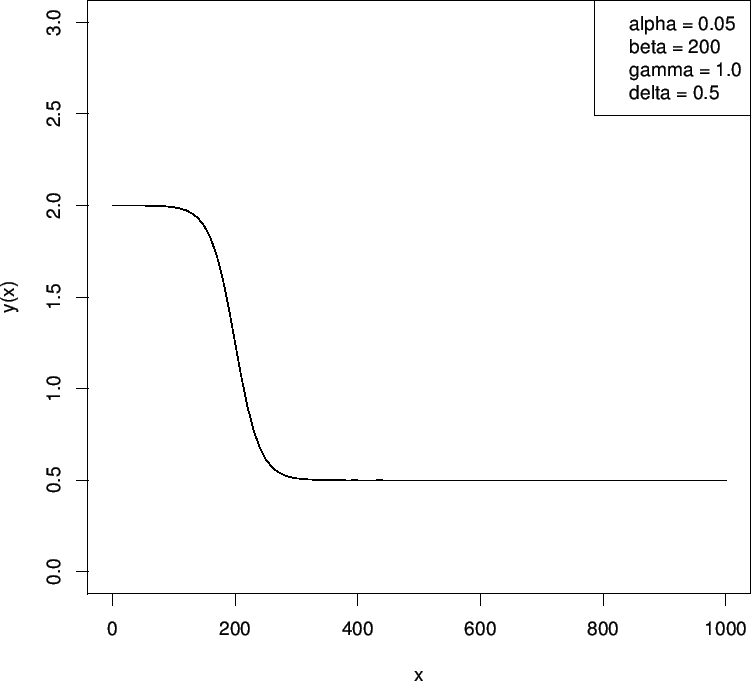
Die Bestrafungsfunktion mit den Standardwerten
gibt den minimalen Bestrafungsfaktor
fuer Basenpaare an.
Die Bestrafungsfunktion mit den Standardwerten
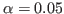 ,
, 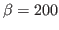 ,
,
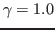 und
und
 sieht dann wie folgt aus.
sieht dann wie folgt aus.
Figure 11:
Basenpaarbestrafungsfunktion
 |
Die Werte fuer die Parameter koennen weiter optimiert werden, indem immer nur einer der Parameter in einem bestimmten Intervall veraendert wird und der F-Score fuer die damit vorhergesagten Sequenzen und Referenzsequenzen ueber die verschiedenen Werte der Parameter maximiert wird. Die Parameteroptimierung geschieht in mehreren Interationen, fuer die die Intervalle der Parameter immer wieder angepasst werden. Gleichzeitig werden die Werte der Parameter gegen den aktuellen FScore geplotted.


Next: Bibliography
Up: Praktikumsbericht RNA
Previous: Analyse mit Nussinov
Daniel Gerighausen
2013-07-19