Das zu untersuchende CASP5 Target T0163 beschreibt das Protein Glycin oxidase des Organismus B. subtilis und besitzt eine Länge von 369 Aminosäuren.
Beide Server berechnen eine Transmembranhelix des Proteins von Aminosäurenposition 7 bis 24 (Ausgabe von Smart bzw. Tmhmm).
genutzte Server: Pfam
1 vermeintlich konservierte Domaine wurde gefunden:
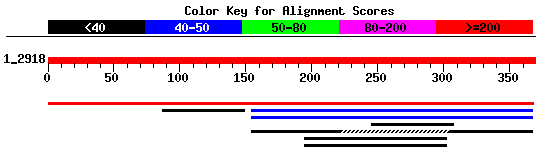
genutzte Server: blastp
Die Nutzung der Domaine DadA erzeugt 3 signifikante Alignments.
genutzte Software: Modeller 6v2
Die für den Modeller benötigten Eingabedaten
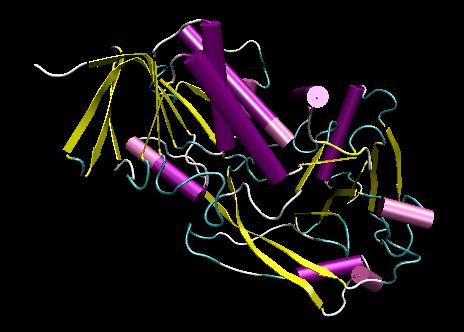
Kontrolle des erhaltenen Ergebnisses auf seine Glaubwürdigkeit:
genutzte Software: ProFit v2.2
Der Vergleich der Struktur des Originals 1NG3 (pdb-file) mit der vom Modeller berechneten Targetstruktur T0163 ergibt einen RMS-Wert von 8.488 Angstroem. Dieser ist kleiner als der Grenzwert 10, was einen guten Wert fuer die Güte des erstellten Modells bedeutet.
Da die gesuchte Struktur von T0163 bereits bekannt war, konnte die Güte der Vorhersage von Proteinstrukturen getestet werden, indem bekannte ähnliche Proteine als Modell genutzt werden. Mit dem Modell 1EL5 berechneten wir eine Proteinstruktur, die in ausreichendem Mass mit unserer gesuchten Struktur übereinstimmt.