Index >
Results for CNB 90322_0-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
90322_ENSMUSG00000045192_MOUSE_4757_5152/1-399 ATCAAAACGAGCCTTTCTCTCGAAAATTGTTCTTTTCATTCGCGAGGAGAAAGATTCTGG
90322_ENSG00000187140_HUMAN_4513_4910/1-399 ATAAAAACGAGCCTTTCTCTCGAAAATTGTTCTTTTCATCCACGAGGAGAAAGGTCCTGG
90322_ENSRNOG00000009021_RAT_3090_3484/1-399 ATCAAAACGAGCCTTTCTCTCGAAAATTGTTCTTTTCATTCGTGAGGAGAAAGATCCTGG
90322_ENSDARG00000021032_ZEBRAFISH_6596_6966/1-399 ATAAAAAGGAGCCATTTGCAGGAAAACTGTTCCTCTCATCTAGAGAGGGAAGTGTCCACG
** **** ***** ** * ***** ***** * **** * *** * * *
90322_ENSMUSG00000045192_MOUSE_4757_5152/1-399 GCAGGATCAGGGCTTAAAAATGACTTGGCATTGCAAGCTAGAGTCTTGCCGACGCAGTTC
90322_ENSG00000187140_HUMAN_4513_4910/1-399 ACAGGATCGGGGCTTAAAAATGACTTGGCATTGAAAGCCGGAGTCTTTCTGACACCGTTT
90322_ENSRNOG00000009021_RAT_3090_3484/1-399 GCAGGATCAAGGCTTAAAAATGACTTGGCATTGGAAGCTAGAGTCTTGTCTACGCTGTTC
90322_ENSDARG00000021032_ZEBRAFISH_6596_6966/1-399 TCATACTC-TGGTTAAAAAATGACTTTTCATTTAAA--TGCAATCCTT-------AATTG
** ** ** * *********** **** ** * ** * **
90322_ENSMUSG00000045192_MOUSE_4757_5152/1-399 GATGCGACACCG-CGGGCCCGGC-GTCCGCTGGGTCCCTCCAGGCTAACGGTAGCTGCTG
90322_ENSG00000187140_HUMAN_4513_4910/1-399 GGAGCGATGCGGCCAGGCCCGGCTGTCCGCTGGGTCCCTCCAGGCTACCGGCAGCTCGAG
90322_ENSRNOG00000009021_RAT_3090_3484/1-399 GATGCGACACCG-CGGGCCCGGC-GTCCGCTGGGTCCCTCCAGGCTAACGGTAGCTGCTG
90322_ENSDARG00000021032_ZEBRAFISH_6596_6966/1-399 GAT---TTTCTA-CGG--TCGAC--ACCG-TGATTCCC-CAAGGCTATACCCACCAGGAG
* * * * ** * *** ** **** * ****** * * *
90322_ENSMUSG00000045192_MOUSE_4757_5152/1-399 G-AGAAGCCGAGGGCTGGGTTTTTAGGAATTCACCATTTTCACCCCCTTCGGAGCCGACA
90322_ENSG00000187140_HUMAN_4513_4910/1-399 G-AGGAGCCCAGGGCTGGGTTTTTAGGAATTCACCATTTTCACCCCCTTCGGAGCCGACA
90322_ENSRNOG00000009021_RAT_3090_3484/1-399 G-AGAAGCCGAGGGCTGGGTTTTTAGGAATTCACCATTTTCACCCCCTTCGGAGCCGACA
90322_ENSDARG00000021032_ZEBRAFISH_6596_6966/1-399 GCAGGATCTCCCG--TTTTTCTCTCTGAATTCACCATTTTCACCCTCTT-GAAGAATCCA
* ** * * * * * * * ******************* *** * ** **
90322_ENSMUSG00000045192_MOUSE_4757_5152/1-399 CTTCATCACGCGGGATCATTCTGTCAAACAATATGATGCTGCTCATTTTTATCTGTCCCG
90322_ENSG00000187140_HUMAN_4513_4910/1-399 CTTCATCATGCGAGATCATTCTGTCAAACAATATGATGCTGCTCATTTTTATCTGTCCCG
90322_ENSRNOG00000009021_RAT_3090_3484/1-399 CTTCATCACGCGGGATCATTCTGTCAAACAATATGATGCTGCTCATTTTTATCTGTCCCG
90322_ENSDARG00000021032_ZEBRAFISH_6596_6966/1-399 CTCTATCAACCTGCAACTTTCTGTCAAACAATATGATCCAGCTCATTTTTAACAGTCCC-
** **** * * * ******************* * *********** * *****
90322_0-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 76.72
Mean single sequence MFE: -95.86
Consensus MFE: -54.61
Energy contribution: -59.92
Covariance contribution: 5.31
Mean z-score: -2.03
Structure conservation index: 0.57
SVM decision value: 0.64
SVM RNA-class probability: 0.808937
Prediction: RNA
######################################################################
>90322_ENSMUSG00000045192_MOUSE_4757_5152/1-399
AUCAAAACGAGCCUUUCUCUCGAAAAUUGUUCUUUUCAUUCGCGAGGAGAAAGAUUCUGGGCAGGAUCAGGGCUUAAAAAUGACUUGGCAUUGCAAGCUAGAGUCUUGCCGACGCAGUUCGAUGCGACACCGCGGGCCCGGCGUCCGCUGGGUCCCUCCAGGCUAACGGUAGCUGCUGGAGAAGCCGAGGGCUGGGUUUUUAGGAAUUCACCAUUUUCACCCCCUUCGGAGCCGACACUUCAUCACGCGGGAUCAUUCUGUCAAACAAUAUGAUGCUGCUCAUUUUUAUCUGUCCCG
........((..(((((((..(((((......)))))...(....))))))))..))(((((((.((((.(((........((((((((.......)))).))))..)))((((((......)))((..(((((((((((((....)))))))))((((((((((....))))..))))))...(((((((..((((.....((......))......))))))))))).................))))..)).....)))........)))).)))))))............... ( -104.20)
>90322_ENSG00000187140_HUMAN_4513_4910/1-399
AUAAAAACGAGCCUUUCUCUCGAAAAUUGUUCUUUUCAUCCACGAGGAGAAAGGUCCUGGACAGGAUCGGGGCUUAAAAAUGACUUGGCAUUGAAAGCCGGAGUCUUUCUGACACCGUUUGGAGCGAUGCGGCCAGGCCCGGCUGUCCGCUGGGUCCCUCCAGGCUACCGGCAGCUCGAGGAGGAGCCCAGGGCUGGGUUUUUAGGAAUUCACCAUUUUCACCCCCUUCGGAGCCGACACUUCAUCAUGCGAGAUCAUUCUGUCAAACAAUAUGAUGCUGCUCAUUUUUAUCUGUCCCG
.......((((.......)))).......((((((((......)))))))).((((((....))))))(((((.(((((((((((((.(((((((((((((.((((..(((.((.(((.....))).)))))..))))))))))(((..(((((..(((((.((((......))))...)))))..)))))((((((((.....((......))......)))).(....))))))))..))))..)))))))(((((..((.....))..))))).....)))))))))...))))). ( -107.10)
>90322_ENSRNOG00000009021_RAT_3090_3484/1-399
AUCAAAACGAGCCUUUCUCUCGAAAAUUGUUCUUUUCAUUCGUGAGGAGAAAGAUCCUGGGCAGGAUCAAGGCUUAAAAAUGACUUGGCAUUGGAAGCUAGAGUCUUGUCUACGCUGUUCGAUGCGACACCGCGGGCCCGGCGUCCGCUGGGUCCCUCCAGGCUAACGGUAGCUGCUGGAGAAGCCGAGGGCUGGGUUUUUAGGAAUUCACCAUUUUCACCCCCUUCGGAGCCGACACUUCAUCACGCGGGAUCAUUCUGUCAAACAAUAUGAUGCUGCUCAUUUUUAUCUGUCCCG
........(((((((..............(((((((((....))))))))).((((((....)))))))))))))((((((((.((((((((((((((((((......)))).))).))))))(((....)))(((((((((....)))))))))((((((((((....))))..))))))...(((((((..((((.....((......))......))))))))))).)))))...........((((.(((((..((.....))..))))).)))))))))))).......... ( -107.90)
>90322_ENSDARG00000021032_ZEBRAFISH_6596_6966/1-399
AUAAAAAGGAGCCAUUUGCAGGAAAACUGUUCCUCUCAUCUAGAGAGGGAAGUGUCCACGUCAUACUCUGGUUAAAAAAUGACUUUUCAUUUAAAUGCAAUCCUUAAUUGGAUUUUCUACGGUCGACACCGUGAUUCCCCAAGGCUAUACCCACCAGGAGGCAGGAUCUCCCGUUUUUCUCUCUGAAUUCACCAUUUUCACCCUCUUGAAGAAUCCACUCUAUCAACCUGCAACUUUCUGUCAAACAAUAUGAUCCAGCUCAUUUUUAACAGUCCC
.((((((.((((...(((((((.......((((((((.....)))))))).(((((..(((.........((....((((((....))))))....))(((((......)))))....)))...))))).(((((((.....((......))..(((((((...((..........(((.....)))..........))..)))))))..)))).)))........)))))))......((((.......))))...)))).))))))........ ( -64.25)
>consensus
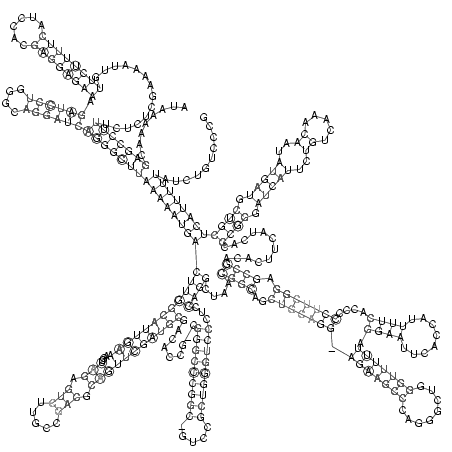
AUAAAAACGAGCCUUUCUCUCGAAAAUUGUUCUUUUCAUCCACGAGGAGAAAGAUCCUGGGCAGGAUCAGGGCUUAAAAAUGACUUGGCAUUGAAAGCUAGAGUCUUGCCGACGCAGUUCGAUGCGACACCG_CGGGCCCGGC_GUCCGCUGGGUCCCUCCAGGCUAACGGCAGCUGCAGG_AGAAGCCCAGGGCUGGGUUUUUAGGAAUUCACCAUUUUCACCCCCUUCGGAGCCGACACUUCAUCACGCGGGAUCAUUCUGUCAAACAAUAUGAUGCUGCUCAUUUUUAUCUGUCCCG
........(((((((..............((((((((......)))))))).((((((....)))))))))))))((((((((((((((((((((...(((.(((.....))).))))))))))).........(((((((((.....)))))))))...))))....((((..(((((((.(((((((........))))))).((((........))))....))))))).))))............((((.(((((..((.....))..))))).)))))))))))).......... (-54.61 = -59.92 + 5.31)
90322_0-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004