Index >
Results for CNB 71921_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
71921_ENSMUSG00000054925_MOUSE_342_706/1-367 -CTCACTTGGAGCTGGTGTACTTGGTGACTGCCTTGGTGCCCTCCGACACCGCGTGCTTG
71921_ENSDARG00000022900_ZEBRAFISH_289_654/1-367 -TTCACTTGGAGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTG
71921_ENSRNOG00000021192_RAT_336_700/1-367 -CTCACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACAGCGTGCTTG
71921_SINFRUG00000141037_FUGU_312_677/1-367 CTCTATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTG
71921_ENSG00000184825_HUMAN_290_654/1-367 -TTTACTTAGCGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCGGACACGGCGTGCTTG
71921_ENSMUSG00000054925_MOUSE_342_706/1-367 GCCAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTC
71921_ENSDARG00000022900_ZEBRAFISH_289_654/1-367 GCCAGTTCTCCGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTG
71921_ENSRNOG00000021192_RAT_336_700/1-367 GCCAACTCCCCGGGAAGCAACAGACGCACCGCCGTCTGGATCTCCCGGGACGTGATGGTC
71921_SINFRUG00000141037_FUGU_312_677/1-367 GCCAGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTG
71921_ENSG00000184825_HUMAN_290_654/1-367 GCCAACTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCTGGAGGTGATGGTC
71921_ENSMUSG00000054925_MOUSE_342_706/1-367 GAGCGCTTGTTGTAATGCGCCAGGCGGGAAGCCTCGCTCGCGATGCGCTCGAAGATGTCG
71921_ENSDARG00000022900_ZEBRAFISH_289_654/1-367 GAGCGCTTGTTGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCG
71921_ENSRNOG00000021192_RAT_336_700/1-367 GAGCGCTTGTTGTAATGAGCCAGGCGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCG
71921_SINFRUG00000141037_FUGU_312_677/1-367 GCGCGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCG
71921_ENSG00000184825_HUMAN_290_654/1-367 GAGCGCTTGTTGTAATGCGCCAGGCGGGAAGCCTCACCCGCGATGCGTTCGAAGATGTCG
71921_ENSMUSG00000054925_MOUSE_342_706/1-367 TTCACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACT
71921_ENSDARG00000022900_ZEBRAFISH_289_654/1-367 TTTACAAATGAGTTCATAATGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACT
71921_ENSRNOG00000021192_RAT_336_700/1-367 TTCACGAACGAGTTCATTATGCCCATGGCCTTGGAAGAGATGCCCGTGTCCGGATGCACT
71921_SINFRUG00000141037_FUGU_312_677/1-367 TTCACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACC
71921_ENSG00000184825_HUMAN_290_654/1-367 TTGACGAAGGAGTTCATGATTCCCATGGCCTTAGAGGAGATGCCGGTGTCGGGGTGGACC
71921_ENSMUSG00000054925_MOUSE_342_706/1-367 TGCTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTC
71921_ENSDARG00000022900_ZEBRAFISH_289_654/1-367 TGTTTTAGTACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTC
71921_ENSRNOG00000021192_RAT_336_700/1-367 TGCTTAAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTC
71921_SINFRUG00000141037_FUGU_312_677/1-367 TGCTTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTC
71921_ENSG00000184825_HUMAN_290_654/1-367 TGCTTCAGCACCTTGTACACGTATACGGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTC
71921_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 85.77
Mean single sequence MFE: -128.17
Consensus MFE: -96.34
Energy contribution: -99.42
Covariance contribution: 3.08
Mean z-score: -1.58
Structure conservation index: 0.75
SVM decision value: 0.05
SVM RNA-class probability: 0.560909
Prediction: RNA
######################################################################
>71921_ENSMUSG00000054925_MOUSE_342_706/1-367
CUCACUUGGAGCUGGUGUACUUGGUGACUGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUC
...((((((.(...((((((..((((.(((....(((((.((((((((((((((.(((..((((..((..((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))....(((..((((((....)))))).)))(((.(((((......(((....))).))))).)))..)))))..))..))))..)))))).))))))))))).))))).....)))))))..))))))...).))))))((((........))))(((....)))... ( -140.00)
>71921_ENSDARG00000022900_ZEBRAFISH_289_654/1-367
UUCACUUGGAGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUC
..............((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..)))))))..... ( -112.76)
>71921_ENSRNOG00000021192_RAT_336_700/1-367
CUCACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACAGCGUGCUUGGCCAACUCCCCGGGAAGCAACAGACGCACCGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGAGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUUAUGCCCAUGGCCUUGGAAGAGAUGCCCGUGUCCGGAUGCACUUGCUUAAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUC
...((((((.(...((((((..((((...((...(((((..((((((((.((((.(((..((((...(((((((.....((((((......))))))....)))))))..((.((((((((((((..(((((...(.((.(((((...))))).))).))))))))))))).((..(((((....)))))..)).......)))).)).))))..)))))))..))))))))..))))).)).....))))..))))))...).))))))((((........))))(((....)))... ( -126.00)
>71921_SINFRUG00000141037_FUGU_312_677/1-367
CUCUAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUC
((((....))))...((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))..(((((((((((..(........)...))))))))))). ( -126.40)
>71921_ENSG00000184825_HUMAN_290_654/1-367
UUUACUUAGCGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAACUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCACCCGCGAUGCGUUCGAAGAUGUCGUUGACGAAGGAGUUCAUGAUUCCCAUGGCCUUAGAGGAGAUGCCGGUGUCGGGGUGGACCUGCUUCAGCACCUUGUACACGUAUACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUC
.......(((((..((((((..((((..(((...(((.(((((.((((((((((((((((........)))))).(((((((((((.((((((((...(((((....))))))))))))).)))))))))))...))))).((((.....((((...(((((((((((....)))).))))).))...(((..((((.....))))..))).))))...)))).))))).))).))))).)))....))))..))))))......((.((((((........)))))).)).))))).. ( -135.70)
>consensus
_UUCACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCACCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUAAACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUC
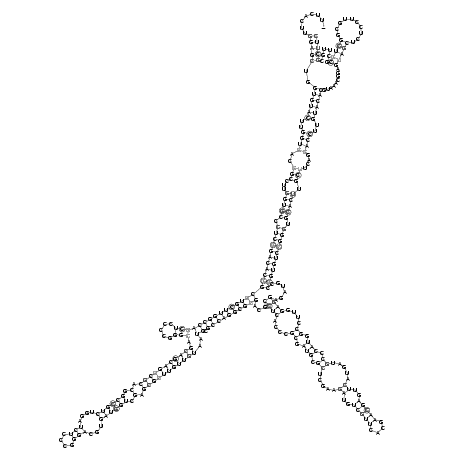
........(((((..((((((..((((..(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.(.((((((....(((....)))...)))))).).)))))))))))..)).))))))))))...((((....((.(((.((.....((..(((((....)))))..)).....)).))).))....))))......))))))))))).))))).)))....))))..)))))).........(((((..........)))))......))))) (-96.34 = -99.42 + 3.08)
71921_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004