Index >
Results for CNB 71567_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
71567_ENSDARG00000022900_ZEBRAFISH_292_669/1-379 -ACTTGGAGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTGGCC
71567_SINFRUG00000141037_FUGU_315_691/1-379 TATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGCC
71567_ENSG00000184260_HUMAN_289_665/1-379 -ACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACGGCGTGCTTGGCC
71567_ENSRNOG00000021192_RAT_339_715/1-379 -ACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACAGCGTGCTTGGCC
71567_ENSMUSG00000054813_MOUSE_327_703/1-379 -ACTTGGAGCTGGTGTACTTGGTGACAGCCTTGGTGCCCTCCGACACCGCGTGCTTGGCC
71567_ENSDARG00000022900_ZEBRAFISH_292_669/1-379 AGTTCTCCGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTGGAG
71567_SINFRUG00000141037_FUGU_315_691/1-379 AGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGGCG
71567_ENSG00000184260_HUMAN_289_665/1-379 AGCTCGCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCGCGGGATGTGATGGTGGAG
71567_ENSRNOG00000021192_RAT_339_715/1-379 AACTCCCCGGGAAGCAACAGACGCACCGCCGTCTGGATCTCCCGGGACGTGATGGTCGAG
71567_ENSMUSG00000054813_MOUSE_327_703/1-379 AGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGAG
71567_ENSDARG00000022900_ZEBRAFISH_292_669/1-379 CGCTTGTTGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCGTTT
71567_SINFRUG00000141037_FUGU_315_691/1-379 CGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGTTC
71567_ENSG00000184260_HUMAN_289_665/1-379 CGCTTGTTGTAGTGCGCCAGGCGGGAAGCCTCTCCCGCGATGCGCTCGAAGATGTCGTTG
71567_ENSRNOG00000021192_RAT_339_715/1-379 CGCTTGTTGTAATGAGCCAGGCGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGTTC
71567_ENSMUSG00000054813_MOUSE_327_703/1-379 CGCTTGTTGTAATGCGCCAGGCGGGACGCCTCGCTCGCGATGCGCTCGAAGATGTCGTTC
71567_ENSDARG00000022900_ZEBRAFISH_292_669/1-379 ACAAATGAGTTCATAATGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACTTGT
71567_SINFRUG00000141037_FUGU_315_691/1-379 ACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCTGC
71567_ENSG00000184260_HUMAN_289_665/1-379 ACGAAGGAGTTCATGATGCCCATGGCCTTGGACGAGATGCCGGTGTCGGGGTGGACCTGC
71567_ENSRNOG00000021192_RAT_339_715/1-379 ACGAACGAGTTCATTATGCCCATGGCCTTGGAAGAGATGCCCGTGTCCGGATGCACTTGC
71567_ENSMUSG00000054813_MOUSE_327_703/1-379 ACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTTGC
71567_ENSDARG00000022900_ZEBRAFISH_292_669/1-379 TTTAGTACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTCTTA
71567_SINFRUG00000141037_FUGU_315_691/1-379 TTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCTTA
71567_ENSG00000184260_HUMAN_289_665/1-379 TTCAGCACCTTGTACACGTAGATGGAGTAGCTCTCTTTGCGGCTGCGCTTGCGCTTCTTG
71567_ENSRNOG00000021192_RAT_339_715/1-379 TTAAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTG
71567_ENSMUSG00000054813_MOUSE_327_703/1-379 TTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTG
71567_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 86.44
Mean single sequence MFE: -129.19
Consensus MFE: -100.68
Energy contribution: -103.16
Covariance contribution: 2.48
Mean z-score: -1.56
Structure conservation index: 0.78
SVM decision value: 0.12
SVM RNA-class probability: 0.591168
Prediction: RNA
######################################################################
>71567_ENSDARG00000022900_ZEBRAFISH_292_669/1-379
ACUUGGAGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUCUUA
....((((...((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..)))))))..)))).. ( -113.26)
>71567_SINFRUG00000141037_FUGU_315_691/1-379
UAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCUUA
.....(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...))))))))))))... ( -125.80)
>71567_ENSG00000184260_HUMAN_289_665/1-379
ACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCGCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCGCGGGAUGUGAUGGUGGAGCGCUUGUUGUAGUGCGCCAGGCGGGAAGCCUCUCCCGCGAUGCGCUCGAAGAUGUCGUUGACGAAGGAGUUCAUGAUGCCCAUGGCCUUGGACGAGAUGCCGGUGUCGGGGUGGACCUGCUUCAGCACCUUGUACACGUAGAUGGAGUAGCUCUCUUUGCGGCUGCGCUUGCGCUUCUUG
....(((((..((((((..((((...((...(((.(((((.((((((((((.((((.((((...(((((((.(((((((((((.(.((((((.(.((((....)))).))))))).).))))))))))).(.((((...((((((......))))))...)))).)......(((((.(((......))).)))))))))..))).)))).))))))))).))))).))).))))).)).....))))..))))))((((.((.(((.((.......)).))).)).)))))))))... ( -138.60)
>71567_ENSRNOG00000021192_RAT_339_715/1-379
ACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACAGCGUGCUUGGCCAACUCCCCGGGAAGCAACAGACGCACCGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGAGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUUAUGCCCAUGGCCUUGGAAGAGAUGCCCGUGUCCGGAUGCACUUGCUUAAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
....(((((..((((((..((((...((...(((((..((((((((.((((.(((..((((...(((((((.....((((((......))))))....)))))))..((.((((((((((((..(((((...(.((.(((((...))))).))).))))))))))))).((..(((((....)))))..)).......)))).)).))))..)))))))..))))))))..))))).)).....))))..))))))(((......((((((........))))))...))))))))... ( -127.00)
>71567_ENSMUSG00000054813_MOUSE_327_703/1-379
ACUUGGAGCUGGUGUACUUGGUGACAGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
....(((((..((((((..((((..(((...(((((.(((((((((((....)..(((...((((((((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))..)))((((((((((.(((.(((....))).))))))...((..(((((....)))))..)).....)))..)))).)))).))))..))))))))))))).))))).)))....))))..))))))(((......((((((........))))))...))))))))... ( -141.30)
>consensus
_ACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUAAAUGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
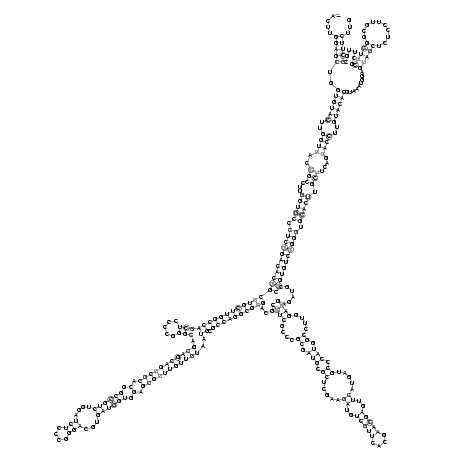
.....(((((..((((((..((((..(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.(.((((((....(((....)))...)))))).).)))))))))))..)).))))))))))...((((....((.(((.((.....((..(((((....)))))..)).....)).))).))....))))......))))))))))).))))).)))....))))..)))))).........(((((..........)))))......)))))... (-100.68 = -103.16 + 2.48)
71567_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004