Index >
Results for CNB 71547_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
71547_ENSMUSG00000054856_MOUSE_326_692/1-367 TCACTTGGAGCTGGTGTACTTGGTGACAGCCTTGGTGCCCTCCGACACGGCGTGCTTGGC
71547_ENSRNOG00000022148_RAT_303_668/1-367 -TACTTGGCGCTTGTGTACTTGGTGACCGCCTTGGTGCCCTCCGACACAGCGTGCTTAGC
71547_SINFRUG00000141037_FUGU_314_680/1-367 CTATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGC
71547_ENSG00000124518_HUMAN_318_683/1-367 TTACTTGGAGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCCGACACCGCGTGCTTGGC
71547_ENSMUSG00000054856_MOUSE_326_692/1-367 CAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGATGTGATGGTCGA
71547_ENSRNOG00000022148_RAT_303_668/1-367 CAGTTCTCCGGGCAGTAGGAGACGCACCGCCGTCTGGATCTCCCGCGATGTGATTGTAGA
71547_SINFRUG00000141037_FUGU_314_680/1-367 CAGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGGC
71547_ENSG00000124518_HUMAN_318_683/1-367 CAGCTCCCCCGGAAGCAGCAGGCGCACGGCGGTCTGGATCTCCCTGGAGGTGATGGTCGA
71547_ENSMUSG00000054856_MOUSE_326_692/1-367 GCGCTTGTTGTAATGCGCCAGGCGGGACGCCTCGCTCGCGATGCGCTCGAAGATGTCGTT
71547_ENSRNOG00000022148_RAT_303_668/1-367 CCGCTTGTTGTAATGCGCCAGGCGAGAAGCCTCGTTTGCGATGCGCTCGAAGATGTCATT
71547_SINFRUG00000141037_FUGU_314_680/1-367 GCGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGTT
71547_ENSG00000124518_HUMAN_318_683/1-367 GCGCTTGTTGTAGTGCGCCAGGCGGGAAGCCTCGCTTGCGATGCGCTCGAAGATGTCGTT
71547_ENSMUSG00000054856_MOUSE_326_692/1-367 CACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTTG
71547_ENSRNOG00000022148_RAT_303_668/1-367 GACAAACGAGTTCATGATGCCCATGGCCTTGGACGAGATGCCCGTGTCCGGATGCACCTG
71547_SINFRUG00000141037_FUGU_314_680/1-367 CACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCTG
71547_ENSG00000124518_HUMAN_318_683/1-367 GACGAAGGAGTTCATGATTCCCATGGCCTTAGAAGAGATGCCGGTGTCGGGGTGGACCTG
71547_ENSMUSG00000054856_MOUSE_326_692/1-367 CTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
71547_ENSRNOG00000022148_RAT_303_668/1-367 CTTCAGCACCTTGTACACATAGATGGAGTAGCTCTCCTTGCGACTGCGCTTGCGCTTCTT
71547_SINFRUG00000141037_FUGU_314_680/1-367 CTTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCTT
71547_ENSG00000124518_HUMAN_318_683/1-367 CTTCAGCACCTTGTACACGTACACGGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
71547_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 87.22
Mean single sequence MFE: -128.98
Consensus MFE: -100.47
Energy contribution: -104.85
Covariance contribution: 4.38
Mean z-score: -1.43
Structure conservation index: 0.78
SVM decision value: 0.04
SVM RNA-class probability: 0.552108
Prediction: RNA
######################################################################
>71547_ENSMUSG00000054856_MOUSE_326_692/1-367
UCACUUGGAGCUGGUGUACUUGGUGACAGCCUUGGUGCCCUCCGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGAUGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
......(((((..((((((..((((..(((...(((((.((((((((((((((.(((..((((..((..((((((((((((((((.((((((((.(.((((....)))).))))))))).)))))))))))....(((..((((((....)))))).)))(((.(((((......(((....))).))))).)))..)))))..))..))))..)))))))).))))))))).))))).)))....))))..))))))(((......((((((........))))))...)))))))).. ( -141.90)
>71547_ENSRNOG00000022148_RAT_303_668/1-367
UACUUGGCGCUUGUGUACUUGGUGACCGCCUUGGUGCCCUCCGACACAGCGUGCUUAGCCAGUUCUCCGGGCAGUAGGAGACGCACCGCCGUCUGGAUCUCCCGCGAUGUGAUUGUAGACCGCUUGUUGUAAUGCGCCAGGCGAGAAGCCUCGUUUGCGAUGCGCUCGAAGAUGUCAUUGACAAACGAGUUCAUGAUGCCCAUGGCCUUGGACGAGAUGCCCGUGUCCGGAUGCACCUGCUUCAGCACCUUGUACACAUAGAUGGAGUAGCUCUCCUUGCGACUGCGCUUGCGCUUCUU
.....(((((.(((((((..((((...((...(((((..((((((((.((((.(((..((((..((..((((.....(((.(((..(((.((((((.....))).))))))(((((((((.(((((.((.....)).)))))(((....))))))))))))))))))......(((((.(((......))).)))))))))..))..))))..)))))))..)))).)))).))))).)).....))))..))))))).....((((.....))))..(((....)))..))))).... ( -110.60)
>71547_SINFRUG00000141037_FUGU_314_680/1-367
CUAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCUU
......(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...)))))))))))).. ( -125.80)
>71547_ENSG00000124518_HUMAN_318_683/1-367
UUACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCCGGAAGCAGCAGGCGCACGGCGGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAGUGCGCCAGGCGGGAAGCCUCGCUUGCGAUGCGCUCGAAGAUGUCGUUGACGAAGGAGUUCAUGAUUCCCAUGGCCUUAGAAGAGAUGCCGGUGUCGGGGUGGACCUGCUUCAGCACCUUGUACACGUACACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
......(((((((((((((.(((((.(((....((...)).))).))))).))))...)))))))))...((((((.((((((((((.((((..((((((((....(((((.((((((((((((..(((((((.(((..((((.....)))))))))))))))))))))).((.(((((.(((......))).))))))))))).)))))....))))).)))(((((((((((...(((...)))))))))).))))......((((.....)))))))).).))))))))))))))). ( -137.60)
>consensus
UUACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGAGGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCGCUUGCGAUGCGCUCGAAGAUGUCGUUCACAAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCCGUGUCGGGGUGCACCUGCUUCAGCACCUUGUACACGUACACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
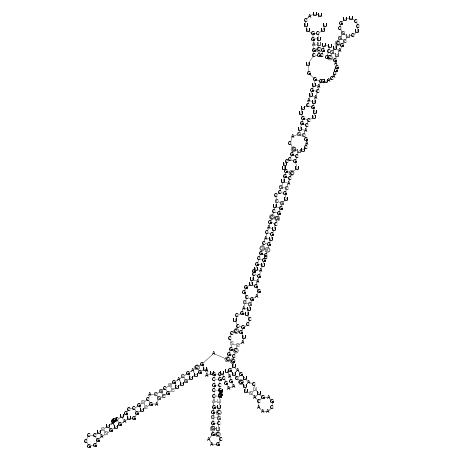
......(((((..((((((..((((..(((...(((((.((((((((((((((.(((..((((..((..((((.(((((((((((.((((.(((...(((((....)))))))).)))).)))))))))))...((((((((((((....))))))))....))))........(((((.(((......))).)))))))))..))..))))..)))))).))))))))))).))))).)))....))))..)))))).........((((((........))))))......))))).. (-100.47 = -104.85 + 4.38)
71547_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004