Index >
Results for CNB 71528_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
71528_ENSMUSG00000049150_MOUSE_327_706/1-381 CACTTGGAGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCCGACACCGCGTGCTTGGCC
71528_SINFRUG00000141037_FUGU_315_694/1-381 TATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGCC
71528_ENSG00000183868_HUMAN_301_681/1-381 TACTTAGCGCTGGTGTACTTGGTGACGGCCTTAGTACCCTCGGACACGGCGTGCTTGGCC
71528_ENSRNOG00000028264_RAT_327_705/1-381 -ACTTGGAGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCGGACACCGCGTGCTTGGCC
71528_ENSMUSG00000049150_MOUSE_327_706/1-381 AGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGAG
71528_SINFRUG00000141037_FUGU_315_694/1-381 AGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGGCG
71528_ENSG00000183868_HUMAN_301_681/1-381 AACTCCCCAGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCTGGAGGTGATGGTCGAG
71528_ENSRNOG00000028264_RAT_327_705/1-381 AGCTCCCCGGGAAGCAGCAGGCGCACAGCCGTCTGGACCTCCCGGGACGTGATGGTCGAG
71528_ENSMUSG00000049150_MOUSE_327_706/1-381 CGCTTGTTGTAATGCGCCAGGCGGGACGCCTCGCTCGCGATGCGCTCGAAGATGTCGTTC
71528_SINFRUG00000141037_FUGU_315_694/1-381 CGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGTTC
71528_ENSG00000183868_HUMAN_301_681/1-381 CGCTTGTTGTAATGCGCCAGGCGGGAAGCCTCACCTGCGATGCGCTCGAAAATGTCGTTC
71528_ENSRNOG00000028264_RAT_327_705/1-381 CGCTTGTTGTAATGCGCCAGGCGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGTTC
71528_ENSMUSG00000049150_MOUSE_327_706/1-381 ACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTTGC
71528_SINFRUG00000141037_FUGU_315_694/1-381 ACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCTGC
71528_ENSG00000183868_HUMAN_301_681/1-381 ACAAACGAATTCATGATGCCCATGGCCTTGGACGAAATGCCGGTGTCAGGGTGGACCTGC
71528_ENSRNOG00000028264_RAT_327_705/1-381 ACGAACGAGTTCATGATGCCCATGGCCTTGGAAGAGATGCCCGTGTCCGGGTGCACTTGC
71528_ENSMUSG00000049150_MOUSE_327_706/1-381 TTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTG
71528_SINFRUG00000141037_FUGU_315_694/1-381 TTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCTTA
71528_ENSG00000183868_HUMAN_301_681/1-381 TTCAGAACCTTGTACACATAGATGGAATAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTG
71528_ENSRNOG00000028264_RAT_327_705/1-381 TTAAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTG
71528_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 88.67
Mean single sequence MFE: -131.65
Consensus MFE: -107.90
Energy contribution: -112.78
Covariance contribution: 4.88
Mean z-score: -1.55
Structure conservation index: 0.82
SVM decision value: 0.28
SVM RNA-class probability: 0.667722
Prediction: RNA
######################################################################
>71528_ENSMUSG00000049150_MOUSE_327_706/1-381
CACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
.....(((((((((((((.(((((.(((....((...)).))).))))).))))...))))))))).((((.(((.(((((((((..(((((..(((((((((((((((..((..((((((((..(((((...(((..(((((...))))))))..)))))))))))))..))..))))).))..((((..((((.....))))..))))..)))))(((((((((((..(((((..(((....)))...))))).)).)))))).)))....)))..))))))))))))))))).)))) ( -142.60)
>71528_SINFRUG00000141037_FUGU_315_694/1-381
UAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCUUA
.....(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...))))))))))))... ( -125.80)
>71528_ENSG00000183868_HUMAN_301_681/1-381
UACUUAGCGCUGGUGUACUUGGUGACGGCCUUAGUACCCUCGGACACGGCGUGCUUGGCCAACUCCCCAGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCACCUGCGAUGCGCUCGAAAAUGUCGUUCACAAACGAAUUCAUGAUGCCCAUGGCCUUGGACGAAAUGCCGGUGUCAGGGUGGACCUGCUUCAGAACCUUGUACACAUAGAUGGAAUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
......((.(((((((((((((.((.((((..(((((((........)).))))).))))...)).)))))..(((((((((((.((((((((...(((((....))))))))))))).)))))))))))..))))))))))(((((((.......((((.((((.........((((((((....)...(((((.....)))))...)))))))...((((((((((((((.((......))...)))))).))))......(((.......)))...)))).))))))))))))))). ( -122.71)
>71528_ENSRNOG00000028264_RAT_327_705/1-381
ACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACCGCGUGCUUGGCCAGCUCCCCGGGAAGCAGCAGGCGCACAGCCGUCUGGACCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAAGAGAUGCCCGUGUCCGGGUGCACUUGCUUAAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
....(((((((((((((.(((((....((..((...))..)).))))).))))...))))))))).((((((((.(((((((((..(((((..(((.....(((((((..((..((((((((..(((((...(((..(((((...))))))))..)))))))))))))..))..))))).))..((((..((((.....))))..))))...(((.((((.((((.((.(((((..(((....)))...))))).)).)))).).))).))))))..)))))))))))))))))))))) ( -135.50)
>consensus
UACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACAGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCACCCGCGAUGCGCUCGAAGAUGUCGUUCACAAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCCGUGUCAGGGUGCACCUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
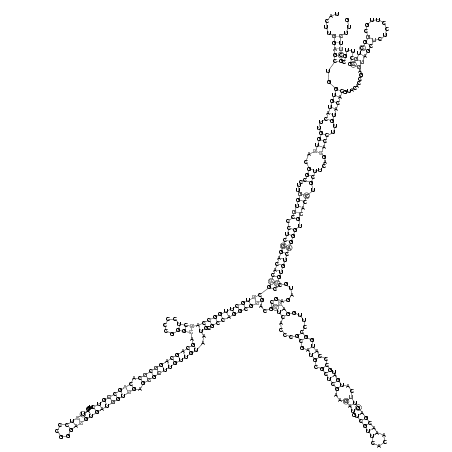
.....(((((..((((((..((((..(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.((((((((...(((((....))))))))))))).)))))))))))..)).))))))))))...((((....((.(((.(((((..(((.(((((....))))))))..))).)).))).))....))))......))))))))))).))))).)))....))))..)))))).........((((((........))))))......)))))... (-107.90 = -112.78 + 4.88)
71528_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004