Index >
Results for CNB 71177_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
71177_ENSG00000184260_HUMAN_287_665/1-381 -TCACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACGGCGTGCTTGG
71177_ENSDARG00000022900_ZEBRAFISH_289_669/1-381 TTCACTTGGAGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTGG
71177_ENSMUSG00000054925_MOUSE_343_721/1-381 -TCACTTGGAGCTGGTGTACTTGGTGACTGCCTTGGTGCCCTCCGACACCGCGTGCTTGG
71177_ENSRNOG00000027856_RAT_334_712/1-381 -TCACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACAGCGTGCTTGG
71177_SINFRUG00000141037_FUGU_314_691/1-381 CT-ATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGG
71177_ENSG00000184260_HUMAN_287_665/1-381 CCAGCTCGCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCGCGGGATGTGATGGTGG
71177_ENSDARG00000022900_ZEBRAFISH_289_669/1-381 CCAGTTCTCCGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTGG
71177_ENSMUSG00000054925_MOUSE_343_721/1-381 CCAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCG
71177_ENSRNOG00000027856_RAT_334_712/1-381 CCAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCG
71177_SINFRUG00000141037_FUGU_314_691/1-381 CCAGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGG
71177_ENSG00000184260_HUMAN_287_665/1-381 AGCGCTTGTTGTAGTGCGCCAGGCGGGAAGCCTCTCCCGCGATGCGCTCGAAGATGTCGT
71177_ENSDARG00000022900_ZEBRAFISH_289_669/1-381 AGCGCTTGTTGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCGT
71177_ENSMUSG00000054925_MOUSE_343_721/1-381 AGCGCTTGTTGTAATGCGCCAGGCGGGAAGCCTCGCTCGCGATGCGCTCGAAGATGTCGT
71177_ENSRNOG00000027856_RAT_334_712/1-381 AGCGCTTGTTGTAATGCGCTAGACGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGT
71177_SINFRUG00000141037_FUGU_314_691/1-381 CGCGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGT
71177_ENSG00000184260_HUMAN_287_665/1-381 TGACGAAGGAGTTCATGATGCCCATGGCCTTGGACGAGATGCCGGTGTCGGGGTGGACCT
71177_ENSDARG00000022900_ZEBRAFISH_289_669/1-381 TTACAAATGAGTTCATAATGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACTT
71177_ENSMUSG00000054925_MOUSE_343_721/1-381 TCACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTT
71177_ENSRNOG00000027856_RAT_334_712/1-381 TCACGAACGAGTTCATGATGCCCATGGCCTTAGAAGAGATGCCCGTGTCCGGGTGCACTT
71177_SINFRUG00000141037_FUGU_314_691/1-381 TCACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCT
71177_ENSG00000184260_HUMAN_287_665/1-381 GCTTCAGCACCTTGTACACGTAGATGGAGTAGCTCTCTTTGCGGCTGCGCTTGCGCTTCT
71177_ENSDARG00000022900_ZEBRAFISH_289_669/1-381 GTTTTAGTACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTCT
71177_ENSMUSG00000054925_MOUSE_343_721/1-381 GCTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCT
71177_ENSRNOG00000027856_RAT_334_712/1-381 GCTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCT
71177_SINFRUG00000141037_FUGU_314_691/1-381 GCTTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCT
71177_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 87.05
Mean single sequence MFE: -129.57
Consensus MFE: -102.36
Energy contribution: -104.16
Covariance contribution: 1.80
Mean z-score: -1.45
Structure conservation index: 0.79
SVM decision value: -0.03
SVM RNA-class probability: 0.517242
Prediction: RNA
######################################################################
>71177_ENSG00000184260_HUMAN_287_665/1-381
UCACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCGCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCGCGGGAUGUGAUGGUGGAGCGCUUGUUGUAGUGCGCCAGGCGGGAAGCCUCUCCCGCGAUGCGCUCGAAGAUGUCGUUGACGAAGGAGUUCAUGAUGCCCAUGGCCUUGGACGAGAUGCCGGUGUCGGGGUGGACCUGCUUCAGCACCUUGUACACGUAGAUGGAGUAGCUCUCUUUGCGGCUGCGCUUGCGCUUCU
......(((((..((((((..((((...((...(((.(((((.((((((((((.((((.((((...(((((((.(((((((((((.(.((((((.(.((((....)))).))))))).).))))))))))).(.((((...((((((......))))))...)))).)......(((((.(((......))).)))))))))..))).)))).))))))))).))))).))).))))).)).....))))..))))))((((.((.(((.((.......)).))).)).))))))))). ( -138.60)
>71177_ENSDARG00000022900_ZEBRAFISH_289_669/1-381
UUCACUUGGAGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUCU
.......((((...((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..)))))))..)))) ( -113.16)
>71177_ENSMUSG00000054925_MOUSE_343_721/1-381
UCACUUGGAGCUGGUGUACUUGGUGACUGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCU
......(((((..((((((..((((.(((....(((((.((((((((((((((.(((..((((..((..((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))....(((..((((((....)))))).)))(((.(((((......(((....))).))))).)))..)))))..))..))))..)))))).))))))))))).))))).....)))))))..))))))(((......((((((........))))))...)))))))). ( -141.00)
>71177_ENSRNOG00000027856_RAT_334_712/1-381
UCACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACAGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCUAGACGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUAGAAGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCU
......(((((..((((((..((((...((...(((((.(((((((((.((((.((((((((((..(.(((((.(((((((((((.((((((((((..((....))..)).)))))))).)))))))))))..((((.....)))).......))))).)..))(((((..((..(((((....)))))..)).))).))...)))))....)))..))))..))))))))).))))).)).....))))..))))))(((......((((((........))))))...)))))))). ( -129.30)
>71177_SINFRUG00000141037_FUGU_314_691/1-381
CUAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCU
......(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...)))))))))))). ( -125.80)
>consensus
_UCACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUAAAUGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCU
.......(((((..((((((..((((...((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.(.((((((....(((....)))...)))))).).)))))))))))..)).))))))))))...((((....((.(((.((.....((..(((((....)))))..)).....)).))).))....))))......))))))))))).))))).)).....))))..)))))).........(((((..........)))))......))))). (-102.36 = -104.16 + 1.80)
71177_0.rnaz
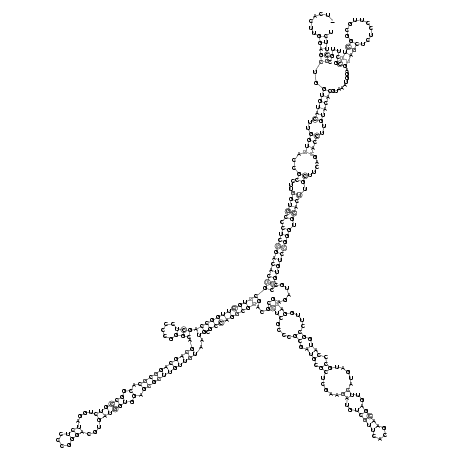
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004