Index >
Results for CNB 70688_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
70688_SINFRUG00000141037_FUGU_322_661/1-341 AGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGCCAGCTCCC
70688_ENSRNOG00000027815_RAT_9323_9661/1-341 AGCTGGTCTACTTGGTGACGGCCTTGGTGCCCTCGGACACCGCCTGCTTGGCCAGCTCCC
70688_ENSDARG00000022900_ZEBRAFISH_298_637/1-341 AGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTGGCCAGTTCTC
70688_ENSMUSG00000049150_MOUSE_334_673/1-341 AGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCCGACACCGCGTGCTTGGCCAGCTCCC
70688_ENSG00000183868_HUMAN_309_647/1-341 -GCTGGTGTACTTGGTGACGGCCTTAGTACCCTCGGACACGGCGTGCTTGGCCAACTCCC
70688_SINFRUG00000141037_FUGU_322_661/1-341 CCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGGCGCGCTTGT
70688_ENSRNOG00000027815_RAT_9323_9661/1-341 CGAGTAGTAGCAGGCGCACGGCCGTCTGGACCTCCCGGGACGTGATGGTCGAGCGCTTGT
70688_ENSDARG00000022900_ZEBRAFISH_298_637/1-341 CGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTGGAGCGCTTGT
70688_ENSMUSG00000049150_MOUSE_334_673/1-341 CGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGAGCGCTTGT
70688_ENSG00000183868_HUMAN_309_647/1-341 CAGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCTGGAGGTGATGGTCGAGCGCTTGT
70688_SINFRUG00000141037_FUGU_322_661/1-341 TGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGTTCACAAACG
70688_ENSRNOG00000027815_RAT_9323_9661/1-341 TGTAATGAGGCAGGCGCGACGCCTCGCCCGCGATG-GCTCGAAGATGTCGTTCACGAACG
70688_ENSDARG00000022900_ZEBRAFISH_298_637/1-341 TGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCGTTTACAAATG
70688_ENSMUSG00000049150_MOUSE_334_673/1-341 TGTAATGCGCCAGGCGGGACGCCTCGCTCGCGATGCGCTCGAAGATGTCGTTCACGAACG
70688_ENSG00000183868_HUMAN_309_647/1-341 TGTAATGCGCCAGGCGGGAAGCCTCACCTGCGATGCGCTCGAAAATGTCGTTCACAAACG
70688_SINFRUG00000141037_FUGU_322_661/1-341 AGTTCATGA-TGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCTGCTTCAGC
70688_ENSRNOG00000027815_RAT_9323_9661/1-341 AGTTCATAAATGCCCATGGCCTTAGAAGAGATGCCAGTGTCCGGGTGCACTTGCTTAAGC
70688_ENSDARG00000022900_ZEBRAFISH_298_637/1-341 AGTTCATAA-TGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACTTGTTTTAGT
70688_ENSMUSG00000049150_MOUSE_334_673/1-341 AGTTCATGA-TGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTTGCTTCAGC
70688_ENSG00000183868_HUMAN_309_647/1-341 AATTCATGA-TGCCCATGGCCTTGGACGAAATGCCGGTGTCAGGGTGGACCTGCTTCAGA
70688_SINFRUG00000141037_FUGU_322_661/1-341 ACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCTTATCTCCT
70688_ENSRNOG00000027815_RAT_9323_9661/1-341 ACCTTGTACACGTACACCGAGTAGCTCTCCTTGGGGCTGCGCTTGCGCTTCTAGCCGTCC
70688_ENSDARG00000022900_ZEBRAFISH_298_637/1-341 ACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTCTTATCCCCC
70688_ENSMUSG00000049150_MOUSE_334_673/1-341 ACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTGCCGTCC
70688_ENSG00000183868_HUMAN_309_647/1-341 ACCTTGTACACATAGATGGAATAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTGCCGTCT
70688_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 84.50
Mean single sequence MFE: -127.27
Consensus MFE: -90.78
Energy contribution: -93.26
Covariance contribution: 2.48
Mean z-score: -1.60
Structure conservation index: 0.71
SVM decision value: 0.00
SVM RNA-class probability: 0.534951
Prediction: RNA
######################################################################
>70688_SINFRUG00000141037_FUGU_322_661/1-341
AGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCUUAUCUCCU
.....((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))(.(((((((((((..(........)...))))))))))))......... ( -124.80)
>70688_ENSRNOG00000027815_RAT_9323_9661/1-341
AGCUGGUCUACUUGGUGACGGCCUUGGUGCCCUCGGACACCGCCUGCUUGGCCAGCUCCCCGAGUAGUAGCAGGCGCACGGCCGUCUGGACCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGAGGCAGGCGCGACGCCUCGCCCGCGAUGGCUCGAAGAUGUCGUUCACGAACGAGUUCAUAAAUGCCCAUGGCCUUAGAAGAGAUGCCAGUGUCCGGGUGCACUUGCUUAAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGGGGCUGCGCUUGCGCUUCUAGCCGUCC
.((..(.....)..))((((((...(((((.(((((((((.((((((((((........))))))))((((((((((.((((((((((..((....))..)).)))))))).))))))))))...((((((.(((((((.((((((....))).))))))..((..(((((....)))))..)).....))))....))))))........))..))))))))).))))).((.(((((....((((....))))....((((((((....))))))))))))).)).....)))))). ( -131.50)
>70688_ENSDARG00000022900_ZEBRAFISH_298_637/1-341
AGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUCUUAUCCCCC
.....((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..))))))).............. ( -112.76)
>70688_ENSMUSG00000049150_MOUSE_334_673/1-341
AGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUGCCGUCC
(((..((((((..((((..(((...(((((.(((((((((((....)..(((...((((((((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))..)))((((((((((.(((.(((....))).))))))...((..(((((....)))))..)).....)))..)))).)))).))))..))))))))))))).))))).)))....))))..)))))).(((.....))))))......(((((.(((....))).....))))).. ( -140.50)
>70688_ENSG00000183868_HUMAN_309_647/1-341
GCUGGUGUACUUGGUGACGGCCUUAGUACCCUCGGACACGGCGUGCUUGGCCAACUCCCCAGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCACCUGCGAUGCGCUCGAAAAUGUCGUUCACAAACGAAUUCAUGAUGCCCAUGGCCUUGGACGAAAUGCCGGUGUCAGGGUGGACCUGCUUCAGAACCUUGUACACAUAGAUGGAAUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUGCCGUCU
...((.(((((.(((....)))..)))))))..((((..(((((((((((........)))))).(((((((((((.((((((((...(((((....))))))))))))).)))))))))))...))))).((((((((((.......((((.((((.........((((((((....)...(((((.....)))))...)))))))...((((((((((((((.((......))...)))))).))))......(((.......)))...)))).)))))))))))))))))))))) ( -126.81)
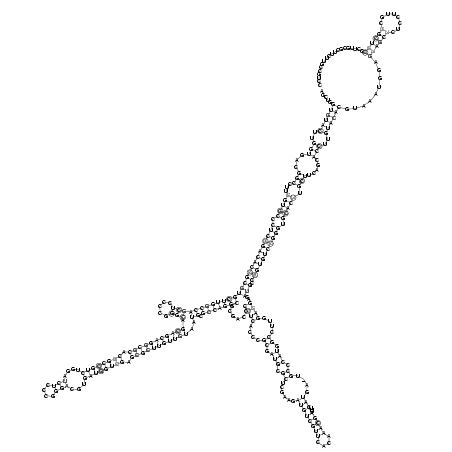
>consensus
AGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCACCCGCGAUGCGCUCGAAGAUGUCGUUCACAAACGAGUUCAUGA_UGCCCAUGGCCUUGGAGGAGAUGCCAGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUAAAUGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUGCCGUCC
.....((((((..(((...(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.((((((((....(((....)))...)))))))).)))))))))))..)).)))))))......((((....((.(((.((......((((((((....)))))...)))....)).))).))....))))..))).))))))))))).))))).))).....)))..)))))).........((((((........)))))).................... (-90.78 = -93.26 + 2.48)
70688_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004