Index >
Results for CNB 54448-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
54448_ENSG00000172046_HUMAN_5797_6065/1-272 AATTCTCACCGAGGCCAGGTGTGTCTCTGGCTTTGGGGTTACATGCTCCATGGGTGTGCG
54448_ENSDARG00000004132_ZEBRAFISH_4598_4863/1-272 -ATTATTACCGTGGCAACAGCAGGTTCTTGTTTGAGGGACACATGTTCGGAAACTGAGCG
54448_ENSMUSG00000006676_MOUSE_7928_8194/1-272 -ATTCTTACCGAGGCCAAGTGTGGGTCTGGCTTTGGGGCTACATGCTCCAAGGGGGTGCG
54448_ENSRNOG00000016106_RAT_4364_4632/1-272 GGTTCTCACCGAGGCCAAGTGCGGTTCTGGCTTTGGGGTAACATGCTCCAAGGGGGTGCG
** * **** *** * * *** * ** *** ***** ** * ***
54448_ENSG00000172046_HUMAN_5797_6065/1-272 GGTTGCCACACTGTCTAGCCCTGTGTCCTCAGATCGTGCCTTGGATTT---ATCCTTCTC
54448_ENSDARG00000004132_ZEBRAFISH_4598_4863/1-272 TGGGGACACAGCATCCAAACCAGAGTCCTCTGGGGCTCTAGGGGGTTTGGGTTTCTCCTC
54448_ENSMUSG00000006676_MOUSE_7928_8194/1-272 GGCCACCACACCATCCAGCCCTGAGTCCTCTGATCGAGCCTTGGGTTT---TTCCTTCTC
54448_ENSRNOG00000016106_RAT_4364_4632/1-272 AGCCACCACACCATCCAGCCCCGAGTCCTCTGACCGAGCCTTGGGTTT---TTCCTTCTC
* **** ** * ** * ****** * ** *** * ** ***
54448_ENSG00000172046_HUMAN_5797_6065/1-272 CACAGCCCGGGCCTCCTCCTGGCCTGTCAGGGGGTGGGGAGCACCTCCTGGTGGGGTTGA
54448_ENSDARG00000004132_ZEBRAFISH_4598_4863/1-272 TCCAACCCGTGGTTCTTCTTTTGCAGGAAGTGCATGCTGGCTGCTTCCTGGCTGACTCTT
54448_ENSMUSG00000006676_MOUSE_7928_8194/1-272 CACAGCCCTGGCTTCCTCCTGGCCTGTCAGGGGGTGGGGGCCACCTCCTGGAGGGGTTGA
54448_ENSRNOG00000016106_RAT_4364_4632/1-272 TACAGCCCTGGCTTCCTCCTGGCCAGTCAGAGGGAGGGGGCCACCTCCTGGAGGGGTTGA
** *** * ** ** * * * ** * * * * ****** * *
54448_ENSG00000172046_HUMAN_5797_6065/1-272 ATCCAGAGGGGTTGGACCTGTCGGCACGGCAACCTTTGCACCACCCACTGCACCTTAAAA
54448_ENSDARG00000004132_ZEBRAFISH_4598_4863/1-272 TTCAAGTGTAGAAGGGCTGGAGGCCACAGCAACCTTGGCGCCCCCCACTGCACCTTAAAC
54448_ENSMUSG00000006676_MOUSE_7928_8194/1-272 ATCCAAAGGGGTTGGGCCTGTCGGCACGGCAACCTTTGCACCACCCACTGCACCTTAAAA
54448_ENSRNOG00000016106_RAT_4364_4632/1-272 ATCCAAAGGGGTTGGACCTGTCGGCACGGCAACCTTTGCACCACCCACTGCACCTTAAAA
** * * * ** * * * *** ******** ** ** ****************
54448_ENSG00000172046_HUMAN_5797_6065/1-272 AGGGGGAATGAGAGGGCTGGTGGTTAGCAGCA
54448_ENSDARG00000004132_ZEBRAFISH_4598_4863/1-272 AGG----ACAACAGAGCTTGTGGTTAGCAGC-
54448_ENSMUSG00000006676_MOUSE_7928_8194/1-272 AGGGGGAATGAGAGGGCTGGTGGTTAGCAGC-
54448_ENSRNOG00000016106_RAT_4364_4632/1-272 AGGGGGAATGAGAGGGCTGGTGGTTAGCAGCA
*** * * ** *** ************
54448-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 272
Mean pairwise identity: 75.39
Mean single sequence MFE: -116.08
Consensus MFE: -67.53
Energy contribution: -68.34
Covariance contribution: 0.81
Mean z-score: -1.94
Structure conservation index: 0.58
SVM decision value: 0.71
SVM RNA-class probability: 0.830181
Prediction: RNA
######################################################################
>54448_ENSG00000172046_HUMAN_5797_6065/1-272
AAUUCUCACCGAGGCCAGGUGUGUCUCUGGCUUUGGGGUUACAUGCUCCAUGGGUGUGCGGGUUGCCACACUGUCUAGCCCUGUGUCCUCAGAUCGUGCCUUGGAUUUAUCCUUCUCCACAGCCCGGGCCUCCUCCUGGCCUGUCAGGGGGUGGGGAGCACCUCCUGGUGGGGUUGAAUCCAGAGGGGUUGGACCUGUCGGCACGGCAACCUUUGCACCACCCACUGCACCUUAAAAAGGGGGAAUGAGAGGGCUGGUGGUUAGCAGCA
..(((((((((((((((((......)))))))))))..........(((.(((((((((((((((((((((...........)))).........(((((.((((((((.....((((((....((((((.......)))))).(((((((((.....))))))))))))))).))))))))((.(((.....))).)))))))))))))))..))).)))))).....((((....))))))).)))))).((((........)))). ( -125.40)
>54448_ENSDARG00000004132_ZEBRAFISH_4598_4863/1-272
AUUAUUACCGUGGCAACAGCAGGUUCUUGUUUGAGGGACACAUGUUCGGAAACUGAGCGUGGGGACACAGCAUCCAAACCAGAGUCCUCUGGGGCUCUAGGGGGUUUGGGUUUCUCCUCUCCAACCCGUGGUUCUUCUUUUGCAGGAAGUGCAUGCUGGCUGCUUCCUGGCUGACUCUUUUCAAGUGUAGAAGGGCUGGAGGCCACAGCAACCUUGGCGCCCCCCACUGCACCUUAAACAGGACAACAGAGCUUGUGGUUAGCAGC
............(((((.((((((((((((((((((..((...(((((....).))))((((((...((((..(((..(((((((((....))))))).))(((((.(((........))).))))).)))...........((((((((((......)).))))))))))))...........((((..((((((((.......))))..)))).)))).))))))))..)))))))))(.....)))))))))).))).))... ( -103.30)
>54448_ENSMUSG00000006676_MOUSE_7928_8194/1-272
AUUCUUACCGAGGCCAAGUGUGGGUCUGGCUUUGGGGCUACAUGCUCCAAGGGGGUGCGGGCCACCACACCAUCCAGCCCUGAGUCCUCUGAUCGAGCCUUGGGUUUUUCCUUCUCCACAGCCCUGGCUUCCUCCUGGCCUGUCAGGGGGUGGGGGCCACCUCCUGGAGGGGUUGAAUCCAAAGGGGUUGGGCCUGUCGGCACGGCAACCUUUGCACCACCCACUGCACCUUAAAAAGGGGGAAUGAGAGGGCUGGUGGUUAGCAGC
.......((((((((......(((.(((((((((((((.....))))))))).((((.((....)).))))..))))))).((....)).....).))))))).......((...((((((((((..(((((((((.((((((((((((((((...))))))))).((.(((((.(((((....))))).))))).)))))).)))......((((........))))........)))))))).)..)))))).))))..)).... ( -121.70)
>54448_ENSRNOG00000016106_RAT_4364_4632/1-272
GGUUCUCACCGAGGCCAAGUGCGGUUCUGGCUUUGGGGUAACAUGCUCCAAGGGGGUGCGAGCCACCACACCAUCCAGCCCCGAGUCCUCUGACCGAGCCUUGGGUUUUUCCUUCUCUACAGCCCUGGCUUCCUCCUGGCCAGUCAGAGGGAGGGGGCCACCUCCUGGAGGGGUUGAAUCCAAAGGGGUUGGACCUGUCGGCACGGCAACCUUUGCACCACCCACUGCACCUUAAAAAGGGGGAAUGAGAGGGCUGGUGGUUAGCAGCA
........(((..(((......)))..)))(((((((((.....))))))))).(.(((.((((((((..((.(((((((((...(((((((((.(((((..(((((.............))))).)))))((....))...)))))))))..)))))..((((((.((((((((.(((((....))))).))))(((((...))))).....((((........))))))))....))))))..)).))))..)))))))).))).). ( -113.92)
>consensus
_AUUCUCACCGAGGCCAAGUGUGGUUCUGGCUUUGGGGUUACAUGCUCCAAGGGGGUGCGGGCCACCACACCAUCCAGCCCUGAGUCCUCUGAUCGAGCCUUGGGUUU___UUCCUUCUCCACAGCCCGGGCUUCCUCCUGGCCAGUCAGGGGGUGGGGGCCACCUCCUGGAGGGGUUGAAUCCAAAGGGGUUGGACCUGUCGGCACGGCAACCUUUGCACCACCCACUGCACCUUAAAAAGGGGGAAUGAGAGGGCUGGUGGUUAGCAGC_
........(((((((((..........))))))))).......((((.....((((((........))))))....(((((((.(((((((.(....(((..(((((................))))).))).(((((((.(((...(((((((((.....)))))))))((.(((((.(((((....))))).))))).)))))............(((........))).........))))))).).)))))))))).))))))))... (-67.53 = -68.34 + 0.81)
54448-rev.rnaz
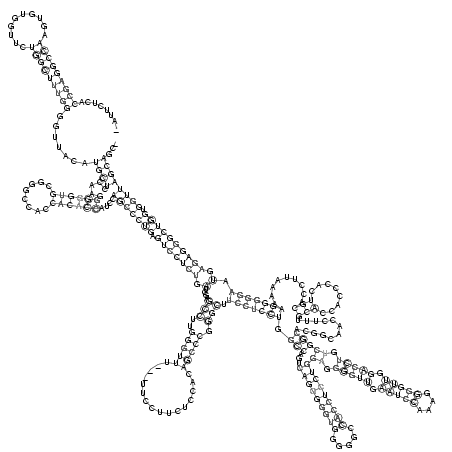
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004