Index >
Results for CNB 502451
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
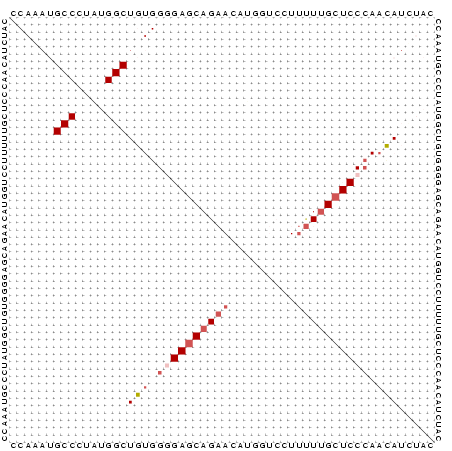
502451_ENSG00000166535_HUMAN_9684_9739/1-58 -CAGATGCCCAGTGGCTGTGGCGAGCAGAACATGGTCTTGTTTGCTCCCATCATCTA-
502451_ENSMUSG00000052587_MOUSE_1864_1921/1-58 CCAAATGCCCTATGGCTGTGGGGAGCAGAACATGGTCCTTTTTGCTCCCAACATCTAC
502451_ENSRNOG00000014814_RAT_8495_8552/1-58 CCAAATGCCCTATGGCTGTGGGGAGCAGAACATGGTCCTTTTTGCTCCAAACATCTAT
502451_ENSDARG00000008835_ZEBRAFISH_2351_2408/1-58 ACAGATGCCTTATGGCTGTGGAGAACAGAACATGATCATTCTCGCTCCCAATATTTAC
** ***** ********* ** ********* ** * * ***** * ** **
//
502451.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 58
Mean pairwise identity: 82.76
Mean single sequence MFE: -17.53
Consensus MFE: -10.01
Energy contribution: -11.83
Covariance contribution: 1.81
Mean z-score: -2.31
Structure conservation index: 0.57
SVM decision value: 0.37
SVM RNA-class probability: 0.710592
Prediction: RNA
######################################################################
>502451_ENSG00000166535_HUMAN_9684_9739/1-58
CAGAUGCCCAGUGGCUGUGGCGAGCAGAACAUGGUCUUGUUUGCUCCCAUCAUCUA
.(((((((....))).((((.(((((((.((......))))))))))))).)))). ( -19.30)
>502451_ENSMUSG00000052587_MOUSE_1864_1921/1-58
CCAAAUGCCCUAUGGCUGUGGGGAGCAGAACAUGGUCCUUUUUGCUCCCAACAUCUAC
......(((....)))(((.((((((((((.........)))))))))).)))..... ( -19.70)
>502451_ENSRNOG00000014814_RAT_8495_8552/1-58
CCAAAUGCCCUAUGGCUGUGGGGAGCAGAACAUGGUCCUUUUUGCUCCAAACAUCUAU
(((...(((....)))..)))(((((((((.........))))))))).......... ( -19.00)
>502451_ENSDARG00000008835_ZEBRAFISH_2351_2408/1-58
ACAGAUGCCUUAUGGCUGUGGAGAACAGAACAUGAUCAUUCUCGCUCCCAAUAUUUAC
......(((....)))((.((((...((((........))))..))))))........ ( -12.10)
>consensus
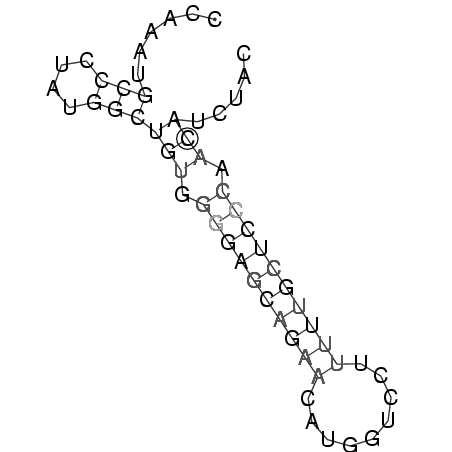
CCAAAUGCCCUAUGGCUGUGGGGAGCAGAACAUGGUCCUUUUUGCUCCCAACAUCUAC
......(((....)))(((.((((((((((.........)))))))))).)))..... (-10.01 = -11.83 + 1.81)
502451.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004