Index >
Results for CNB 502368-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
502368_ENSG00000159592_HUMAN_9630_9688/1-59 TTTAGGTCCAGTGTCTCCTTTCAGTAAGGTGAGGCATCCAACCTCATGGCCAGGATCTG
502368_ENSRNOG00000016336_RAT_9791_9846/1-59 TTTAGGTCCAGTGTCT--TTTCAGCAAGTTGAGGCATCCAACCTCATGGCCAGGATCT-
502368_ENSMUSG00000034042_MOUSE_9884_9939/1-59 TTTAGGTCCAGTGTCT--TTTTAGCAAGATGAGGCATCCAACCTCATGGCCAGGATCT-
502368_SINFRUG00000128130_FUGU_3910_3968/1-59 TTTAGGTCCAGTGTTTCCGGTCAGTGGATCGAGGCATCAAACCTCATGGCCAGGATCTG
************** * * ** ******** *******************
502368-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 59
Mean pairwise identity: 86.32
Mean single sequence MFE: -16.62
Consensus MFE: -12.91
Energy contribution: -13.22
Covariance contribution: 0.31
Mean z-score: -1.49
Structure conservation index: 0.78
SVM decision value: 0.12
SVM RNA-class probability: 0.590887
Prediction: RNA
######################################################################
>502368_ENSG00000159592_HUMAN_9630_9688/1-59
UUUAGGUCCAGUGUCUCCUUUCAGUAAGGUGAGGCAUCCAACCUCAUGGCCAGGAUCUG
..(((((((.(((((((((((....)))).)))))))(((......)))...))))))) ( -18.90)
>502368_ENSRNOG00000016336_RAT_9791_9846/1-59
UUUAGGUCCAGUGUCUUUUCAGCAAGUUGAGGCAUCCAACCUCAUGGCCAGGAUCU
...((((((.(((((((..(.....)..)))))))(((......)))...)))))) ( -14.90)
>502368_ENSMUSG00000034042_MOUSE_9884_9939/1-59
UUUAGGUCCAGUGUCUUUUUAGCAAGAUGAGGCAUCCAACCUCAUGGCCAGGAUCU
...((((((...(((((......)))))((((.......)))).......)))))) ( -14.30)
>502368_SINFRUG00000128130_FUGU_3910_3968/1-59
UUUAGGUCCAGUGUUUCCGGUCAGUGGAUCGAGGCAUCAAACCUCAUGGCCAGGAUCUG
..(((((((.(((((((.((((....)))))))))))....((....))...))))))) ( -18.40)
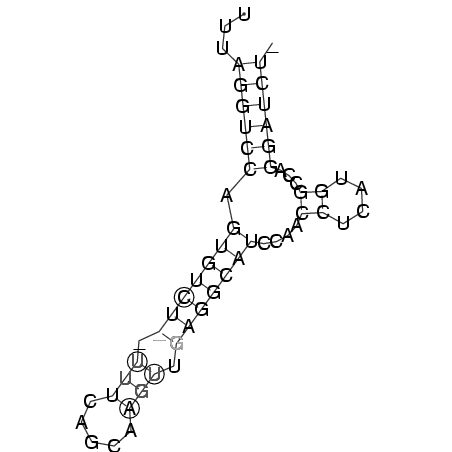
>consensus
UUUAGGUCCAGUGUCU__UUUCAGCAAGUUGAGGCAUCCAACCUCAUGGCCAGGAUCU_
...((((((.(((((((.(((.....))).)))))))....((....))...)))))). (-12.91 = -13.22 + 0.31)
502368-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004