Index >
Results for CNB 501427_9-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
501427_ENSMUSG00000039825_MOUSE_10_714/1-814 ----TGGTG--GAG--GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCTGGAC
501427_ENSRNOG00000011386_RAT_10_729/1-814 ----TGGTG--GAG--GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCTGGAC
501427_SINFRUG00000136522_FUGU_3814_4614/1-814 GCATTAGCTCCTATCGAGACCACCGAGCTCCCTGCACTGATGCTGCTCTGACTGCGAGGC
501427_ENSG00000135540_HUMAN_9280_9998/1-814 ----TGGCG--GAA--GTCATGC--TGCTGCCAGCACTAACTGTGCTCTGGCTCCGGGAG
501427_ENSDARG00000025235_ZEBRAFISH_250_1001/1-814 ----TAGCT--GAT--ACACCAC--TACTACTTGCACTGATGCTGCTCTGGCTCCTTGGA
*.* * . .. .* :.** * *.**.* *******.** * *.
501427_ENSMUSG00000039825_MOUSE_10_714/1-814 CTGGGGAGCCAGGGTTCCTCAAAGTCACTGCAGGGGCTCTGGGAAGGGGAGCTGCCG---
501427_ENSRNOG00000011386_RAT_10_729/1-814 CTGGGGAGCCAGGGTTCCTCACAGTCACTGCAGGGGCTCTGGGAAGGGGAACCGCTG---
501427_SINFRUG00000136522_FUGU_3814_4614/1-814 CTTAAGAGCCAGGGGTCATCATAGTCGCTGCTGGGGCTGTGAGACGGCGAGGAAGGCGAT
501427_ENSG00000135540_HUMAN_9280_9998/1-814 CGGGGCAGCCAGGGCTCTTCCAAGTCACTGCAGGGGCTCTGGGAGGGCGAGCTGCCA---
501427_ENSDARG00000025235_ZEBRAFISH_250_1001/1-814 CGAAGCATCCAGGGGTCCTCATAGTCACTACAAGGGCTATGAGATGGTGACGTGGAGGAT
* .. * ****** ** **. ****.**.*:.***** **.** ** ** .
501427_ENSMUSG00000039825_MOUSE_10_714/1-814 ---CCCTTGCCAGG---------GAGGCTTAGCTGCAGTGAATGCTGGAGCGAGGCGATC
501427_ENSRNOG00000011386_RAT_10_729/1-814 ---CCTTGGCCAGG---------GAGGCTTAGCTGCAGAGAATGCTGGAGTGAGGCGATC
501427_SINFRUG00000136522_FUGU_3814_4614/1-814 GCCCCTTTTCCTTTCCCTCCCCCCAGACCCATCTGTAGACTCTGCTGCAGGCTGGCAATG
501427_ENSG00000135540_HUMAN_9280_9998/1-814 ---CTCTTGCCTGG---------GAGGCTCAGCTGCAGCGAGTGCTGGAGTGTGGCGATC
501427_ENSDARG00000025235_ZEBRAFISH_250_1001/1-814 ---CCTTTGCCTTT---------AAGCCCATTCTGAAGTGACTGTTGGAGAGTGGCAATC
* * **: ** * : *** ** : ** ** ** :***.**
501427_ENSMUSG00000039825_MOUSE_10_714/1-814 AGGCTCTCATT-----GAGCAC--------------------CTGACC-GT--TT-GT-C
501427_ENSRNOG00000011386_RAT_10_729/1-814 AGGCTCTCGTT-----GAGCAC--------------------CTGCCC-AT--TT-GT-C
501427_SINFRUG00000136522_FUGU_3814_4614/1-814 AGCGTCTCACTGAGCATGGCTCCATTTGCATCGTTAGCTCCATTCACC-GTGCTGACACC
501427_ENSG00000135540_HUMAN_9280_9998/1-814 AGGCTCTCGTT-----GAGCAC--------------------CTGCCC-GT--TG-CA-C
501427_ENSDARG00000025235_ZEBRAFISH_250_1001/1-814 AATGATTCATTGAGAAGAGAACCATTCCTAATATTTG----ACTGCCCAAT-ATT-GG-C
*. : **. * .*.:* * .** .* * *
501427_ENSMUSG00000039825_MOUSE_10_714/1-814 TG-------GTTGTTCTTTTTGGGAATCCTTCGCAGAGAATCGGTCCGGGATGGTGGCAG
501427_ENSRNOG00000011386_RAT_10_729/1-814 TG-------GTTGTTCTTCTTGGGAATCCTTCGCAGAGAATCTGTCCGGGATGGTGGCGG
501427_SINFRUG00000136522_FUGU_3814_4614/1-814 TCCGACGGGCTTTTTCGGGCCTGGCTTGCGTCTGAGAGAGTCTGTTCGAGCCGGAGGCAG
501427_ENSG00000135540_HUMAN_9280_9998/1-814 TG-------GCTGCTCTTCTTGGGAATCCTGCGGAGGGAGTCTGTCCGGGAGGGTGGCAG
501427_ENSDARG00000025235_ZEBRAFISH_250_1001/1-814 AGAGGGAGAGCTGTTTTTCCCAGGCTTTCTCCGTAGAGAGTCTGTGCGTGCAGGTGGAAG
: * * **.:* * * **.**.** ** ** *. **:**..*
501427_9-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 61.53
Mean single sequence MFE: -109.80
Consensus MFE: -41.08
Energy contribution: -42.08
Covariance contribution: 1.00
Mean z-score: -1.95
Structure conservation index: 0.37
SVM decision value: 0.24
SVM RNA-class probability: 0.652539
Prediction: RNA
######################################################################
>501427_ENSMUSG00000039825_MOUSE_10_714/1-814
UGGUGGAGGUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGGGAGCUGCCGCCCUUGCCAGGGAGGCUUAGCUGCAGUGAAUGCUGGAGCGAGGCGAUCAGGCUCUCAUUGAGCACCUGACCGUUUGUCUGGUUGUUCUUUUUGGGAAUCCUUCGCAGAGAAUCGGUCCGGGAUGGUGGCAG
.(((((.((.....)).)))))(((((.......((((((((((((.......))))))))))))(((......(((((((((.((((((.......))))))(((.((((....)))).)))....))))))))).....))))))))((.((((.((((.....)))).((.(((((.((.(((((..(((((.....)))))....).)))).)).))))).))..)))).)).. ( -105.10)
>501427_ENSRNOG00000011386_RAT_10_729/1-814
UGGUGGAGGUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCACAGUCACUGCAGGGGCUCUGGGAAGGGGAACCGCUGCCUUGGCCAGGGAGGCUUAGCUGCAGAGAAUGCUGGAGUGAGGCGAUCAGGCUCUCGUUGAGCACCUGCCCAUUUGUCUGGUUGUUCUUCUUGGGAAUCCUUCGCAGAGAAUCUGUCCGGGAUGGUGGCGG
.(((((.((.....)).)))))..(((((.((..((((((((((((.......))))))))))))((((.((((((.((((..(((.((..(((((((.(((((......(((.((((((.((((.(((.(((......))).))).))))..))..((((.....)))).)))).)))......))))).)))))))..))...)))..)))).))..))))..)))))).))))). ( -99.30)
>501427_SINFRUG00000136522_FUGU_3814_4614/1-814
GCAUUAGCUCCUAUCGAGACCACCGAGCUCCCUGCACUGAUGCUGCUCUGACUGCGAGGCCUUAAGAGCCAGGGGUCAUCAUAGUCGCUGCUGGGGCUGUGAGACGGCGAGGAAGGCGAUGCCCCUUUUCCUUUCCCUCCCCCCAGACCCAUCUGUAGACUCUGCUGCAGGCUGGCAAUGAGCGUCUCACUGAGCAUGGCUCCAUUUGCAUCGUUAGCUCCAUUCACCGUGCUGACACCUCCGACGGGCUUUUUCGGGCCUGGCUUGCGUCUGAGAGAGUCUGUUCGAGCCGGAGGCAG
((((((((((.....)))........((.....)).)))))))(((((.....(.(((((((((...((((((((((.((((((((.(....).))))))))((.((.(((((((((...)))....)))))).)).))......))))).(((((((......))))))))))))..)))).))))).).)))))((.((((.........((((((.(........).))))))..(((.((((((((((((((((((......).))))))))))))))))).)))..)))).)). ( -121.90)
>501427_ENSG00000135540_HUMAN_9280_9998/1-814
UGGCGGAAGUCAUGCUGCUGCCAGCACUAACUGUGCUCUGGCUCCGGGAGCGGGGCAGCCAGGGCUCUUCCAAGUCACUGCAGGGGCUCUGGGAGGGCGAGCUGCCACUCUUGCCUGGGAGGCUCAGCUGCAGCGAGUGCUGGAGUGUGGCGAUCAGGCUCUCGUUGAGCACCUGCCCGUUGCACUGGCUGCUCUUCUUGGGAAUCCUGCGGAGGGAGUCUGUCCGGGAGGGUGGCAG
((((....)))).((((((((((((((.....))))..)))).(((....)))))))))....(((((((((((((........)))).)))))))))...((((((((((..((((((((((((..((((((.(((((((..((((..(((..(((((((.....))))..)))..)))..))))))).))))(((....)))..))))))...)))))).)))))))))))))))) ( -120.90)
>501427_ENSDARG00000025235_ZEBRAFISH_250_1001/1-814
UAGCUGAUACACCACUACUACUUGCACUGAUGCUGCUCUGGCUCCUUGGACGAAGCAUCCAGGGGUCCUCAUAGUCACUACAAGGGCUAUGAGAUGGUGACGUGGAGGAUCCUUUGCCUUUAAGCCCAUUCUGAAGUGACUGUUGGAGAGUGGCAAUCAAUGAUUCAUUGAGAAGAGAACCAUUCCUAAUAUUUGACUGCCCAAUAUUGGCAGAGGGAGAGCUGUUUUUCCCAGGCUUUCUCCGUAGAGAGUCUGUGCGUGCAGGUGGAAG
.................((((((((((((((((..(((((((.(((((((.......))))))))))((.(((((((((.((.(((((..(((..(....)(..(((....)))..))))..)))))....)).))))))))).))))))..)).))))..(((((.(((.(.(((((.((...............(((((.......))))).(((((((....))))))).)).))))).).))).))))).....))))))))))... ( -101.80)
>consensus
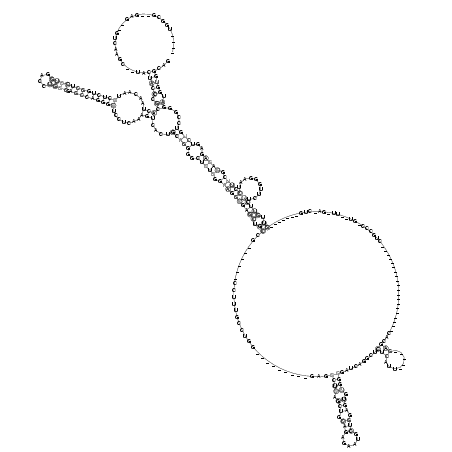
____UGGCG__GAG__GUCAAGC__UACUACCCGCACUAACAAUGCUCUGGCUCCUGGACCUGGGGAGCCAGGGGUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGCGAGCUGCCG______CCUUUGCCUGG_________GAGGCUCAGCUGCAGAGAAUGCUGGAGUGUGGCGAUCAGGCUCUCAUU_____GAGCAC____________________CUGCCC_GU__UU_GA_CUG_______GUUGUUCUUCUUGGGAAUCCUUCGCAGAGAGUCUGUCCGGGAUGGUGGCAG
...........................(((((.(((((......(((((((((((((....)))).))))))))).......)))....(((((..(((((.((((((((((.((...............................((((.(((.(((......))).))).)))).........(((........)))..................................................)).)))))..........))))).)))))..)))))....)).)))))... (-41.08 = -42.08 + 1.00)
501427_9-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004