Index >
Results for CNB 501416_8-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 -CTTGTGTCGCTCTGGGACGGAGTGACCCCACACAGAGAATACACGT-TGGGAGT--GG-
501416_ENSRNOG00000011386_RAT_133_729/1-680 GCTTGTGTCGCTCTGAGATGGAGTGAGCCCGCACAGAGAATACACGT-TGGGAGT--GG-
501416_ENSG00000135540_HUMAN_9403_9999/1-680 GCTTGTGTCACTCTGCGATGGCGTGGCCCCGCACAGGGAGTAGACAT-TGGGGGT--GG-
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 GCTGGTCTCACTGTGAGTGGGGGTCACGTGGCACAGAGAGTAAACATTAGAGACACCAGC
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 ACTTGTGTCACTATGAGATGGTGTGACGGGACAGATGGAGTATACATGAGCCATTCCTG-
** ** **.** ** *: ** ** . .** * .**.** **.* :* . : *
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 --------TGGTG--GAG--GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCT
501416_ENSRNOG00000011386_RAT_133_729/1-680 --------TGGTG--GAG--GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCT
501416_ENSG00000135540_HUMAN_9403_9999/1-680 --------TGGCG--GAA--GTCATGC--TGCTGCCAGCACTAACTGTGCTCTGGCTCCG
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 GCAAGCATTAGCTCCTATCGAGACCACCGAGCTCCCTGCACTGATGCTGCTCTGACTGCG
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 --------TAGCT--GAT--ACACCAC--TACTACTTGCACTGATGCTGCTCTGGCTCCT
*.* * . .. .* :.** * *.**.* *******.** *
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 GGACCTGGGGAGCCAGGGTTCCTCAAAGTCACTGCAGGGGCTCTGGGAAGGGGAGCTGCC
501416_ENSRNOG00000011386_RAT_133_729/1-680 GGACCTGGGGAGCCAGGGTTCCTCACAGTCACTGCAGGGGCTCTGGGAAGGGGAACCGCT
501416_ENSG00000135540_HUMAN_9403_9999/1-680 GGAGCGGGGCAGCCAGGGCTCTTCCAAGTCACTGCAGGGGCTCTGGGAGGGCGAGCTGCC
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 AGGCCTTAAGAGCCAGGGGTCATCATAGTCGCTGCTGGGGCTGTGAGACGGCGAGGAAGG
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 TGGACGAAGCATCCAGGGGTCCTCATAGTCACTACAAGGGCTATGAGATGGTGACGTGGA
*. * .. * ****** ** **. ****.**.*:.***** **.** ** ** .
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 G------CCCTTGCCAGG---------GAGGCTTAGCTGCAGTGAATGCTGGAGCGAGGC
501416_ENSRNOG00000011386_RAT_133_729/1-680 G------CCTTGGCCAGG---------GAGGCTTAGCTGCAGAGAATGCTGGAGTGAGGC
501416_ENSG00000135540_HUMAN_9403_9999/1-680 A------CTCTTGCCTGG---------GAGGCTCAGCTGCAGCGAGTGCTGGAGTGTGGC
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 CGATGCCCCTTTTCCTTTCCCTCCCCCCAGACCCATCTGTAGACTCTGCTGCAGGCTGGC
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 GGAT---CCTTTGCCTTT---------AAGCCCATTCTGAAGTGACTGTTGGAGAGTGGC
* * **: ** * : *** ** : ** ** ** :***
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 GATCAGGCTCTCATT-----GAGCAC--------------
501416_ENSRNOG00000011386_RAT_133_729/1-680 GATCAGGCTCTCGTT-----GAGCAC--------------
501416_ENSG00000135540_HUMAN_9403_9999/1-680 GATCAGGCTCTCGTT-----GAGCAC--------------
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 AATGAGCGTCTCACTGAGCATGGCTCCATTTGCATCGTTA
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 AATCAATGATTCATTGAGAAGAGAACCATTCCTAATATTT
.** *. : **. * .*.:*
501416_8-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 280
Mean pairwise identity: 62.78
Mean single sequence MFE: -101.28
Consensus MFE: -39.96
Energy contribution: -39.92
Covariance contribution: -0.04
Mean z-score: -1.78
Structure conservation index: 0.39
SVM decision value: 0.02
SVM RNA-class probability: 0.542661
Prediction: RNA
######################################################################
>501416_ENSMUSG00000039825_MOUSE_133_713/1-680
CUUGUGUCGCUCUGGGACGGAGUGACCCCACACAGAGAAUACACGUUGGGAGUGGUGGUGGAGGUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGGGAGCUGCCGCCCUUGCCAGGGAGGCUUAGCUGCAGUGAAUGCUGGAGCGAGGCGAUCAGGCUCUCAUUGAGCAC
.((((.(((((((((..(((((((....))).(((((.......((((((((.((((((((........))))))))))).)))))....)))))...))))..))((((((((....))))))))...(((((((((.((((((.......))))))(((.((((....)))).)))....))))))))).....))))))).))))....((((.....)))).. ( -106.20)
>501416_ENSRNOG00000011386_RAT_133_729/1-680
GCUUGUGUCGCUCUGAGAUGGAGUGAGCCCGCACAGAGAAUACACGUUGGGAGUGGUGGUGGAGGUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCACAGUCACUGCAGGGGCUCUGGGAAGGGGAACCGCUGCCUUGGCCAGGGAGGCUUAGCUGCAGAGAAUGCUGGAGUGAGGCGAUCAGGCUCUCGUUGAGCAC
(.((((.(((((((.((.((.((((((((.((.(((((.......((((((((.((((((((........))))))))))).)))))....)))))))((((((.((.(((.(((((((((((((..(((...))).))))))))))....((....))))).))).)))))))).)))))).)).)).......))))))))).)))).)..((((.....)))).. ( -99.50)
>501416_ENSG00000135540_HUMAN_9403_9999/1-680
GCUUGUGUCACUCUGCGAUGGCGUGGCCCCGCACAGGGAGUAGACAUUGGGGGUGGUGGCGGAAGUCAUGCUGCUGCCAGCACUAACUGUGCUCUGGCUCCGGGAGCGGGGCAGCCAGGGCUCUUCCAAGUCACUGCAGGGGCUCUGGGAGGGCGAGCUGCCACUCUUGCCUGGGAGGCUCAGCUGCAGCGAGUGCUGGAGUGUGGCGAUCAGGCUCUCGUUGAGCAC
..........(((((((.((((...(((((((.(..(((((((((((.(..((((.((((((.((.....)).)))))).))))..).))).))).)))))..).))))))).)))).((((......))))..)))))))((((.(.((((((((..((((((....(((.....)))...(((.((((....)))).))))))))).))..)))))).).)))).. ( -112.10)
>501416_SINFRUG00000136522_FUGU_3948_4614/1-680
GCUGGUCUCACUGUGAGUGGGGGUCACGUGGCACAGAGAGUAAACAUUAGAGACACCAGCGCAAGCAUUAGCUCCUAUCGAGACCACCGAGCUCCCUGCACUGAUGCUGCUCUGACUGCGAGGCCUUAAGAGCCAGGGGUCAUCAUAGUCGCUGCUGGGGCUGUGAGACGGCGAGGAAGGCGAUGCCCCUUUUCCUUUCCCUCCCCCCAGACCCAUCUGUAGACUCUGCUGCAGGCUGGCAAUGAGCGUCUCACUGAGCAUGGCUCCAUUUGCAUCGUUA
(((((((((.(((((.(((.....)))....)))))...(....)....))))..)))))(((.((((((((((.....)))........((.....)).))))))))))...((.((((((((((((...((((((((((.((((((((.(....).))))))))((.((.(((((((((...)))....)))))).)).))......))))).(((((((......))))))))))))..)))).)))))...((((...)))).....))))).... ( -103.00)
>501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680
ACUUGUGUCACUAUGAGAUGGUGUGACGGGACAGAUGGAGUAUACAUGAGCCAUUCCUGUAGCUGAUACACCACUACUACUUGCACUGAUGCUGCUCUGGCUCCUUGGACGAAGCAUCCAGGGGUCCUCAUAGUCACUACAAGGGCUAUGAGAUGGUGACGUGGAGGAUCCUUUGCCUUUAAGCCCAUUCUGAAGUGACUGUUGGAGAGUGGCAAUCAAUGAUUCAUUGAGAAGAGAACCAUUCCUAAUAUUU
.(((.(.(((..(((((.(((((((.(((.((((((((..((....))..))))..))))..))).)))))))..........((.((((((..(((((((.(((((((.......))))))))))((.(((((((((.((.(((((..(((..(....)(..(((....)))..))))..)))))....)).))))))))).))))))..)).)))).)).)))))))).).)))................. ( -85.60)
>consensus
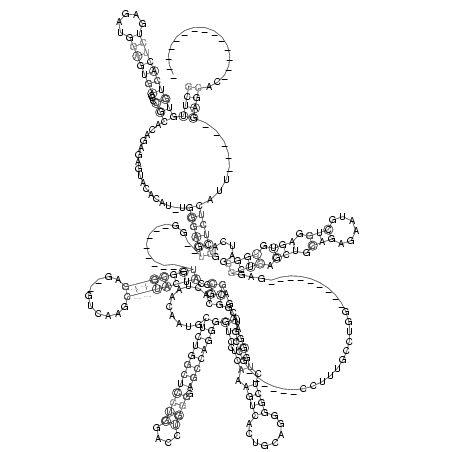
GCUUGUGUCACUCUGAGAUGGAGUGACCCCGCACAGAGAGUACACAU_UGGGAGU__GG_________UGGCG__GAG__GUCAAGC__UACUACCCGCACUAACAAUGCUCUGGCUCCUGGACCUGGGGAGCCAGGGGUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGCGAGCUGCCG______CCUUUGCCUGG_________GAGGCUCAGCUGCAGAGAAUGCUGGAGUGUGGCGAUCAGGCUCUCAUU_____GAGCAC______________
((((((((((((((.....)))))))...))).................((((((.............((((((((..........))))))))..(((..........((((((((((((....)))).))))))))(((.(((.((((........)))).)))))).))).....................................((((.(((.(((......))).))).))))......))))))........))))................ (-39.96 = -39.92 + -0.04)
501416_8-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004