Index >
Results for CNB 501415_5-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
501415_ENSDARG00000026402_ZEBRAFISH_374_1002/1-682 AGCATCCAGGGGTCCTCATAGTCACTACAAGGGCTATGAGATGGTGAC-----GTGGAGG
501415_SINFRUG00000136522_FUGU_3947_4616/1-682 AAGAGCCAGGGGTCATCATAGTCGCTGCTGGGGCTGTGAGACGGCGA-GGAAGGCGATGC
501415_ENSMUSG00000039825_MOUSE_129_716/1-682 GGGAGCCAGGGTTCCTCAAAGTCACTGCAGGGGCTCTGGGAAGGGGA------GC--TGC
501415_ENSG00000135540_HUMAN_9399_10000/1-682 GGCAGCCAGGGCTCTTCCAAGTCACTGCAGGGGCTCTGGGAGGGCGA------GC--TGC
501415_ENSRNOG00000011386_RAT_130_730/1-682 GGGAGCCAGGGTTCCTCACAGTCACTGCAGGGGCTCTGGGAAGGGGA------AC--CGC
* ****** ** ** **** ** * ***** ** ** ** ** *
501415_ENSDARG00000026402_ZEBRAFISH_374_1002/1-682 A-----TCCTTTGCCT---TTAAGCCCATTCTGAAGTGACTGTTGGAGAGTGGCAATCAA
501415_SINFRUG00000136522_FUGU_3947_4616/1-682 CCCTTTTCCTTTCCCTCCCCCCAGACCCATCTGTAGACTCTGCTGCAGGCTGGCAATGAG
501415_ENSMUSG00000039825_MOUSE_129_716/1-682 C-----GCCCTTGCCA---GGGAGGCTTAGCTGCAGTGAATGCTGGAGCGAGGCGATCAG
501415_ENSG00000135540_HUMAN_9399_10000/1-682 C-----ACTCTTGCCT---GGGAGGCTCAGCTGCAGCGAGTGCTGGAGTGTGGCGATCAG
501415_ENSRNOG00000011386_RAT_130_730/1-682 T-----GCCTTGGCCA---GGGAGGCTTAGCTGCAGAGAATGCTGGAGTGAGGCGATCAG
* * ** ** * *** ** ** ** ** *** ** *
501415_ENSDARG00000026402_ZEBRAFISH_374_1002/1-682 TGATTCATTGAGAAGAGAACCATTCCTAATATT-TGAC--TGCCCAATATTGGCAGAG-G
501415_SINFRUG00000136522_FUGU_3947_4616/1-682 CGTCTCACTGAGCATGGCTCCATTTGCATCGTTAGCTCCATTCACCGTGCTGACACCTCC
501415_ENSMUSG00000039825_MOUSE_129_716/1-682 GCTCTCATTGAGCACCTGACCGTTTGTCTGGTT-GTTC--TT---TTTGGGAATCCTTCG
501415_ENSG00000135540_HUMAN_9399_10000/1-682 GCTCTCGTTGAGCACCTGCCCGTTGCACTGGCT-GCTC--TT---CTTGGGAATCCTGCG
501415_ENSRNOG00000011386_RAT_130_730/1-682 GCTCTCGTTGAGCACCTGCCCATTTGTCTGGTT-GTTC--TT---CTTGGGAATCCTTCG
** **** * ** ** * * * *
501415_ENSDARG00000026402_ZEBRAFISH_374_1002/1-682 GAGAGCTGTTTTTC--CCAGGCTTTCTCCGTAGAGAGTCTGTGCGTGCAGGTGGAAGGGG
501415_SINFRUG00000136522_FUGU_3947_4616/1-682 GACGGGCTTTTTCGGGCCTGGCTTGCGTCTGAGAGAGTCTGTTCGAGCCGGAGGCAGCGG
501415_ENSMUSG00000039825_MOUSE_129_716/1-682 CAGAGAATCGGTCCGGGATGG-TGGCAGGGGTG-GTTTCTTTGCTTTCTTCAG--AGAGA
501415_ENSG00000135540_HUMAN_9399_10000/1-682 GAGGGAGTCTGTCCGGGAGGG-TGGCAGGGGAG-GCTTCTTTGCTTTCTTCAA--AGAGA
501415_ENSRNOG00000011386_RAT_130_730/1-682 CAGAGAATCTGTCCGGGATGG-TGGCGGTGGTG-GTTTTTTTGCTTTCTTCAG--AGAGA
* * * ** * * * * * * * * * ** *
501415_ENSDARG00000026402_ZEBRAFISH_374_1002/1-682 TGGTTTCTTAGGCTTGCGTAGAGAAATGGAACGAGATAGCGAGTGGTCGCTGCACAAGCT
501415_SINFRUG00000136522_FUGU_3947_4616/1-682 TGGCTTCTTAGACTTGCGGAGGGAGATGCTGCGTGACAGCGACTGGTCGCTGTACAGGCT
501415_ENSMUSG00000039825_MOUSE_129_716/1-682 T-GTTTCTC-GA-AAG-GAT--TTTATCCTG-GTGAGCG-GCCTGATCCCCTT---GGCT
501415_ENSG00000135540_HUMAN_9399_10000/1-682 T-GTTTCTT-GA-TAG-GGA--TTTATCCTG-GTAATTG-GACCGGTCCCCTT---GGTT
501415_ENSRNOG00000011386_RAT_130_730/1-682 T-GTTTCTT-GA-GAG-GAT--TCTATCCTG-CTGAGTG-GCCTGATCCCCTT---GGCT
* * **** * * * ** * * * * ** * * *
501415_5-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 62.65
Mean single sequence MFE: -115.18
Consensus MFE: -37.56
Energy contribution: -34.80
Covariance contribution: -2.76
Mean z-score: -2.12
Structure conservation index: 0.33
SVM decision value: 0.11
SVM RNA-class probability: 0.586649
Prediction: RNA
######################################################################
>501415_ENSDARG00000026402_ZEBRAFISH_374_1002/1-682
AGCAUCCAGGGGUCCUCAUAGUCACUACAAGGGCUAUGAGAUGGUGACGUGGAGGAUCCUUUGCCUUUAAGCCCAUUCUGAAGUGACUGUUGGAGAGUGGCAAUCAAUGAUUCAUUGAGAAGAGAACCAUUCCUAAUAUUUGACUGCCCAAUAUUGGCAGAGGGAGAGCUGUUUUUCCCAGGCUUUCUCCGUAGAGAGUCUGUGCGUGCAGGUGGAAGGGGUGGUUUCUUAGGCUUGCGUAGAGAAAUGGAACGAGAUAGCGAGUGGUCGCUGCACAAGCU
(((.((((.((((((((...((((((((...........).)))))))...))))))))(((((...((((((((((((..((......))..))))))....((((((...)))))).((((((.((((((((..((((((.(((((.......))))).(((((((....)))))))(((((((((....))))))))).......))))))..)))))))))))))).)))))).)))))....))))....(.((((((....)))))))....))) ( -99.40)
>501415_SINFRUG00000136522_FUGU_3947_4616/1-682
AAGAGCCAGGGGUCAUCAUAGUCGCUGCUGGGGCUGUGAGACGGCGAGGAAGGCGAUGCCCCUUUUCCUUUCCCUCCCCCCAGACCCAUCUGUAGACUCUGCUGCAGGCUGGCAAUGAGCGUCUCACUGAGCAUGGCUCCAUUUGCAUCGUUAGCUCCAUUCACCGUGCUGACACCUCCGACGGGCUUUUUCGGGCCUGGCUUGCGUCUGAGAGAGUCUGUUCGAGCCGGAGGCAGCGGUGGCUUCUUAGACUUGCGGAGGGAGAUGCUGCGUGACAGCGACUGGUCGCUGUACAGGCU
....((((((((((.((((((((.(....).))))))))((.((.(((((((((...)))....)))))).)).))......))))).(((((((......))))))))))))....((((((((...(((((((....))..))).)).....((((...(((((((((..(..(((.((((((((((((((((((......).))))))))))))))))).)))..)..)))).)))))((...........)))))).))))))))((.(((((((((....))))))).)).)). ( -134.00)
>501415_ENSMUSG00000039825_MOUSE_129_716/1-682
GGGAGCCAGGGUUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGGGAGCUGCCGCCCUUGCCAGGGAGGCUUAGCUGCAGUGAAUGCUGGAGCGAGGCGAUCAGGCUCUCAUUGAGCACCUGACCGUUUGUCUGGUUGUUCUUUUUGGGAAUCCUUCGCAGAGAAUCGGUCCGGGAUGGUGGCAGGGGUGGUUUCUUUGCUUUCUUCAGAGAGAUGUUUCUCGAAAGGAUUUUAUCCUGGUGAGCGGCCUGAUCCCCUUGGCU
...((((((((((((......(((((((((.((((((.......))))))(((.((((....)))).)))....))))))))).....)))))..((.((((((((((((((.........(((((.((.(((((..(((((.....)))))....).)))).)).)))))((((((((.((((((((.....))))))))(((((..(((((....)))))..)))))..))))))))))))).)))))))))))))))))) ( -117.20)
>501415_ENSG00000135540_HUMAN_9399_10000/1-682
GGCAGCCAGGGCUCUUCCAAGUCACUGCAGGGGCUCUGGGAGGGCGAGCUGCCACUCUUGCCUGGGAGGCUCAGCUGCAGCGAGUGCUGGAGUGUGGCGAUCAGGCUCUCGUUGAGCACCUGCCCGUUGCACUGGCUGCUCUUCUUGGGAAUCCUGCGGAGGGAGUCUGUCCGGGAGGGUGGCAGGGGAGGCUUCUUUGCUUUCUUCAAAGAGAUGUUUCUUGAUAGGGAUUUAUCCUGGUAAUUGGACCGGUCCCCUUGGUU
...((((((((((((((((((((........)))).)))))))))((.((((((((((..((((((((((((..((((((.(((((((..((((..(((..(((((((.....))))..)))..)))..))))))).))))(((....)))..))))))...)))))).))))))))))))))))(((((((......)))))))))...((((....))))....((((((..(((........)))..))))))))))))) ( -119.70)
>501415_ENSRNOG00000011386_RAT_130_730/1-682
GGGAGCCAGGGUUCCUCACAGUCACUGCAGGGGCUCUGGGAAGGGGAACCGCUGCCUUGGCCAGGGAGGCUUAGCUGCAGAGAAUGCUGGAGUGAGGCGAUCAGGCUCUCGUUGAGCACCUGCCCAUUUGUCUGGUUGUUCUUCUUGGGAAUCCUUCGCAGAGAAUCUGUCCGGGAUGGUGGCGGUGGUGGUUUUUUUGCUUUCUUCAGAGAGAUGUUUCUUGAGAGGAUUCUAUCCUGCUGAGUGGCCUGAUCCCCUUGGCU
...((((((((....(((..((((((((((((.(((((((((((.(((((((((((....((((((((((((.(((.(((......))).))).))))((((((((......((.((....)).))...))))))))...))))))))..(((((..((((.....))))..)))))...))))))))).......)).))))).))))))(((..((((....))))..))).))))))..)))))).)))...)))))))) ( -105.61)
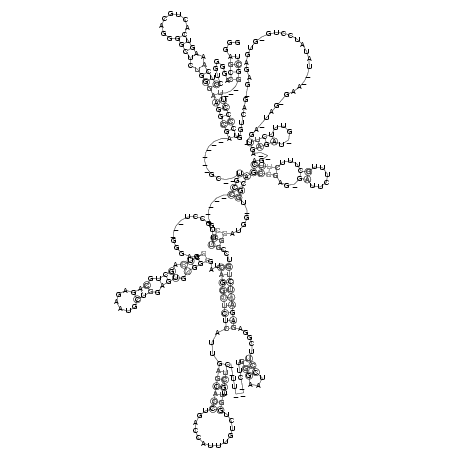
>consensus
GGGAGCCAGGGGUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGCGA______GC__UGCC_____GCCUUUGCCU___GGGAGGCUCAGCUGCAGAGAAUGCUGGAGUGUGGCGAUCAGGCUCUCAUUGAGCACCUGACCAUUUGUCUGGUU_GCUC__UU___CUUGGGAAUCCUUCGGAGAGAAUCUGUCCGGGAUGG_UGGCAGCGGAG_GAUUCUUUGCUUUCUUCAG__AGAGAU_GUUUCUU_GA_UAG_GAA__UAUAUCCUG_GUGAGAG_GACUGGUCCCCUU___GGCU
...((((.......((((.((((........)))).))))(((((((..........((((......(((..............((((.(((.(((......))).))).))))...(((((((((...(((((((.............)))..))))..........(((...))).....)))))))))...)))......))))((((...((......))...)))).....((((.....)))).....................................)))))))...)))) (-37.56 = -34.80 + -2.76)
501415_5-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004