Index >
Results for CNB 489516-rev
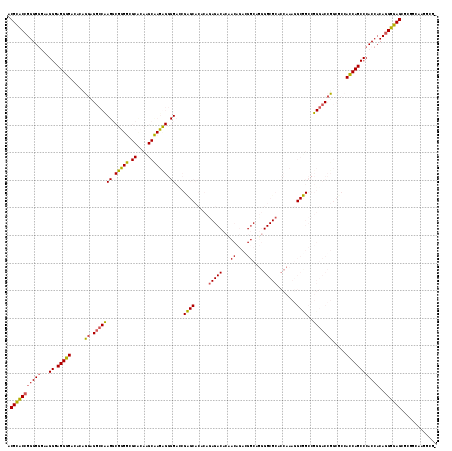
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
489516_ENSG00000089213_HUMAN_5694_5848/1-156 ACTTACCTGGTGTAAGGTATGGTACATAAGAGTCCAATGCTATTTGCGCAAGCGATAGGG
489516_ENSRNOG00000001354_RAT_4711_4865/1-156 ACTCACCTGGTGTAAGGTATGGTACACAGGAGTCCAATGCTGTTCGCACAAGCAATAGGG
489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156 ACTCACCTGGTATAAGGTATGGTACACAGGAGTCCAATGCTGTTCGCACAAGCAATGGGG
489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156 -CTTGCCTGGTGTAGGGGATGGTGCAGAGCAGGCCGATGCTGTTGGCACAGGCGATGGGG
** ****** ** ** ***** ** * ** ** ***** ** ** ** ** ** ***
489516_ENSG00000089213_HUMAN_5694_5848/1-156 TAACGAGCACACAGAGACACAACAGAGGTCATGGTCTCGCCAAAGGCTTCCTGGACCTGC
489516_ENSRNOG00000001354_RAT_4711_4865/1-156 TACCGAGCACACAGAGACACAACAGACGTCATGGTCTCCCCAAAGGCTTCCTGGACCTGC
489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156 TACCGAGCACACAAAGACACGACAGAGGTCATGGTCTCACCAAAGGCTTCCTGGACCTGC
489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156 TATCGGGCACACAGAGACACCACTGACGTCATGGTCTCTCCAAACGCCTCCTGCACCTGC
** ** ******* ****** ** ** *********** ***** ** ***** ******
489516_ENSG00000089213_HUMAN_5694_5848/1-156 TCGACCATCCCACCACATGTGAGTTCTTCAATTCT-
489516_ENSRNOG00000001354_RAT_4711_4865/1-156 TCCACCATCCCACCACAGGTGAGCTCTTCAATTCT-
489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156 TCCACCATCCCACCACAGGTGAGCTCTTCAATTCT-
489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156 TCCATCATCCCACCACAGGTGAGCTCCTCCACCCTT
** * ************ ***** ** ** * **
489516-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 156
Mean pairwise identity: 86.50
Mean single sequence MFE: -51.83
Consensus MFE: -39.69
Energy contribution: -39.12
Covariance contribution: -0.56
Mean z-score: -1.94
Structure conservation index: 0.77
SVM decision value: 0.74
SVM RNA-class probability: 0.838764
Prediction: RNA
######################################################################
>489516_ENSG00000089213_HUMAN_5694_5848/1-156
ACUUACCUGGUGUAAGGUAUGGUACAUAAGAGUCCAAUGCUAUUUGCGCAAGCGAUAGGGUAACGAGCACACAGAGACACAACAGAGGUCAUGGUCUCGCCAAAGGCUUCCUGGACCUGCUCGACCAUCCCACCACAUGUGAGUUCUUCAAUUCU
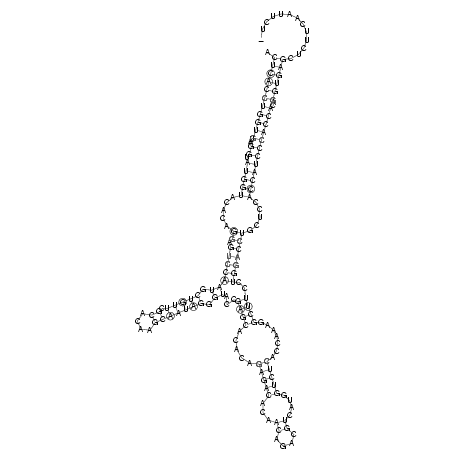
((((((.(((((...((.(((((.....((.(((((.((((..((((....))))...))))..((((.....(((((.((((....))..))))))).......))))..))))))).....))))))))))))...))))))........... ( -47.90)
>489516_ENSRNOG00000001354_RAT_4711_4865/1-156
ACUCACCUGGUGUAAGGUAUGGUACACAGGAGUCCAAUGCUGUUCGCACAAGCAAUAGGGUACCGAGCACACAGAGACACAACAGACGUCAUGGUCUCCCCAAAGGCUUCCUGGACCUGCUCCACCAUCCCACCACAGGUGAGCUCUUCAAUUCU
.(((((((((((...((.(((((.....(((((....((((.........))))...((((.((((((.....(((((.((((....))..))))))).......))))...))))))))))))))))))))))..)))))))............ ( -46.50)
>489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156
ACUCACCUGGUAUAAGGUAUGGUACACAGGAGUCCAAUGCUGUUCGCACAAGCAAUGGGGUACCGAGCACACAAAGACACGACAGAGGUCAUGGUCUCACCAAAGGCUUCCUGGACCUGCUCCACCAUCCCACCACAGGUGAGCUCUUCAAUUCU
.((((((((......((.(((((.....(((.((((.((((.........)))).)))).).))((((...........(....)(((((..(((((......))))).....))))))))).))))))).....))))))))............ ( -49.00)
>489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156
CUUGCCUGGUGUAGGGGAUGGUGCAGAGCAGGCCGAUGCUGUUGGCACAGGCGAUGGGGUAUCGGGCACACAGAGACACCACUGACGUCAUGGUCUCUCCAAACGCCUCCUGCACCUGCUCCAUCAUCCCACCACAGGUGAGCUCCUCCACCCUU
((..((((.((...(((((((((..(((((((.(((((((.((.((....)).).).)))))))(((.....((((.((((.((....)))))).)))).....))).......))))))))))))))))..))))))..))............. ( -63.90)
>consensus
ACUCACCUGGUGUAAGGUAUGGUACACAGGAGUCCAAUGCUGUUCGCACAAGCAAUAGGGUACCGAGCACACAGAGACACAACAGACGUCAUGGUCUCACCAAAGGCUUCCUGGACCUGCUCCACCAUCCCACCACAGGUGAGCUCUUCAAUUCU_
.(((((((((((...((.(((((.....((.(((((((.(((((.((....))))))).))...((((.....(((((...((....))....))))).......))))..))))))).....))))))))))))..))))))............. (-39.69 = -39.12 + -0.56)
489516-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004