Index >
Results for CNB 473673
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
473673_ENSG00000177669_HUMAN_4164_4404/1-245 -CTTCTCTT-TCAGGTACCTCTTTCTCCTGACTGGAGGAGGTGCCCTGGCCGTGGCTGCC
473673_ENSRNOG00000013246_RAT_7392_7632/1-245 -CCTTTCTT-TCAGGTACCTCTTTCTCCTGGCTGGAGGAGGTGTCCTGGCTTTGGCCGCC
473673_ENSMUSG00000042953_MOUSE_6061_6302/1-245 -CCTTTCTT-TCAGGTACCTCTTTCTCCTGGCTGGAGGAGGTGTCCTGGCTTTTGCTGCC
473673_ENSDARG00000022302_ZEBRAFISH_7796_8038/1-245 TCATCTCTTGTAAGGTATGTCTATTTGGCGATGGGAGGATTCATGCTGGCTATTGCAACA
* * **** * ***** *** * * * ****** ***** * ** *
473673_ENSG00000177669_HUMAN_4164_4404/1-245 ATGGGTTCCTACGCCGTGCTCGTCTTCACCCCTGCTGTCTGCGCTGTGGCT-CTCCTCTG
473673_ENSRNOG00000013246_RAT_7392_7632/1-245 ATGGGTCCCTACGCTCTGCTCATTTTCATCCCTGCTCTCTGTGCCGTGGCT-ATGATCTC
473673_ENSMUSG00000042953_MOUSE_6061_6302/1-245 ATGGGTCCCTACTCTCTGCTCATCTTCATCCCTGCGCTCTGCGCTGTGGCT-CTGGTCTC
473673_ENSDARG00000022302_ZEBRAFISH_7796_8038/1-245 ATGGGTCCATATAGCTCACTGCTGTTCCTGAGTGCTAT-TAAACTGCTGTTACTGATCCA
****** * ** ** * *** *** * * * * * * * **
473673_ENSG00000177669_HUMAN_4164_4404/1-245 TTCCCTGGCTCCTCAGCAAGTCCACAGGTGGACCTTCTGCTTTCAGATGAGCTGGCAGAC
473673_ENSRNOG00000013246_RAT_7392_7632/1-245 CTCCCTCAGTCCACAGGAAGTCCATGGGCTGACTTTCTTCTTTCAGATGGGTTGGCAAAC
473673_ENSMUSG00000042953_MOUSE_6061_6302/1-245 CTTCCTCAGTCCACAGGAAGTCCATAGGCTGACCTTCTTCTTTCAGATGGGCTGGCAGAC
473673_ENSDARG00000022302_ZEBRAFISH_7796_8038/1-245 CTATATACATCCAATGCATCTTCATCGGTGGATTCTGGGACTGCAGATGTTTTGGCAAAC
* * *** * * * ** ** ** * * ****** ***** **
473673_ENSG00000177669_HUMAN_4164_4404/1-245 CTTGTGTCACCTAGGTCTGCACTACACTGAGTATTATCTGCATGAGCCTCCTTCTGTGAG
473673_ENSRNOG00000013246_RAT_7392_7632/1-245 CCTGTGCCACCTGGGTCTTCACTACAAGGAGTACTACCTGTGTGAGCCTCCCCCTGTGAG
473673_ENSMUSG00000042953_MOUSE_6061_6302/1-245 CCTGTGCCATCTGGGTCTTCACTACACCGAATACTACCTGGGTGAGCCTCCACCCGTGAG
473673_ENSDARG00000022302_ZEBRAFISH_7796_8038/1-245 CTGCTGGCATTTGTACGTCCAGTACCAGATATACTGGCTTCAAGAGGCACCAGACTCAAG
* ** ** * * ** *** ** * ** *** * ** **
473673_ENSG00000177669_HUMAN_4164_4404/1-245 GTAA-
473673_ENSRNOG00000013246_RAT_7392_7632/1-245 GTAA-
473673_ENSMUSG00000042953_MOUSE_6061_6302/1-245 GTAAA
473673_ENSDARG00000022302_ZEBRAFISH_7796_8038/1-245 GTAT-
***
//
473673.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 245
Mean pairwise identity: 69.20
Mean single sequence MFE: -81.48
Consensus MFE: -36.48
Energy contribution: -36.85
Covariance contribution: 0.37
Mean z-score: -2.02
Structure conservation index: 0.45
SVM decision value: 0.41
SVM RNA-class probability: 0.726603
Prediction: RNA
######################################################################
>473673_ENSG00000177669_HUMAN_4164_4404/1-245
CUUCUCUUUCAGGUACCUCUUUCUCCUGACUGGAGGAGGUGCCCUGGCCGUGGCUGCCAUGGGUUCCUACGCCGUGCUCGUCUUCACCCCUGCUGUCUGCGCUGUGGCUCUCCUCUGUUCCCUGGCUCCUCAGCAAGUCCACAGGUGGACCUUCUGCUUUCAGAUGAGCUGGCAGACCUUGUGUCACCUAGGUCUGCACUACACUGAGUAUUAUCUGCAUGAGCCUCCUUCUGUGAGGUAA
.........((((((((((((((........)))))))))).))))((((..((.((.((((((....)).))))))..............((.....))))..))))...((((........(((((...((((.(((((....)))))....))))..(((((((....((((((((.(......).))))))))(((......))).)))))))...))))).........))))... ( -84.03)
>473673_ENSRNOG00000013246_RAT_7392_7632/1-245
CCUUUCUUUCAGGUACCUCUUUCUCCUGGCUGGAGGAGGUGUCCUGGCUUUGGCCGCCAUGGGUCCCUACGCUCUGCUCAUUUUCAUCCCUGCUCUCUGUGCCGUGGCUAUGAUCUCCUCCCUCAGUCCACAGGAAGUCCAUGGGCUGACUUUCUUCUUUCAGAUGGGUUGGCAAACCCUGUGCCACCUGGGUCUUCACUACAAGGAGUACUACCUGUGUGAGCCUCCCCCUGUGAGGUAA
(((.......)))((((((......((((..(((((((((.....(((....)))(((((((.....((((....((..............))....)))))))))))....))))))))).((((((((..((....)).))))))))..........))))..(((..(((..((((..........))))..((((.(((.((.......))))))))))))..)))....)))))). ( -79.04)
>473673_ENSMUSG00000042953_MOUSE_6061_6302/1-245
CCUUUCUUUCAGGUACCUCUUUCUCCUGGCUGGAGGAGGUGUCCUGGCUUUUGCUGCCAUGGGUCCCUACUCUCUGCUCAUCUUCAUCCCUGCGCUCUGCGCUGUGGCUCUGGUCUCCUUCCUCAGUCCACAGGAAGUCCAUAGGCUGACCUUCUUCUUUCAGAUGGGCUGGCAGACCCUGUGCCAUCUGGGUCUUCACUACACCGAAUACUACCUGGGUGAGCCUCCACCCGUGAGGUAAA
........(((((((((((((((........)))))))))).)))))..((((((..(((((((...........((((((((........((((...))))(((((((..((.......))..)).)))))(((((.....(((....)))....))))))))))))).(((((((((.(......).))))))......((((.(........).)))).)))...))))))).)))))) ( -86.10)
>473673_ENSDARG00000022302_ZEBRAFISH_7796_8038/1-245
UCAUCUCUUGUAAGGUAUGUCUAUUUGGCGAUGGGAGGAUUCAUGCUGGCUAUUGCAACAAUGGGUCCAUAUAGCUCACUGCUGUUCCUGAGUGCUAUUAAACUGCUGUUACUGAUCCACUAUAUACAUCCAAUGCAUCUUCAUCGGUGGAUUCUGGGACUGCAGAUGUUUUGGCAAACCUGCUGGCAUUUGUACGUCCAGUACCAGAUAUACUGGCUUCAAGAGGCACCAGACUCAAGGUAU
...(((((((...(((((((((...(((((..((((((((((((....((....))....))))))))..(((((.....)))))))))...)))))..........(.((((((((((((........................))))))))...((((((((((((((..((((....)))))))))))))).)))))))).)))))))))).....))))))).(((........))).. ( -76.76)
>consensus
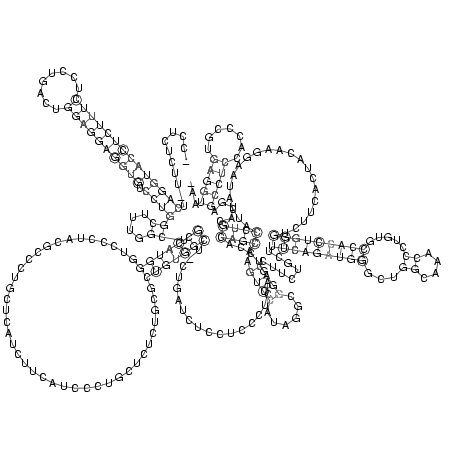
_CCUCUCUU_UCAGGUACCUCUUUCUCCUGACUGGAGGAGGUGUCCUGGCUUUGGCUGCCAUGGGUCCCUACGCCCUGCUCAUCUUCAUCCCUGCUCUCUGCGCUGUGGCU_CUGAUCUCCUCCCUCAGUCCACAGCAAGUCCAUAGGCGGACCUUCUGCUUUCAGAUGGGCUGGCAAACCCUGUGCCACCUGGGUCUUCACUACAAGGAAUACUACCUGCAUGAGCCUCCACCCGUGAGGUAA_
...........((((((((((((((........)))))))))).))))((....)).(((((((.......................................))))))).......................((((..((((......))))........(((((((((...((....)).....))))))))).....................)))).....(((((.......)))))... (-36.48 = -36.85 + 0.37)
473673.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004