Index >
Results for CNB 470004-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
470004_ENSG00000182512_HUMAN_8267_8506/1-243 GTACCCTCGAGCACACCAGACTTGCAGAAAAAGCATACTCCAGAGGAAGCTGAGGCATGC
470004_ENSMUSG00000021102_MOUSE_8376_8616/1-243 GTACCCTAGAGCACACCAGACTTGCAGAGAAAGCAGACAACAGAGGAAGCTGAGGCATGC
470004_SINFRUG00000145950_FUGU_5916_6140/1-243 --------GGGCACAACAAGGTTGC--AGAAAGCAT-C-CCAAAGGATGAAAAGGCGTGA
470004_ENSRNOG00000004206_RAT_8331_8570/1-243 -TACCCTCGAGCACACCAGACTTGCAGAAAAAGCATACTCCAGAGGAAGCTGAGGCATGC
* ***** ** **** * ****** * ** **** * **** **
470004_ENSG00000182512_HUMAN_8267_8506/1-243 CTGCTCGAGAGCCAGCTGTTCCATGTGCAATTTTCCTCTGATAGTTTCTGGTCACTGTTG
470004_ENSMUSG00000021102_MOUSE_8376_8616/1-243 CTGCTCGAGAGGCAGCAGGCCCATGTGCAATTTTCCTCTGATAGGGCCTGGGTACTGTTG
470004_SINFRUG00000145950_FUGU_5916_6140/1-243 CTTCCAGAGGTCTGGGAATTCCACGTGCGATGTT-CTTTGA-A---CAAACACCTGCTTT
470004_ENSRNOG00000004206_RAT_8331_8570/1-243 CTGCTCGAGAGCCAGCGGTTCCATGTGCAATTTTCCTCTGATAAATCCTGGGTACTGTTG
** * *** * *** **** ** ** ** *** * **
470004_ENSG00000182512_HUMAN_8267_8506/1-243 CCACGGTGATAATGACTGGGCTATGTCATTATCTATCCGCCAACAGTAAGAGAAGCTTTG
470004_ENSMUSG00000021102_MOUSE_8376_8616/1-243 CCACGGTGATAATGGCTGTGCCGTGTCATTGTCGATCCACCAACAGTCAGAGAAGCTTGG
470004_SINFRUG00000145950_FUGU_5916_6140/1-243 CCACGGTGGCGATGGCCTTGTTCTGTAGCCATCGCATTTCTTGAGGCAGGTAGAAAAAAA
470004_ENSRNOG00000004206_RAT_8331_8570/1-243 CCACGGTGATAATGACTGTGCCGTGTCATTGTCGATCCACCAACAGTCAGAGAAGCTTTG
******** *** * * *** ** * * * *
470004_ENSG00000182512_HUMAN_8267_8506/1-243 CAGTCGAGATATTGTTT--AGCAGATGGAGTGTTTTCTGTTGAACACTAAGTACTGCCAC
470004_ENSMUSG00000021102_MOUSE_8376_8616/1-243 CAGTCAAGATATTGTTT--AGCAGACGGAGCGGTTTCTGTTGGACACTAAGTACTGCCAC
470004_SINFRUG00000145950_FUGU_5916_6140/1-243 CAG-CATGATATTGTTTGAAGCGGGGAGAGCTGTTCCCGTCAGACACTGTCCGCTGTCAC
470004_ENSRNOG00000004206_RAT_8331_8570/1-243 CAGTCAAGATATTGTTT--AGCAGACGGAGCGGTTTCTGTTGGACACTAAGTGCTGCTAC
*** * ********** *** * *** ** * ** ***** *** **
470004_ENSG00000182512_HUMAN_8267_8506/1-243 AA-
470004_ENSMUSG00000021102_MOUSE_8376_8616/1-243 AAA
470004_SINFRUG00000145950_FUGU_5916_6140/1-243 AGC
470004_ENSRNOG00000004206_RAT_8331_8570/1-243 AAG
*
470004-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 243
Mean pairwise identity: 71.81
Mean single sequence MFE: -79.57
Consensus MFE: -48.69
Energy contribution: -49.87
Covariance contribution: 1.19
Mean z-score: -1.56
Structure conservation index: 0.61
SVM decision value: 0.39
SVM RNA-class probability: 0.717927
Prediction: RNA
######################################################################
>470004_ENSG00000182512_HUMAN_8267_8506/1-243
GUACCCUCGAGCACACCAGACUUGCAGAAAAAGCAUACUCCAGAGGAAGCUGAGGCAUGCCUGCUCGAGAGCCAGCUGUUCCAUGUGCAAUUUUCCUCUGAUAGUUUCUGGUCACUGUUGCCACGGUGAUAAUGACUGGGCUAUGUCAUUAUCUAUCCGCCAACAGUAAGAGAAGCUUUGCAGUCGAGAUAUUGUUUAGCAGAUGGAGUGUUUUCUGUUGAACACUAAGUACUGCCACAA
((((.((((((((((...((..(((.......)))...))....(((((((((((....)))(((....))))))))..))).))))).........(((..(((((((..(.(((((((...(((.(((((((((........)))))))))...))).))))))))..)))))))...))))))))....(((((((((((..........)))))))))))....))))........ ( -79.90)
>470004_ENSMUSG00000021102_MOUSE_8376_8616/1-243
GUACCCUAGAGCACACCAGACUUGCAGAGAAAGCAGACAACAGAGGAAGCUGAGGCAUGCCUGCUCGAGAGGCAGCAGGCCCAUGUGCAAUUUUCCUCUGAUAGGGCCUGGGUACUGUUGCCACGGUGAUAAUGGCUGUGCCGUGUCAUUGUCGAUCCACCAACAGUCAGAGAAGCUUGGCAGUCAAGAUAUUGUUUAGCAGACGGAGCGGUUUCUGUUGGACACUAAGUACUGCCACAAA
........((((...((((..((((.......))))....(((((((((.(...((((((((((((....)..)))))))....)))).).))))))))).......))))(.(((((((......((((((((((........))))))))))......))))))))......))))((((((.....((.(((((((((((..........))))))))))).))...))))))..... ( -82.40)
>470004_SINFRUG00000145950_FUGU_5916_6140/1-243
GGGCACAACAAGGUUGCAGAAAGCAUCCCAAAGGAUGAAAAGGCGUGACUUCCAGAGGUCUGGGAAUUCCACGUGCGAUGUUCUUUGAACAAACACCUGCUUUCCACGGUGGCGAUGGCCUUGUUCUGUAGCCAUCGCAUUUCUUGAGGCAGGUAGAAAAAAACAGCAUGAUAUUGUUUGAAGCGGGGAGAGCUGUUCCCGUCAGACACUGUCCGCUGUCACAGC
..((((.........((......(((((....))))).....))(((..((((((....))))))....)))))))..(((((...)))))...(((((((((........((((((((...........)))))))).......)))))))))........(((((..((((.((((((..(((((((......))))))))))))).)))).)))))...... ( -78.96)
>470004_ENSRNOG00000004206_RAT_8331_8570/1-243
UACCCUCGAGCACACCAGACUUGCAGAAAAAGCAUACUCCAGAGGAAGCUGAGGCAUGCCUGCUCGAGAGCCAGCGGUUCCAUGUGCAAUUUUCCUCUGAUAAAUCCUGGGUACUGUUGCCACGGUGAUAAUGACUGUGCCGUGUCAUUGUCGAUCCACCAACAGUCAGAGAAGCUUUGCAGUCAAGAUAUUGUUUAGCAGACGGAGCGGUUUCUGUUGGACACUAAGUGCUGCUACAAG
........(((((....((((.(((....((((......(((((((((.((..(((((.((((((....)..)))))...))))).))..))))))))).....(((((...(((((((......((((((((((........))))))))))......)))))))))).)).))))))))))).......(((((((((((..........)))))))))))....)))))........ ( -77.00)
>consensus
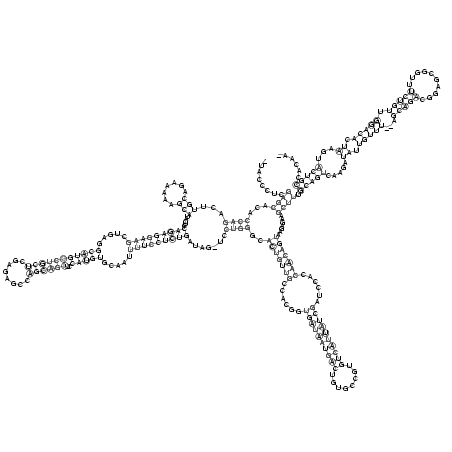
_UACCCUCGAGCACACCAGACUUGCAGAAAAAGCAUACUCCAGAGGAAGCUGAGGCAUGCCUGCUCGAGAGCCAGCAGUUCCAUGUGCAAUUUUCCUCUGAUAG_UCCUGGGCACUGUUGCCACGGUGAUAAUGACUGUGCCGUGUCAUUAUCGAUCCACCAACAGUAAGAGAAGCUUUGCAGUCAAGAUAUUGUUU__AGCAGACGGAGCGGUUUCUGUUGGACACUAAGUACUGCCACAA_
........((((...((((...(((.......))).....(((((((((.....(((((((((((........))))))..))))).....))))))))).......))))..(((((((......((((((((((........))))))))))......))))))).......)))).(((((.....((.(((((...(((((..........))))).))))).))...)))))...... (-48.69 = -49.87 + 1.19)
470004-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004