Index >
Results for CNB 461749
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
461749_ENSMUSG00000034255_MOUSE_8785_8897/1-115 -CCCTCCAGCTTCGAAGCCGAGATGGCTC-GGAGTACCTGATCCAGCACGACTCCGAAGC
461749_SINFRUG00000131017_FUGU_9484_9597/1-115 CCTCCCCAG-TTAAAAACTAAGAACGCGGTGGAATTCCTGATACAGTACGACACAGAAAG
461749_ENSRNOG00000028569_RAT_8775_8886/1-115 -CCCTCCAGCTCCGGAGCCGAGATGGCTC-AGAGTACCTGATCCAGCATGACTCAGAGGC
461749_ENSDARG00000006344_ZEBRAFISH_6914_7025/1-115 -TCATTCAGCTTAAGACTCGGACTGGTGC-CGAATACTTGATCCAGTACGACACCGATAG
*** * * * ** * * **** *** * *** * **
461749_ENSMUSG00000034255_MOUSE_8785_8897/1-115 CATCATCAGCACCTGGCACAAAGCCATTGCCGAAGGCATCTCAGAGCTGGTAGGC
461749_SINFRUG00000131017_FUGU_9484_9597/1-115 CATCATTGTTGACTGGCACCGAGTCCTGACCGATACCATACGACAACTGGTGAGA
461749_ENSRNOG00000028569_RAT_8775_8886/1-115 CATCATCAGCACCTGGCACAAGGCCATTGCCGAAGGCATCGAGGAGCTGGTAGG-
461749_ENSDARG00000006344_ZEBRAFISH_6914_7025/1-115 CATCATCCAAGACTGGCATAAAGTTATCTTAGATACCATACACCAGCTGGTGAG-
****** ****** * * ** *** * ***** *
//
461749.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 115
Mean pairwise identity: 65.01
Mean single sequence MFE: -39.80
Consensus MFE: -17.40
Energy contribution: -19.52
Covariance contribution: 2.13
Mean z-score: -2.61
Structure conservation index: 0.44
SVM decision value: 1.95
SVM RNA-class probability: 0.983664
Prediction: RNA
######################################################################
>461749_ENSMUSG00000034255_MOUSE_8785_8897/1-115
CCCUCCAGCUUCGAAGCCGAGAUGGCUCGGAGUACCUGAUCCAGCACGACUCCGAAGCCAUCAUCAGCACCUGGCACAAAGCCAUUGCCGAAGGCAUCUCAGAGCUGGUAGGC
.(((((((((..((.(((..(((((((((((((..(((...)))....)))))).))))))).((.(((..((((.....)))).))).)).)))...))..)))))).))). ( -53.40)
>461749_SINFRUG00000131017_FUGU_9484_9597/1-115
CCUCCCCAGUUAAAAACUAAGAACGCGGUGGAAUUCCUGAUACAGUACGACACAGAAAGCAUCAUUGUUGACUGGCACCGAGUCCUGACCGAUACCAUACGACAACUGGUGAGA
.(((.((((((........((.((.((((((....)).....((((.(((((..((.....))..)))))))))..)))).)).))...((........))..)))))).))). ( -28.40)
>461749_ENSRNOG00000028569_RAT_8775_8886/1-115
CCCUCCAGCUCCGGAGCCGAGAUGGCUCAGAGUACCUGAUCCAGCAUGACUCAGAGGCCAUCAUCAGCACCUGGCACAAGGCCAUUGCCGAAGGCAUCGAGGAGCUGGUAGG
.(((((((((((.(((((..((((((((.((((..(((...)))....)))).)).)))))).((.(((..((((.....)))).))).)).))).))..)))))))).))) ( -52.30)
>461749_ENSDARG00000006344_ZEBRAFISH_6914_7025/1-115
UCAUUCAGCUUAAGACUCGGACUGGUGCCGAAUACUUGAUCCAGUACGACACCGAUAGCAUCAUCCAAGACUGGCAUAAAGUUAUCUUAGAUACCAUACACCAGCUGGUGAG
((((.(((((......(((.(((((...(((....)))..))))).)))....((((((...(((((....))).))...))))))................))))))))). ( -25.10)
>consensus
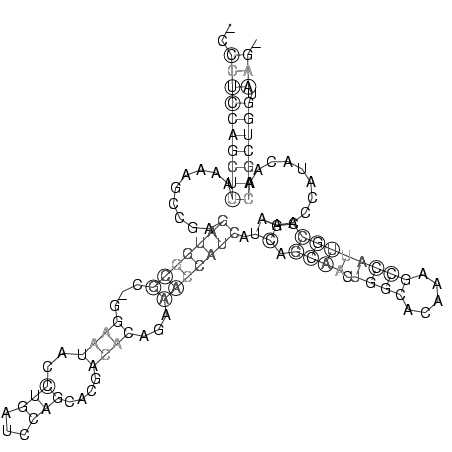
_CCCUCCAGCUUAAAAGCCGAGAUGGCGC_GGAAUACCUGAUCCAGCACGACACAGAAACCAUCAUCAGCAACUGGCACAAAGCCAUUGCCGAAACCAUACAACAGCUGGUAAG_
..((((((((((.........(((((((...((((..(((...)))....))))...))))))).((.((((.((((.....)))))))).))..........))))))).))). (-17.40 = -19.52 + 2.13)
461749.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004