Index >
Results for CNB 461745-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
461745_ENSMUSG00000034255_MOUSE_6286_6370/1-86 CACCTCTAACACATTCTTCTTGCTGGATTTGTCTTTGGGGGCCCAAGAGAGAGAGGCCCC
461745_SINFRUG00000131017_FUGU_9289_9373/1-86 -ACCTCCAAAACATTCTTTTTGCTGGATTTGTCTTTTGTGGCCCAGCAAATAGTCGCTCC
461745_ENSRNOG00000028569_RAT_5899_5982/1-86 -ACCTCTAGCACATTCTTCTTGCTGGATTTGTCTTTGGGGGCCCACGAGAGCGAGGCCCC
461745_ENSDARG00000006344_ZEBRAFISH_5690_5774/1-86 -ACCTCCAGAACGTTTTTCTTGCTTGATTTGTCCTTGGGCGCCCGGTTAATTGTGGCGCC
***** * ** ** ** ***** ******** ** * **** * * ** **
461745_ENSMUSG00000034255_MOUSE_6286_6370/1-86 CTTCAGTTCGACTGTGTATTCAGGG-
461745_SINFRUG00000131017_FUGU_9289_9373/1-86 TTTCAGGTCCACTGTGACCTCTGGGA
461745_ENSRNOG00000028569_RAT_5899_5982/1-86 CCTCAGTTCGACTGTATATTCAGGG-
461745_ENSDARG00000006344_ZEBRAFISH_5690_5774/1-86 TTTCAGATCCACTGTGAATTCTGGGA
**** ** ***** ** ***
461745-rev.aln
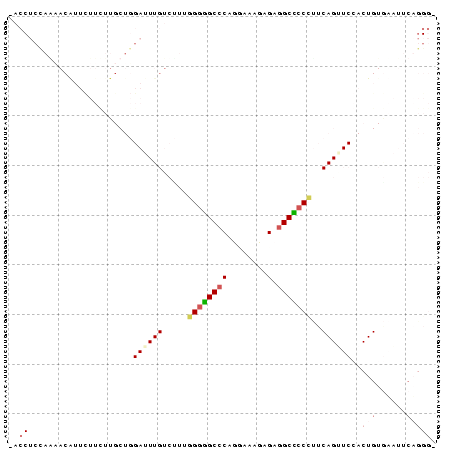
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 86
Mean pairwise identity: 77.15
Mean single sequence MFE: -26.90
Consensus MFE: -14.71
Energy contribution: -15.78
Covariance contribution: 1.06
Mean z-score: -2.06
Structure conservation index: 0.55
SVM decision value: 0.42
SVM RNA-class probability: 0.730303
Prediction: RNA
######################################################################
>461745_ENSMUSG00000034255_MOUSE_6286_6370/1-86
CACCUCUAACACAUUCUUCUUGCUGGAUUUGUCUUUGGGGGCCCAAGAGAGAGAGGCCCCCUUCAGUUCGACUGUGUAUUCAGGG
..(((...(((((.((.....((((((.........((((((((........).)))))))))))))..)).)))))....))). ( -28.20)
>461745_SINFRUG00000131017_FUGU_9289_9373/1-86
ACCUCCAAAACAUUCUUUUUGCUGGAUUUGUCUUUUGUGGCCCAGCAAAUAGUCGCUCCUUUCAGGUCCACUGUGACCUCUGGGA
..............((.((((((((....(((......))))))))))).))....((((...(((((......)))))..)))) ( -22.90)
>461745_ENSRNOG00000028569_RAT_5899_5982/1-86
ACCUCUAGCACAUUCUUCUUGCUGGAUUUGUCUUUGGGGGCCCACGAGAGCGAGGCCCCCCUCAGUUCGACUGUAUAUUCAGGG
.(((((((((.........))))))).........((((((((.(....).).)))))))..(((.....)))........)). ( -29.00)
>461745_ENSDARG00000006344_ZEBRAFISH_5690_5774/1-86
ACCUCCAGAACGUUUUUCUUGCUUGAUUUGUCCUUGGGCGCCCGGUUAAUUGUGGCGCCUUUCAGAUCCACUGUGAAUUCUGGGA
...(((((((.....(((......((((((.....((((((((((....))).)))))))..))))))......)))))))))). ( -27.50)
>consensus
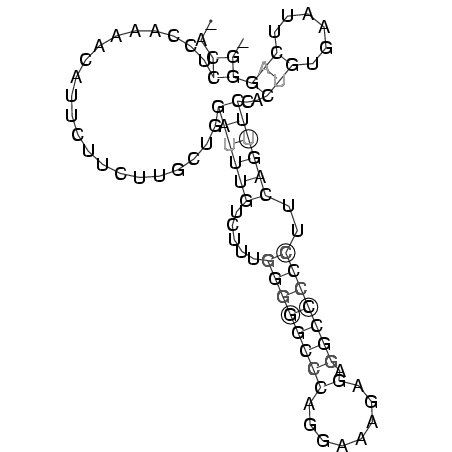
_ACCUCCAAAACAUUCUUCUUGCUGGAUUUGUCUUUGGGGGCCCAGGAAAGAGAGGCCCCCUUCAGUUCCACUGUGAAUUCAGGG_
..((.....................((((((.....((((((((........).)))))))..))))))..(((......))))). (-14.71 = -15.78 + 1.06)
461745-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004