Index >
Results for CNB 458276-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
458276_ENSG00000112245_HUMAN_7737_7838/1-102 ACAGTTCACTTGTCTGCATAGAGGTCGTGCTGTGCCTGGCAGTAATCTCCACTGCCCTTC
458276_SINFRUG00000146964_FUGU_4046_4146/1-102 -CAGTTCACTCGTCTGCATAGACAGCGTGCTGAGCCTGGCAGTAGTCTCCACTGCCCTTC
458276_ENSDARG00000006242_ZEBRAFISH_6072_6173/1-102 GCCGTTCACTTCTCTGCATAAACGCCGTGCTGAGCCTGGCAGTAATCCCCACTGCCCTTC
458276_ENSMUSG00000046524_MOUSE_9044_9143/1-102 ACAGTTCACTTGTCTGCATAGAGGTCGTGCTGTGCCTGGCAGTAATCTCCACTGCCCTTC
458276_ENSRNOG00000011771_RAT_9394_9495/1-102 ACAGTTCACTTGTCTGCATAGAGGTCGTGCTGTGCCTGGCAGTAGTCTCCACTGCCCTTC
* ******* ******** * ******* *********** ** ************
458276_ENSG00000112245_HUMAN_7737_7838/1-102 AGAAACTCCATAAATGTGTGACCAAGAACACCACAGAATTGC
458276_SINFRUG00000146964_FUGU_4046_4146/1-102 AGAAACTCCATAAATGTGTGACCCAGAACACCACAGAACTGC
458276_ENSDARG00000006242_ZEBRAFISH_6072_6173/1-102 AGAAACTCCATAAATGTGTGACCCAAAACACCACAGAACTGC
458276_ENSMUSG00000046524_MOUSE_9044_9143/1-102 AGAAACTCCATAAATGTG-GACCGAGAACACCACAGAACTG-
458276_ENSRNOG00000011771_RAT_9394_9495/1-102 AGAAACTCCATAAATGTGTGACCGAGAACACCACAGAACTGC
****************** **** * ************ **
458276-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 102
Mean pairwise identity: 92.16
Mean single sequence MFE: -26.44
Consensus MFE: -21.50
Energy contribution: -22.34
Covariance contribution: 0.84
Mean z-score: -1.33
Structure conservation index: 0.81
SVM decision value: -0.02
SVM RNA-class probability: 0.523322
Prediction: RNA
######################################################################
>458276_ENSG00000112245_HUMAN_7737_7838/1-102
ACAGUUCACUUGUCUGCAUAGAGGUCGUGCUGUGCCUGGCAGUAAUCUCCACUGCCCUUCAGAAACUCCAUAAAUGUGUGACCAAGAACACCACAGAAUUGC
.((((((.....(((((((((........))))))..((((((.......))))))....)))............((((........))))....)))))). ( -24.40)
>458276_SINFRUG00000146964_FUGU_4046_4146/1-102
CAGUUCACUCGUCUGCAUAGACAGCGUGCUGAGCCUGGCAGUAGUCUCCACUGCCCUUCAGAAACUCCAUAAAUGUGUGACCCAGAACACCACAGAACUGC
((((((....((((....))))...((((((((...((((((.......)))))).))))).............((((........))))))).)))))). ( -28.80)
>458276_ENSDARG00000006242_ZEBRAFISH_6072_6173/1-102
GCCGUUCACUUCUCUGCAUAAACGCCGUGCUGAGCCUGGCAGUAAUCCCCACUGCCCUUCAGAAACUCCAUAAAUGUGUGACCCAAAACACCACAGAACUGC
((.((((........((......)).((((((((...((((((.......)))))).))))).............((((........))))))).)))).)) ( -24.20)
>458276_ENSMUSG00000046524_MOUSE_9044_9143/1-102
ACAGUUCACUUGUCUGCAUAGAGGUCGUGCUGUGCCUGGCAGUAAUCUCCACUGCCCUUCAGAAACUCCAUAAAUGUGGACCGAGAACACCACAGAACUG
.((((((.....(((((((((........))))))..((((((.......))))))....)))...........(((((..........))))))))))) ( -28.10)
>458276_ENSRNOG00000011771_RAT_9394_9495/1-102
ACAGUUCACUUGUCUGCAUAGAGGUCGUGCUGUGCCUGGCAGUAGUCUCCACUGCCCUUCAGAAACUCCAUAAAUGUGUGACCGAGAACACCACAGAACUGC
.((((((.....(((((((((........))))))..((((((.......))))))....)))............((((........))))....)))))). ( -26.70)
>consensus
ACAGUUCACUUGUCUGCAUAGAGGUCGUGCUGUGCCUGGCAGUAAUCUCCACUGCCCUUCAGAAACUCCAUAAAUGUGUGACCCAGAACACCACAGAACUGC
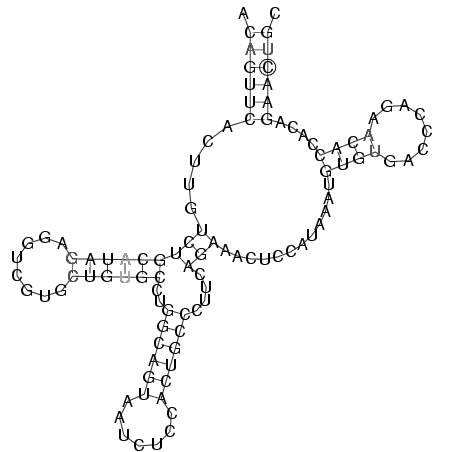
.((((((.....(((((((((........))))))..((((((.......))))))....)))............((((........))))....)))))). (-21.50 = -22.34 + 0.84)
458276-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004