Index >
Results for CNB 406997_2
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
406997_ENSDARG00000010275_ZEBRAFISH_7208_7547/1-353 GGCAGCGGCTCCCGGAGCTC--A-GAAATAGATTTCACTGTATTT-GAGAGCGCGCTGGC
406997_ENSRNOG00000006908_RAT_1091_1431/1-353 GGCAGGGGCCTCTGATCCAG--G-GCAATGGCTTTTACTGGATGT-GAGAGCACTCTTGG
406997_ENSG00000184486_HUMAN_1476_1817/1-353 GGCATGGGCCCCTGATCCAT--G-GCAATGGATTTTACTGGATGT-GAGAGCGCTCTTGG
406997_ENSMUSG00000052621_MOUSE_1401_1741/1-353 GGCAGGGGCCTCTGATCCAG--G-GCAATGGATTTTACTGGATGT-GAGAGCACTCTTGG
406997_SINFRUG00000138620_FUGU_8265_8616/1-353 GGCACAGAAACGTAGACAGTACGAGCGTCGGAGCACAACATGTGTTGACAGCACATCCGT
406997_ENSDARG00000010275_ZEBRAFISH_7208_7547/1-353 CCTTCCAAATGAATTACCGTAGACCCCACAGTCCA--AATAACAAGACTCGCATCATTTA
406997_ENSRNOG00000006908_RAT_1091_1431/1-353 TATTAGGAGTGACTCACTGAAAACCTTCCTAACCC--AATAACAAGA-TCACATCATGTA
406997_ENSG00000184486_HUMAN_1476_1817/1-353 TCTTAGGAGTGACTCACCACAAACCCACCCTTCCC--AATAACAAGA-TCACATCATGTA
406997_ENSMUSG00000052621_MOUSE_1401_1741/1-353 TATTAGGAGCGACTCACTGCAAACCTTCCTATCCC--AATAACAAGA-TCACATCATGTA
406997_SINFRUG00000138620_FUGU_8265_8616/1-353 CCCTTTTACTGAATTAACAGCGACCTCTTCATCTTGTAAGAACAGGACTCGCATCGTTTA
406997_ENSDARG00000010275_ZEBRAFISH_7208_7547/1-353 TGCGTTGTTAAGCCATGTAATTGAACGATCCATAGTTTGCATATTATACCATTAAAGTAA
406997_ENSRNOG00000006908_RAT_1091_1431/1-353 TGCATTATTAAGCTACATAATTGAACAGTCTATAGTTTGCATATCAGACTATTAAAATAA
406997_ENSG00000184486_HUMAN_1476_1817/1-353 TGCATTATTAAGCCACATAATTGAACGGTCCATAGTTTGCATATCAGACTATTAAAATAA
406997_ENSMUSG00000052621_MOUSE_1401_1741/1-353 TGCATTATTAAGCTACATAATTGAACAGTCTATAGTTTGCATATCAGACTATTAAAATAA
406997_SINFRUG00000138620_FUGU_8265_8616/1-353 TGCGTTATTAAGTCCCGCAATTGGGCAGCCAGTAGTTTGCATATTAGTCTATTAAGGTAA
406997_ENSDARG00000010275_ZEBRAFISH_7208_7547/1-353 TGTGCTTTGGTTTTCTCCCAACACGACTATTT-AAGCTTCTGTGGCGT--TGTGGTAATT
406997_ENSRNOG00000006908_RAT_1091_1431/1-353 TGTGTTTTGCTTTTCTCTGAACACGTATTGTTCAAGCCTCTG-CGAAT-ATGTGGTGATT
406997_ENSG00000184486_HUMAN_1476_1817/1-353 TGTGTTTTGCTTTTCTCTGAACACGTATTGTTCAAGCCTCTG-CGAGT-ATGTAGTGATT
406997_ENSMUSG00000052621_MOUSE_1401_1741/1-353 TGTGTTTTGCTTTTCTCTGAACACGTATTGTTCAAGCCTCTG-CGAAT-ATGTGGTGATT
406997_SINFRUG00000138620_FUGU_8265_8616/1-353 TATGTTTTGCCTGTCTCAACACACGGCTAGCCAGAGCCCCTGGCGACACATTTGGCAATT
406997_ENSDARG00000010275_ZEBRAFISH_7208_7547/1-353 CTCTGAATTACA-
406997_ENSRNOG00000006908_RAT_1091_1431/1-353 CTGATAATTATCA
406997_ENSG00000184486_HUMAN_1476_1817/1-353 CTGATAATTACCT
406997_ENSMUSG00000052621_MOUSE_1401_1741/1-353 CTGATAATTATCA
406997_SINFRUG00000138620_FUGU_8265_8616/1-353 CTCAGAATTACAC
406997_2.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 253
Mean pairwise identity: 72.80
Mean single sequence MFE: -67.08
Consensus MFE: -21.75
Energy contribution: -22.03
Covariance contribution: 0.28
Mean z-score: -1.75
Structure conservation index: 0.32
SVM decision value: 0.24
SVM RNA-class probability: 0.652482
Prediction: RNA
######################################################################
>406997_ENSDARG00000010275_ZEBRAFISH_7208_7547/1-353
GGCAGCGGCUCCCGGAGCUCAGAAAUAGAUUUCACUGUAUUUGAGAGCGCGCUGGCCCUUCCAAAUGAAUUACCGUAGACCCCACAGUCCAAAUAACAAGACUCGCAUCAUUUAUGCGUUGUUAAGCCAUGUAAUUGAACGAUCCAUAGUUUGCAUAUUAUACCAUUAAAGUAAUGUGCUUUGGUUUUCUCCCAACACGACUAUUUAAGCUUCUGUGGCGUUGUGGUAAUUCUCUGAAUUACA
((.((.((((..(((..((((((.((((......)))).))))))..).))..)))))).))....((((((((...(((.((((((......((((((....(((((.....)))))))))))(((..(((......)))....((((((.(((((((((.........))))))))).((((.......))))...))))))....))).)))))).)))..))))))))........... ( -66.30)
>406997_ENSRNOG00000006908_RAT_1091_1431/1-353
GGCAGGGGCCUCUGAUCCAGGGCAAUGGCUUUUACUGGAUGUGAGAGCACUCUUGGUAUUAGGAGUGACUCACUGAAAACCUUCCUAACCCAAUAACAAGAUCACAUCAUGUAUGCAUUAUUAAGCUACAUAAUUGAACAGUCUAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUGCGAAUAUGUGGUGAUUCUGAUAAUUAUCA
.(((((((((((((...)))))...((((((.(((..((((((((((((((((((....)))))))).)))...(((....))).................)))))))..))).........)))))).....(((((((((..((((((((.....)))))))).......((((((((...........))))))))))))))))))))))))).......((((((((.....)))))))) ( -68.50)
>406997_ENSG00000184486_HUMAN_1476_1817/1-353
GGCAUGGGCCCCUGAUCCAUGGCAAUGGAUUUUACUGGAUGUGAGAGCGCUCUUGGUCUUAGGAGUGACUCACCACAAACCCACCCUUCCCAAUAACAAGAUCACAUCAUGUAUGCAUUAUUAAGCCACAUAAUUGAACGGUCCAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUGCGAGUAUGUAGUGAUUCUGAUAAUUACCU
((...(((..((.(((((((....))))))).....)).((((.(((((((((((....)))))))).)))..))))......)))...)).....((..(((((..((((..((((((((........))))(((((((((..((((((((.....)))))))).......((((((((...........))))))))))))))))).....))))..))))..)))))..)).......... ( -63.60)
>406997_ENSMUSG00000052621_MOUSE_1401_1741/1-353
GGCAGGGGCCUCUGAUCCAGGGCAAUGGAUUUUACUGGAUGUGAGAGCACUCUUGGUAUUAGGAGCGACUCACUGCAAACCUUCCUAUCCCAAUAACAAGAUCACAUCAUGUAUGCAUUAUUAAGCUACAUAAUUGAACAGUCUAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUGCGAAUAUGUGGUGAUUCUGAUAAUUAUCA
.((((((((....((((((......))))))......(((((((........((((...((((((((......)))......)))))..))))........)))))))(((((.((........)))))))..(((((((((..((((((((.....)))))))).......((((((((...........))))))))))))))))))))))))).......((((((((.....)))))))) ( -67.89)
>406997_SINFRUG00000138620_FUGU_8265_8616/1-353
GGCACAGAAACGUAGACAGUACGAGCGUCGGAGCACAACAUGUGUUGACAGCACAUCCGUCCCUUUUACUGAAUUAACAGCGACCUCUUCAUCUUGUAAGAACAGGACUCGCAUCGUUUAUGCGUUAUUAAGUCCCGCAAUUGGGCAGCCAGUAGUUUGCAUAUUAGUCUAUUAAGGUAAUAUGUUUUGCCUGUCUCAACACACGGCUAGCCAGAGCCCCUGGCGACACAUUUGGCAAUUCUCAGAAUUACAC
(((.....((((((..(((((.(((.(.((((((.((((....))))...))...)))).).))).)))))........(((((((.(((.........))).)))..))))........)))))).....((((.......)))).))).(((((((((((((((.(((....))))))))))).(((((((((....(....)....(((((.....))))))))).....)))))......))))))).. ( -69.10)
>consensus
GGCAGGGGCCCCUGAUCCAG__G_GCAAUGGAUUUUACUGGAUGU_GAGAGCACUCUUGGUCUUAGGAGUGACUCACCGCAAACCUCCCCAUCCC__AAUAACAAGA_UCACAUCAUGUAUGCAUUAUUAAGCCACAUAAUUGAACAGUCCAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUG_CGAAU_AUGUGGUGAUUCUGAUAAUUACCA
(((...(((.....((((((.................)))))).......(((...............(((......))).....................(((.((......)).))).)))........)))......((((((((...((((((((.....)))))))).......((((((((...........)))))))).)))))))))))................((((((.....)))))).. (-21.75 = -22.03 + 0.28)
406997_2.rnaz
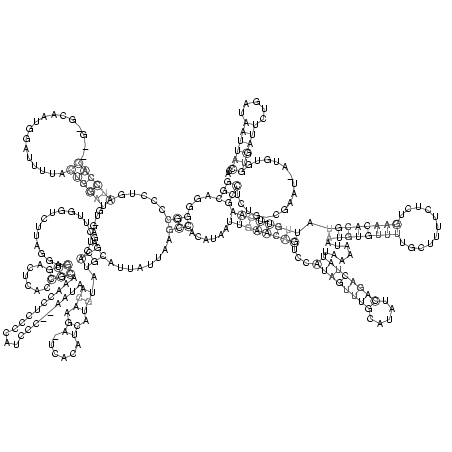
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004