Index >
Results for CNB 406119
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
406119_ENSG00000156925_HUMAN_9249_9455/1-209 CCCAATCAGCGGGGGTCGCTCGGCCGCGGCTGCCATGTTCTCCGCTCCGCGCTGCCAATC
406119_ENSMUSG00000005314_MOUSE_9261_9468/1-209 CCCAATCAGCTGGGGTCGCCCGGCAGCGGCTGCCATGTTCTCCGCTTCGCGCTGCCAATC
406119_ENSRNOG00000000861_RAT_9138_9344/1-209 CCCAATCAGCTGTGGTCGCC-GGCAGCGGCTGCCATGTTCTCCGCTTCGCGCTGCCAATC
406119_ENSDARG00000009849_ZEBRAFISH_9146_9352/1-209 ACCAATCAAATCTACTCACT-GAAACGAACTGCCATTTTCAATACTAAACCTTGCCAATC
406119_SINFRUG00000136278_FUGU_6731_6937/1-209 -CCAATCACACGAACCCAATTGAGACGAGCTGCCATCTCCAATACCAAACGGTGCCAATC
******* * * ******* * * * * ********
406119_ENSG00000156925_HUMAN_9249_9455/1-209 AGCCTCGCGGCGGCCAATGGGCAGTCCGGCCGGAGGTCAGGTGACCGCGGGCCAGCTGGC
406119_ENSMUSG00000005314_MOUSE_9261_9468/1-209 ATTGTGTCGGTGGCCAATGGGCGACAGGGCCGGGGGTCAGGTGATCTCAGGCCAGCTGGC
406119_ENSRNOG00000000861_RAT_9138_9344/1-209 AGTGTGGCGGCGGCCAATGGGCGGCAGGGCCGGGGGTCAGGTGATCTCAGGCCAGCTGGC
406119_ENSDARG00000009849_ZEBRAFISH_9146_9352/1-209 AACGAACGTGCAGCCAATTAACGCCCTTCCTATCCATCACGTGACTAAGAGGGAGCTGTG
406119_SINFRUG00000136278_FUGU_6731_6937/1-209 ACCGCGGAGCCGACCAACCAGAGGCTGCCTGCGCGGTCACGTGATCGCCGGGTAGCTGGG
* **** *** **** * *****
406119_ENSG00000156925_HUMAN_9249_9455/1-209 TCCCCATTGGCGCGCGCCTCGCGGCCTCCCGCTCGG-TTTATGTGCGAGGAGTGAGTGAT
406119_ENSMUSG00000005314_MOUSE_9261_9468/1-209 TCCCCATTGGTGCGCGCTGCCCAGCCTCCCGCTCGG-TTTATGTGCGAGGAGTGAGTGAT
406119_ENSRNOG00000000861_RAT_9138_9344/1-209 TCCCCATTGGTGCGCGCTGCCCAGCCTCCCGCTCGG-TTTATGTGCGAGGAGTGAGTGAT
406119_ENSDARG00000009849_ZEBRAFISH_9146_9352/1-209 CTTTCATTGGACAGCGTAGAGCTGCCTCCCGAGCTCATTTATGTGCAAGGAGTGAGTGAT
406119_SINFRUG00000136278_FUGU_6731_6937/1-209 CGCTGATTGGCTGTCGTAACACTGCCTCCCGAGTTCATTTATGTTCTGGGAGTGAGTGAT
***** ** * ******** ******* * ************
406119_ENSG00000156925_HUMAN_9249_9455/1-209 TGACTTTATCAGTCCAAGGACATTACTC-
406119_ENSMUSG00000005314_MOUSE_9261_9468/1-209 TGACTTTATCAGTCCAAGGACATTACTCT
406119_ENSRNOG00000000861_RAT_9138_9344/1-209 TGACTTTATCAGTCCAAGGACATTACTCT
406119_ENSDARG00000009849_ZEBRAFISH_9146_9352/1-209 TGACTTTATCAGTCCAAGGACATCATTC-
406119_SINFRUG00000136278_FUGU_6731_6937/1-209 TGACTTTATCAGTCCAAGGACATCATTC-
*********************** * **
//
406119.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 209
Mean pairwise identity: 71.92
Mean single sequence MFE: -83.78
Consensus MFE: -52.44
Energy contribution: -53.44
Covariance contribution: 1.00
Mean z-score: -1.69
Structure conservation index: 0.63
SVM decision value: 1.02
SVM RNA-class probability: 0.902041
Prediction: RNA
######################################################################
>406119_ENSG00000156925_HUMAN_9249_9455/1-209
CCCAAUCAGCGGGGGUCGCUCGGCCGCGGCUGCCAUGUUCUCCGCUCCGCGCUGCCAAUCAGCCUCGCGGCGGCCAAUGGGCAGUCCGGCCGGAGGUCAGGUGACCGCGGGCCAGCUGGCUCCCCAUUGGCGCGCGCCUCGCGGCCUCCCGCUCGGUUUAUGUGCGAGGAGUGAGUGAUUGACUUUAUCAGUCCAAGGACAUUACUC
...((((((((((((((((.((((((.(((((((.((((..(((((.((.((((.....))))..)).)))))..)))))))))))))))))(((((..(....).(((.(((((.(((....)))))))).)))))))))))))).)))))).)))).((((.(..(((.(((.(((......)))))).)))..).))))..... ( -104.70)
>406119_ENSMUSG00000005314_MOUSE_9261_9468/1-209
CCCAAUCAGCUGGGGUCGCCCGGCAGCGGCUGCCAUGUUCUCCGCUUCGCGCUGCCAAUCAUUGUGUCGGUGGCCAAUGGGCGACAGGGCCGGGGGUCAGGUGAUCUCAGGCCAGCUGGCUCCCCAUUGGUGCGCGCUGCCCAGCCUCCCGCUCGGUUUAUGUGCGAGGAGUGAGUGAUUGACUUUAUCAGUCCAAGGACAUUACUCU
........(((((((((((((((((((((..((..........))..).)))))))....((((.((.....))))))))))))).(.((((((((((((.((..(...)..)).)))))))))....))).)......))))))(((((((.((.....)).))).)))).((((((((((.....)))(((....)))))))))). ( -89.70)
>406119_ENSRNOG00000000861_RAT_9138_9344/1-209
CCCAAUCAGCUGUGGUCGCCGGCAGCGGCUGCCAUGUUCUCCGCUUCGCGCUGCCAAUCAGUGUGGCGGCGGCCAAUGGGCGGCAGGGCCGGGGGUCAGGUGAUCUCAGGCCAGCUGGCUCCCCAUUGGUGCGCGCUGCCCAGCCUCCCGCUCGGUUUAUGUGCGAGGAGUGAGUGAUUGACUUUAUCAGUCCAAGGACAUUACUCU
..((((((.(((((((.((((....)))).)))))......((((((.((((((((.......))))))))(((((((((.(((..((((.((((((....)))))).)))).....))).))))))))).(((((.(((.(((.....))).)))....))))).)))))))))))))).........(((....)))........ ( -95.60)
>406119_ENSDARG00000009849_ZEBRAFISH_9146_9352/1-209
ACCAAUCAAAUCUACUCACUGAAACGAACUGCCAUUUUCAAUACUAAACCUUGCCAAUCAACGAACGUGCAGCCAAUUAACGCCCUUCCUAUCCAUCACGUGACUAAGAGGGAGCUGUGCUUUCAUUGGACAGCGUAGAGCUGCCUCCCGAGCUCAUUUAUGUGCAAGGAGUGAGUGAUUGACUUUAUCAGUCCAAGGACAUCAUUC
.....((((.((.(((((((.....(((........))).........((((((...((((.(((.(..((((.........(((((....(((.....).))....))))).))))..).))).))))...((((((((((........)))))...))))))))))))))))))))))))........(((....)))....... ( -53.00)
>406119_SINFRUG00000136278_FUGU_6731_6937/1-209
CCAAUCACACGAACCCAAUUGAGACGAGCUGCCAUCUCCAAUACCAAACGGUGCCAAUCACCGCGGAGCCGACCAACCAGAGGCUGCCUGCGCGGUCACGUGAUCGCCGGGUAGCUGGGCGCUGAUUGGCUGUCGUAACACUGCCUCCCGAGUUCAUUUAUGUUCUGGGAGUGAGUGAUUGACUUUAUCAGUCCAAGGACAUCAUUC
.(((((((((((........((((.(......).))))....(((....)))((((((((.(((((......))..(((...((((((((.((((((....)))))))))))))))))))).))))))))..))))........(((((.((..((....))..)))))))...))))))).........(((....)))....... ( -75.90)
>consensus
CCCAAUCAGCUGGGGUCGCU_GGCAGCGGCUGCCAUGUUCUCCGCUACGCGCUGCCAAUCAGCGUGGCGGCGGCCAAUGGGCGGCAGGGCCGGCGGUCAGGUGAUCGCAGGCCAGCUGGCUCCCCAUUGGCGCGCGCAGCCCAGCCUCCCGCUCGG_UUUAUGUGCGAGGAGUGAGUGAUUGACUUUAUCAGUCCAAGGACAUUACUC_
......((((((..(((....)))..)))))).................((((((((.......))))))))(((((((((.((((.((((.((((((....)))))).))))....)))).)))))))))....(((....)))(((((((.(........).))).)))).((((((((((.....)))(((....)))))))))). (-52.44 = -53.44 + 1.00)
406119.rnaz
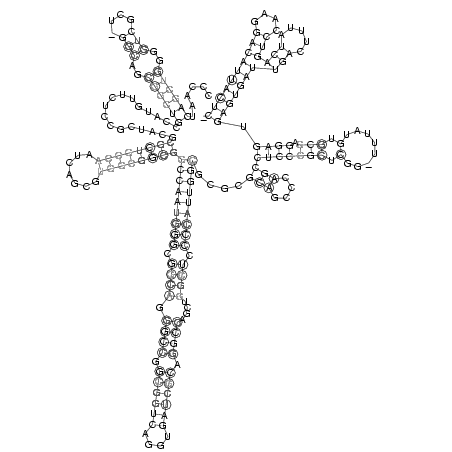
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004