Index >
Results for CNB 388305-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
388305_ENSG00000130589_HUMAN_8665_8892/1-228 GGGCCTCCTCGCGACTGCGGGCAAAGGTGCACCGGTTTCGGTGGCGCCGGCAGCCGAGCC
388305_ENSRNOG00000013267_RAT_1103_1330/1-228 GTGCCTCCTCTGGGCTGCTGGCAAAAGTGCATCTGTTCCAGTGCCGCTTACAGCCCAACC
388305_ENSDARG00000012600_ZEBRAFISH_9662_9883/1-228 CAGCCTCTTCTGGACTGCTGGCAAACGTGCATCTGTCTCGATGTATACGGCACCCCACAT
388305_ENSMUSG00000027580_MOUSE_1064_1289/1-228 -TGCCTCCTCAGGGCTTCGGGCAAAGGTGCATCTATTCCAGTGACGCCTACAGCCCAACC
***** ** * ** * ****** ***** * * * ** ** ** *
388305_ENSG00000130589_HUMAN_8665_8892/1-228 CAGGGCTGTAGTAGTGGCAGACTTGGTAGCACAGGGGCCTTGGGAAGGTGGGCCGGCAGC
388305_ENSRNOG00000013267_RAT_1103_1330/1-228 CTGGGCTATAATAGCGACAGACTTGATAGCGCACAGGTATTGGAAAGCTGGGCCTGAGGC
388305_ENSDARG00000012600_ZEBRAFISH_9662_9883/1-228 CCTCCTGAAAATACCAGCAAACCTCGTATCGTTGCGGTCGTGGGTGAGTGGGCCGCTGGA
388305_ENSMUSG00000027580_MOUSE_1064_1289/1-228 CTGGCCTATAGTAGTGACAGACTTGATAGCGCATAGGCGTTGGGAAGCTAGGCCTGCGGC
* * ** ** ** * ** * ** *** * **** *
388305_ENSG00000130589_HUMAN_8665_8892/1-228 CCACCACGCGCCAGACCTTGCTCTTGGTGGCCTGCTTAAAGCGGGCCAGCAGGATCTCGC
388305_ENSRNOG00000013267_RAT_1103_1330/1-228 CCACCTTACGCCAGACCTTGTTCTTGGCTGCTTGCTTGAAGCGGGCCAGCAAGATCTCCC
388305_ENSDARG00000012600_ZEBRAFISH_9662_9883/1-228 ACA-CACG-GCTCCATCTGGAACCT-CTGACCT--TTG-CTCTGGCTAGCAGGACATCAT
388305_ENSMUSG00000027580_MOUSE_1064_1289/1-228 CCACCTTACGCCAGATCTTGCTCTCTGCTGCTTGCTTGAAGCGGGCCAGCAAGATCTCCC
** * ** * ** * * * * ** * *** **** ** **
388305_ENSG00000130589_HUMAN_8665_8892/1-228 GGGAGCAGTCATGCTCCACCCTACGGAGGACGTAGGTGCTTTCATTGA
388305_ENSRNOG00000013267_RAT_1103_1330/1-228 TGGAGCAGGTATGCTCTACCTCACGTAGAATATAGGTGCTCTCATTGA
388305_ENSDARG00000012600_ZEBRAFISH_9662_9883/1-228 GAGAGCAACGGTGAGGTGTTGCAATGATGGTGTAGGAAATGTGAGAGT
388305_ENSMUSG00000027580_MOUSE_1064_1289/1-228 TGGGGCAGGTGTGCTCTACCTCACGCAGAATGTAGGTACTCTCATTG-
* *** ** * * **** * * * *
388305-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 228
Mean pairwise identity: 66.37
Mean single sequence MFE: -93.15
Consensus MFE: -43.31
Energy contribution: -48.50
Covariance contribution: 5.19
Mean z-score: -1.81
Structure conservation index: 0.46
SVM decision value: 0.47
SVM RNA-class probability: 0.750737
Prediction: RNA
######################################################################
>388305_ENSG00000130589_HUMAN_8665_8892/1-228
GGGCCUCCUCGCGACUGCGGGCAAAGGUGCACCGGUUUCGGUGGCGCCGGCAGCCGAGCCCAGGGCUGUAGUAGUGGCAGACUUGGUAGCACAGGGGCCUUGGGAAGGUGGGCCGGCAGCCCACCACGCGCCAGACCUUGCUCUUGGUGGCCUGCUUAAAGCGGGCCAGCAGGAUCUCGCGGGAGCAGUCAUGCUCCACCCUACGGAGGACGUAGGUGCUUUCAUUGA
((((.....(((((..(((((....(((((((((....))))(((((((((.(((...(((((((((...((....))...((((......)))))))))))))..)))..)))))).)))......)))))....)))))(((((.((((((((.....)))))))).)))))..)))))((((((....))))))..((((((.....)))))).))))....... ( -115.80)
>388305_ENSRNOG00000013267_RAT_1103_1330/1-228
GUGCCUCCUCUGGGCUGCUGGCAAAAGUGCAUCUGUUCCAGUGCCGCUUACAGCCCAACCCUGGGCUAUAAUAGCGACAGACUUGAUAGCGCACAGGUAUUGGAAAGCUGGGCCUGAGGCCCACCUUACGCCAGACCUUGUUCUUGGCUGCUUGCUUGAAGCGGGCCAGCAAGAUCUCCCUGGAGCAGGUAUGCUCUACCUCACGUAGAAUAUAGGUGCUCUCAUUGA
..((((.....))))(((((((......(((.(((...))))))(((((..((((((....)))))).....((((((((.((....((.(((.((((.((((.(((.((((((...)))))).)))...))))))))))).)).))))).)))))..))))).)))))))..........((((((..((((.(((((.....))))).))))..))))))...... ( -85.10)
>388305_ENSDARG00000012600_ZEBRAFISH_9662_9883/1-228
CAGCCUCUUCUGGACUGCUGGCAAACGUGCAUCUGUCUCGAUGUAUACGGCACCCCACAUCCUCCUGAAAAUACCAGCAAACCUCGUAUCGUUGCGGUCGUGGGUGAGUGGGCCGCUGGAACACACGGCUCCAUCUGGAACCUCUGACCUUUGCUCUGGCUAGCAGGACAUCAUGAGAGCAACGGUGAGGUGUUGCAAUGAUGGUGUAGGAAAUGUGAGAGU
....(((((..((..(((((......(((((((......))))))).)))))..))((((..((((....(((((((((((((((..((((((((..((((((((.((.((((((.((.....))))))(((....))).)).)).)))..((..(((.....)))..)).)))))..))))))))))))).)))).....)))))))))).))))))))). ( -81.80)
>388305_ENSMUSG00000027580_MOUSE_1064_1289/1-228
UGCCUCCUCAGGGCUUCGGGCAAAGGUGCAUCUAUUCCAGUGACGCCUACAGCCCAACCCUGGCCUAUAGUAGUGACAGACUUGAUAGCGCAUAGGCGUUGGGAAGCUAGGCCUGCGGCCCACCUUACGCCAGAUCUUGCUCUCUGCUGCUUGCUUGAAGCGGGCCAGCAAGAUCUCCCUGGGGCAGGUGUGCUCUACCUCACGCAGAAUGUAGGUACUCUCAUUG
.((((((((((((.....(((.(((((((((........)))..((((((..((((((((((((((((...(((.....)))..)))).)).)))).))))))..).)))))........))))))..)))((((((((((.((((((.(......).))))))..)))))))))))))))))).))))((((.((((.((.....))..))))))))........ ( -89.90)
>consensus
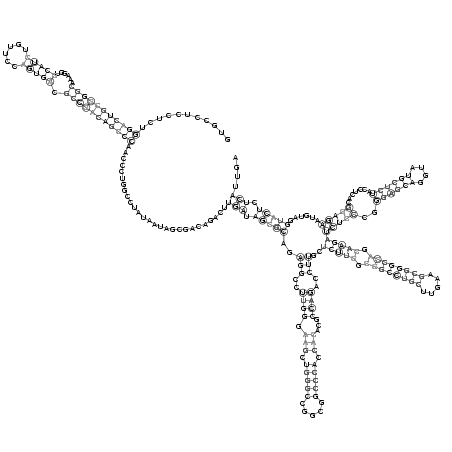
GUGCCUCCUCUGGACUGCGGGCAAAGGUGCAUCUGUUCCAGUGACGCCGACAGCCCAACCCUGGCCUAUAAUAGCGACAGACUUGAUAGCGCAGAGGCCUUGGGAAGCUGGGCCGGCGGCCCACCACACGCCAGACCUUGCUCUUGGCGGCCUGCUUGAAGCGGGCCAGCAAGAUCUCCCGGGAGCAGGUAUGCUCUACCUCACGGAGAAUGUAGGUACUCUCAUUGA
...........(((((((((((......(((((......))))).)))))))))))...........................(((.(((((..(((..((((.(((.(((((.....))))).)))...))))..)))..(((((.((((((((.....)))))))).)))))(((((..((((((....)))))).......)))))......))))).))).... (-43.31 = -48.50 + 5.19)
388305-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004