Index >
Results for CNB 386639-rev
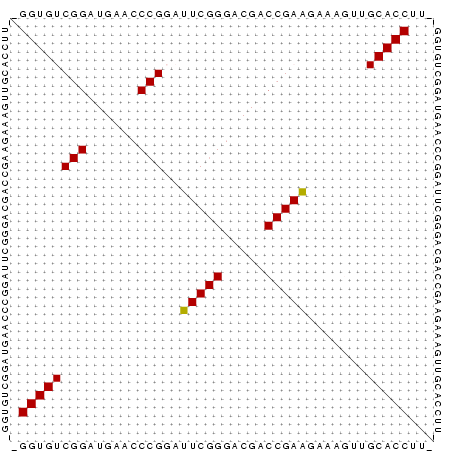
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
386639_ENSG00000158066_HUMAN_940_989/1-50 TGGTGTCGGATGAACCCGGATTCGGGACGACCGAAGGAAGTTGCACCTTG
386639_ENSDARG00000006691_ZEBRAFISH_909_956/1-50 -GGTGTCGGATGAACCCGGATTCGGGACGACCGAAAACTGTTGCACCTT-
386639_ENSMUSG00000051666_MOUSE_9549_9598/1-50 TGGTGTCGGATGAACCCGGATTCGGGACGACCGAAGAAAGTTGCACCGTT
386639_SINFRUG00000134705_FUGU_6366_6413/1-50 -GGTGTCGGATGAACCCGCAGTCGGGACGACCGATTACTGTTGCACCTT-
***************** * ************* ******** *
386639-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 50
Mean pairwise identity: 87.58
Mean single sequence MFE: -16.25
Consensus MFE: -16.33
Energy contribution: -15.95
Covariance contribution: -0.38
Mean z-score: -1.17
Structure conservation index: 1.00
SVM decision value: 0.30
SVM RNA-class probability: 0.676188
Prediction: RNA
######################################################################
>386639_ENSG00000158066_HUMAN_940_989/1-50
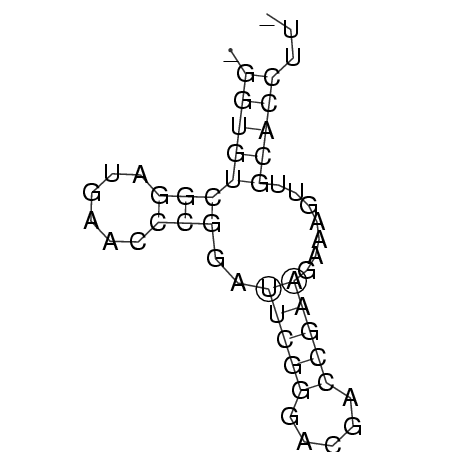
UGGUGUCGGAUGAACCCGGAUUCGGGACGACCGAAGGAAGUUGCACCUUG
.((((((((......)))..(((((.....))))).......)))))... ( -16.10)
>386639_ENSDARG00000006691_ZEBRAFISH_909_956/1-50
GGUGUCGGAUGAACCCGGAUUCGGGACGACCGAAAACUGUUGCACCUU
((((((((......)))..(((((.....))))).......))))).. ( -16.10)
>386639_ENSMUSG00000051666_MOUSE_9549_9598/1-50
UGGUGUCGGAUGAACCCGGAUUCGGGACGACCGAAGAAAGUUGCACCGUU
.((((((((......)))..(((((.....))))).......)))))... ( -16.60)
>386639_SINFRUG00000134705_FUGU_6366_6413/1-50
GGUGUCGGAUGAACCCGCAGUCGGGACGACCGAUUACUGUUGCACCUU
((((((((......))).((((((.....))))))......))))).. ( -16.20)
>consensus
_GGUGUCGGAUGAACCCGGAUUCGGGACGACCGAAGAAAGUUGCACCUU_
.((((((((......)))..(((((.....))))).......)))))... (-16.33 = -15.95 + -0.38)
386639-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004