Index >
Results for CNB 291556-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
291556_ENSMUSG00000024206_MOUSE_3691_3899/1-218 -AAAACACATGCAGATGCTGGATGGGAACCGGGCGCCGGC-CTC-TGGCGC-CGCGTGC-
291556_ENSDARG00000013575_ZEBRAFISH_1285_1497/1-218 CAAAGCACATGCAGATGTTCAATTAAAAACAGGAGCCCACAGTCTTGGCTCTTGTTTGCA
291556_SINFRUG00000129155_FUGU_972_1188/1-218 AAAAACACATACAGATGTTCAATTAAAAACAGGAGCCTACAGTCTTGGCTCGTGTTTGAA
291556_ENSRNOG00000017626_RAT_1660_1867/1-218 -AAAACACATGCAGATGCTGGATGGGAACCCGGCGCAGGC-CTC-TGGCGC-CGCGTGC-
*** ***** ****** * ** ** * ** ** * ** **** * * **
291556_ENSMUSG00000024206_MOUSE_3691_3899/1-218 CCAGCCCTCATTGTTTGCTGAGTAACAACGGTTGCCAGGGGCGACGCAGTCCACTCCCGT
291556_ENSDARG00000013575_ZEBRAFISH_1285_1497/1-218 CTGGCCCACATTGTTTGCAGAGCAACAACGGTTGCTAGTGGCAACACCGTCCACTCCCGT
291556_SINFRUG00000129155_FUGU_972_1188/1-218 CTGGCCCACATTGTTTGCAGAGCAACAACCGTTGCTATGGGCAACACTGTCCACTCCCGT
291556_ENSRNOG00000017626_RAT_1660_1867/1-218 CCAGCCCTCATTGTTTGCTGAGTAACAACGGTTGCCAGGGGCGACGCAGTCCACTCCCGT
* **** ********** *** ****** ***** * *** ** * ************
291556_ENSMUSG00000024206_MOUSE_3691_3899/1-218 TTCCAAGCGCCCGTTGCCCAGCAACCCAGAGAATACTTCCCAAACCAAACAATGTCATCC
291556_ENSDARG00000013575_ZEBRAFISH_1285_1497/1-218 TTCCAGGAGCCTGTTGCCATGCAACCAACAGAATACTTCCCAAACCAAACAATGCCATCC
291556_SINFRUG00000129155_FUGU_972_1188/1-218 TTCCAGGAGCCTGTTGCTAGGCGACCAACAGAATACTTCCCAAACCAAACAATGCCACCC
291556_ENSRNOG00000017626_RAT_1660_1867/1-218 TTCCAAGCGCCCGTTGCCCAGCAACCCGTAGAATACTTCCCAAACCAAACAATGTCATCC
***** * *** ***** ** *** ************************* ** **
291556_ENSMUSG00000024206_MOUSE_3691_3899/1-218 CCGGGGCAAC----TTCTCTATCTCACGTTAAGCAGAT
291556_ENSDARG00000013575_ZEBRAFISH_1285_1497/1-218 CCCAAGTGAT----CCTTCTATCTCACATTAACCAGA-
291556_SINFRUG00000129155_FUGU_972_1188/1-218 CCCTCCCCACCTGATCCTCTGTTACACGCTAAGTAGA-
291556_ENSRNOG00000017626_RAT_1660_1867/1-218 CCGGGGCAAC----TTCTCTATCTCACGTTAAGCAGA-
** * *** * *** *** ***
291556-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 218
Mean pairwise identity: 79.35
Mean single sequence MFE: -64.02
Consensus MFE: -40.03
Energy contribution: -42.97
Covariance contribution: 2.94
Mean z-score: -2.11
Structure conservation index: 0.63
SVM decision value: 0.94
SVM RNA-class probability: 0.886496
Prediction: RNA
######################################################################
>291556_ENSMUSG00000024206_MOUSE_3691_3899/1-218
AAAACACAUGCAGAUGCUGGAUGGGAACCGGGCGCCGGCCUCUGGCGCCGCGUGCCCAGCCCUCAUUGUUUGCUGAGUAACAACGGUUGCCAGGGGCGACGCAGUCCACUCCCGUUUCCAAGCGCCCGUUGCCCAGCAACCCAGAGAAUACUUCCCAAACCAAACAAUGUCAUCCCCGGGGCAACUUCUCUAUCUCACGUUAAGCAGAU
........(((.((((..(((((((((..(((((((((((....).)))).)))))).((((((((((((((..(((((.....((((((...((((((((.((....))..((((....))))..)))))))).)))))).......))))).......)))))))))........)))))...))).))))))..))))..)))... ( -73.50)
>291556_ENSDARG00000013575_ZEBRAFISH_1285_1497/1-218
CAAAGCACAUGCAGAUGUUCAAUUAAAAACAGGAGCCCACAGUCUUGGCUCUUGUUUGCACUGGCCCACAUUGUUUGCAGAGCAACAACGGUUGCUAGUGGCAACACCGUCCACUCCCGUUUCCAGGAGCCUGUUGCCAUGCAACCAACAGAAUACUUCCCAAACCAAACAAUGCCAUCCCCCAAGUGAUCCUUCUAUCUCACAUUAACCAGA
....((((((....))))........(((((((((((.........))))))))))))).((((....(((((((((............((((((..(((((((((.......((((........))))..)))))))))))))))....((.....))......)))))))))...........((((..........)))).....)))). ( -58.60)
>291556_SINFRUG00000129155_FUGU_972_1188/1-218
AAAAACACAUACAGAUGUUCAAUUAAAAACAGGAGCCUACAGUCUUGGCUCGUGUUUGAACUGGCCCACAUUGUUUGCAGAGCAACAACCGUUGCUAUGGGCAACACUGUCCACUCCCGUUUCCAGGAGCCUGUUGCUAGGCGACCAACAGAAUACUUCCCAAACCAAACAAUGCCACCCCCCUCCCCACCUGAUCCUCUGUUACACGCUAAGUAGA
..........(((((.(.(((.....(((((.(((((.........))))).)))))....((((....((((((((...((((((....)))))).((((((....))))))((((........)))).((((((...(....)))))))..............))))))))))))..............))).).)))))............... ( -55.40)
>291556_ENSRNOG00000017626_RAT_1660_1867/1-218
AAAACACAUGCAGAUGCUGGAUGGGAACCCGGCGCAGGCCUCUGGCGCCGCGUGCCCAGCCCUCAUUGUUUGCUGAGUAACAACGGUUGCCAGGGGCGACGCAGUCCACUCCCGUUUCCAAGCGCCCGUUGCCCAGCAACCCGUAGAAUACUUCCCAAACCAAACAAUGUCAUCCCCGGGGCAACUUCUCUAUCUCACGUUAAGCAGA
.....((.((.(((((..(((((((.((.((((((((....)).)))))).)).))))((((((((((((((..(((((...((((((((...((((((((.((....))..((((....))))..)))))))).)))).))))....))))).......)))))))))........)))))...)))..))))))).))........ ( -68.60)
>consensus
_AAAACACAUGCAGAUGCUCAAUGAAAAACAGGAGCCGAC_CUC_UGGCGC_CGCGUGC_CCAGCCCACAUUGUUUGCAGAGCAACAACGGUUGCCAGGGGCAACACAGUCCACUCCCGUUUCCAAGAGCCCGUUGCCAAGCAACCAACAGAAUACUUCCCAAACCAAACAAUGCCAUCCCCCGGGCAAC____UCCUCUAUCUCACGUUAAGCAGA_
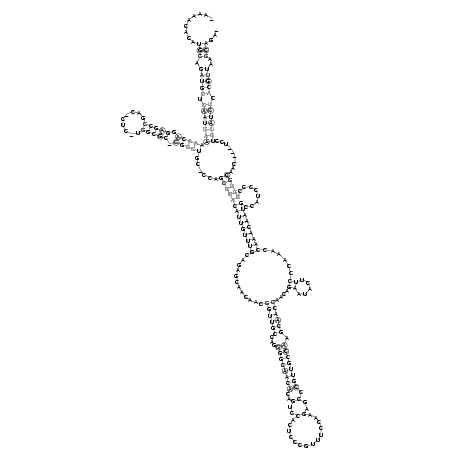
.........(((.(((((.((((((((((((.(((((.........))))).)))))......((((((((((((((............((((((...((((((((..(..(..............)..).)))))))).))))))....((.....))......)))))))))........)))))..........))))))).)))))..)))... (-40.03 = -42.97 + 2.94)
291556-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004