Index >
Results for CNB 268477-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
268477_ENSG00000171532_HUMAN_8460_8581/1-124 CCAGGCGGGGTACCAGCCTCTATGCCAGG-CCATATGGGCTTGGCACGTCACGGGCAGCA
268477_SINFRUG00000148338_FUGU_7947_8058/1-124 ----------TACCAGCTCTGATGCCAGGCCCATAT-GGC-TGGCACGTCATTGGCAAGA
268477_ENSMUSG00000038255_MOUSE_8588_8708/1-124 -CAGGCGGGGTACCAGCCTCTATGCCAGG-CCATATGGGCTTGGCACGTCACGGGCAGCA
268477_ENSRNOG00000028417_RAT_8595_8715/1-124 -CAGGCGGGGTACCAGCCTCTATGCCAGG-CCATATGGGCTTGGCACGTCACGGGCAGCA
268477_ENSDARG00000016854_ZEBRAFISH_8436_8545/1-124 ----------TACCTGCTCTTATGCCAAG-CCATAT-GGT-GGGCACGTCACTGGCAGGA
**** ** ****** * ****** ** ********* **** *
268477_ENSG00000171532_HUMAN_8460_8581/1-124 GCTATCACATGAGAGACGGTGATTGGCATGCGTCTGCACTGGGGGAGAGCCGGCTCCCCC
268477_SINFRUG00000148338_FUGU_7947_8058/1-124 GCCATCACATGAGAGCCGGTGATTGACATGCGGCCGCATTCACAGAGACTGGCTTCCCCC
268477_ENSMUSG00000038255_MOUSE_8588_8708/1-124 GCTATCACATGAGAGACGGTGATTGGCATGCGTCTGCATTGGGGGAGAGCCGGCTCCCCC
268477_ENSRNOG00000028417_RAT_8595_8715/1-124 GCTATCACATGAGAGACGGTGATTGGCATGCGTCTGCATTGGGGGAGAGCCGGCTCCCCC
268477_ENSDARG00000016854_ZEBRAFISH_8436_8545/1-124 GCCATCACATGAGAGCGGGTGATTGACACGCGCTCGCATTCGTAGAGATTGCCTTCCCGA
** ************ ******** ** *** *** * **** ****
268477_ENSG00000171532_HUMAN_8460_8581/1-124 GGG-
268477_SINFRUG00000148338_FUGU_7947_8058/1-124 TGGG
268477_ENSMUSG00000038255_MOUSE_8588_8708/1-124 GGG-
268477_ENSRNOG00000028417_RAT_8595_8715/1-124 GGG-
268477_ENSDARG00000016854_ZEBRAFISH_8436_8545/1-124 GGG-
**
268477-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 124
Mean pairwise identity: 79.11
Mean single sequence MFE: -52.16
Consensus MFE: -30.32
Energy contribution: -31.68
Covariance contribution: 1.36
Mean z-score: -1.90
Structure conservation index: 0.58
SVM decision value: 0.42
SVM RNA-class probability: 0.729641
Prediction: RNA
######################################################################
>268477_ENSG00000171532_HUMAN_8460_8581/1-124
CCAGGCGGGGUACCAGCCUCUAUGCCAGGCCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCACUGGGGGAGAGCCGGCUCCCCCGGG
...((((((((....)))))..(((((((((.....))))))))).))).(((((.(((((((((((.(....)...)))).)))).))))))))..(((((((((......))))))))). ( -61.10)
>268477_SINFRUG00000148338_FUGU_7947_8058/1-124
UACCAGCUCUGAUGCCAGGCCCAUAUGGCUGGCACGUCAUUGGCAAGAGCCAUCACAUGAGAGCCGGUGAUUGACAUGCGGCCGCAUUCACAGAGACUGGCUUCCCCCUGGG
..(((((((((((((..(((((((((.((((((.(.((((((((....))))....))))).)))))).))....))).))))))))...))))).))))...(((...))) ( -43.70)
>268477_ENSMUSG00000038255_MOUSE_8588_8708/1-124
CAGGCGGGGUACCAGCCUCUAUGCCAGGCCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCAUUGGGGGAGAGCCGGCUCCCCCGGG
..((((((((....)))))..(((((((((.....))))))))).))).(((((.(((((((((((.(....)...)))).)))).))))))))..(((((((((......))))))))). ( -58.30)
>268477_ENSRNOG00000028417_RAT_8595_8715/1-124
CAGGCGGGGUACCAGCCUCUAUGCCAGGCCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCAUUGGGGGAGAGCCGGCUCCCCCGGG
..((((((((....)))))..(((((((((.....))))))))).))).(((((.(((((((((((.(....)...)))).)))).))))))))..(((((((((......))))))))). ( -58.30)
>268477_ENSDARG00000016854_ZEBRAFISH_8436_8545/1-124
UACCUGCUCUUAUGCCAAGCCAUAUGGUGGGCACGUCACUGGCAGGAGCCAUCACAUGAGAGCGGGUGAUUGACACGCGCUCGCAUUCGUAGAGAUUGCCUUCCCGAGGG
((((((((((((((((..(((....))).)))..((...((((....))))..)))))))))))))))........((....)).((((..(((.....)))..)))).. ( -39.40)
>consensus
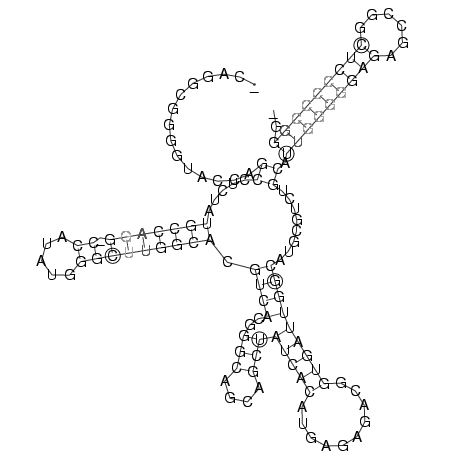
_CAGGCGGGGUACCAGCCUCUAUGCCAGG_CCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCAUUGGGGGAGAGCCGGCUCCCCCGGG_
...............((.....(((((((.((.....))))))))).((((..(((....)))(((((..........)))))))))........)).(((((((((......))))))))).. (-30.32 = -31.68 + 1.36)
268477-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004