Index >
Results for CNB 265692_2
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 TTACAATAAAGCCAAGGGGAGAGAATTAAGAATCTTTGAAGTAGACCAAGGCTCAAAGGA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 TTACAATAAAGCCAAGGGGAGAGAATTAAGAATCTTTGAAGTAGACCAAGGCTCAAAGGA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 TTACAATAAAGCCAAGGGGAGAGAATTAAAAATCTTTGAAGTAGATCAAGGCTCAAAGGA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 G-ACACAAAAGG--AGGAGGGAAAATAAA-AAGCTTTGAAGTGCATTAAAGC-CAAAGAA
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 ATCAG-CCAAGTAGACTTAAAGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGTGAACA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 ATCAG-CCAAGTAGACTTAAAGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGTGAACA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 ATCAG-CCAAGTAGACTTAAAGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGGGAACA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 TGCTGGCCAAGTAGATTTAAAGTAGCCACCGATTTCCAAAACCTTGCTTGAAAGGGAGGA
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 ATAAAAGGAAGTAAA-ATTTATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCG
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 ATAAAAGGAAGTAAA-ATTTATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCG
265692_ENSG00000112238_HUMAN_9283_9748/1-473 ATAAAAGGAAGTAAA-ATTTATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCG
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 ATTGGAGAGGGCGAGGATTTATGGATAAATTATACAGTGGATTTGTCAGTTAAAATATCG
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 TAACTGTCTCAGGGACAATACCCCATGCTGAGGATTAATGGTCCCGCCGGACCTTTGATT
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 TAACTGTCTCAGGGACAATACCCCATGCTGAGGATTAATGGTCCCGCCGGACCTTTGATT
265692_ENSG00000112238_HUMAN_9283_9748/1-473 TAACTGTCTGGAGGACAATACCCCATGCTGAGGATTAATGGTCCCGCCGGACCTTTGATT
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 GAACTGTCTGGAGGACAAAGCCACATGCTGAGGATTAATGGTTGCTCCGGACCTTTGATT
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 CACCAGCGCCTTTTCTTCGCCCTTGACAAATTGGATTTTTAGGAATGGGAAGGTCGCCTG
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 CACCAGCGCCTTTTCTTCGCCCTTGACAAATTGGATTTTTAGGAATGGGAAGGTCGCCTG
265692_ENSG00000112238_HUMAN_9283_9748/1-473 CACCAGTGCCTTTTCTTCGCCCTTGACAAATTGGATTTTTAGGAATGGGAAGGTCGCCTG
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 CCGCACCCTCTTTTCTCTGCCCTTGACAAATTGGATTTTGGGGA-TGGGAAGGTCGCCCG
265692_2.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 87.57
Mean single sequence MFE: -82.38
Consensus MFE: -58.42
Energy contribution: -59.92
Covariance contribution: 1.50
Mean z-score: -1.42
Structure conservation index: 0.71
SVM decision value: -0.05
SVM RNA-class probability: 0.508290
Prediction: RNA
######################################################################
>265692_ENSRNOG00000007683_RAT_9227_9692/1-473
UUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGUGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAGGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGCGCCUUUUCUUCGCCCUUGACAAAUUGGAUUUUUAGGAAUGGGAAGGUCGCCUG
...............(((.(((((........)))))......((((..((((..........))))((((.((((......))))))))..(((((....(..(((((((((((((....((((((...........((((.((.(((.((.(((((.....))))))).))).))(((..((((.....)))).))).))))((((.(((((((.(((((....))))).)))))))..)))).)))))).))))))...)))))))..)((((....))))))))))))).))). ( -81.40)
>265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473
UUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGUGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAGGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGCGCCUUUUCUUCGCCCUUGACAAAUUGGAUUUUUAGGAAUGGGAAGGUCGCCUG
...............(((.(((((........)))))......((((..((((..........))))((((.((((......))))))))..(((((....(..(((((((((((((....((((((...........((((.((.(((.((.(((((.....))))))).))).))(((..((((.....)))).))).))))((((.(((((((.(((((....))))).)))))))..)))).)))))).))))))...)))))))..)((((....))))))))))))).))). ( -81.40)
>265692_ENSG00000112238_HUMAN_9283_9748/1-473
UUACAAUAAAGCCAAGGGGAGAGAAUUAAAAAUCUUUGAAGUAGAUCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGGGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUGGAGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGUGCCUUUUCUUCGCCCUUGACAAAUUGGAUUUUUAGGAAUGGGAAGGUCGCCUG
..............(((.((............(((((((.((........)))))))))......((((((.((((......))))))))..(((((....(..(((((((((((......((((((..(((.(((((......))))).))).((((..(((((.(((...(((.......)))...))))))))....))))((((.(((((((.(((((....))))).)))))))..)))).))))))......)))))))))))..)((((....))))))))))))).))). ( -78.80)
>265692_SINFRUG00000152747_FUGU_5223_5688/1-473
GACACAAAAGGAGGAGGGAAAAUAAAAAGCUUUGAAGUGCAUUAAAGCCAAAGAAUGCUGGCCAAGUAGAUUUAAAGUAGCCACCGAUUUCCAAAACCUUGCUUGAAAGGGAGGAAUUGGAGAGGGCGAGGAUUUAUGGAUAAAUUAUACAGUGGAUUUGUCAGUUAAAAUAUCGGAACUGUCUGGAGGACAAAGCCACAUGCUGAGGAUUAAUGGUUGCUCCGGACCUUUGAUUCCGCACCCUCUUUUCUCUGCCCUUGACAAAUUGGAUUUUGGGGAUGGGAAGGUCGCCCG
...............(((..........((((((((.(((......((((........))))...)))..)))))))).(((.((.((..((((((((....(((.(((((.(((...((((((((.(.(((((((..((((((((........))))))))...))))..((((((...((((((((.....(((((....(....).....))))).)))))))).))))))))).).))))))))..))).)))))..)))...)).))))))..)).))..)))..))). ( -87.90)
>consensus
UUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAG_CCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGGGAACAAUAAAAGGAAGUAAA_AUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAAGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGCGCCUUUUCUUCGCCCUUGACAAAUUGGAUUUUUAGGAAUGGGAAGGUCGCCUG
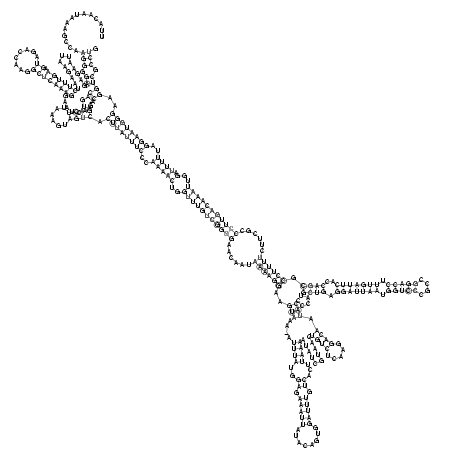
..............(((.((.............((((((.((........))))))))........((.....((((......)))).((((((((..(((((..(((((((((((......((((((..(((....((((..((.(((((........))))).))...))))..........((((.....)))).))).....((((.(((((((.(((((....))))).)))))))..)))).))))))......)))))))))))..).))))..))))))))..)))).))). (-58.42 = -59.92 + 1.50)
265692_2.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004