Index >
Results for CNB 265692_1
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 AGAAGTTAACAAGTCCTCCAAACAGTCCCCGTCCCTTTGTTTAATCTCCTTTACAATAAA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 AGAAGTTAACAAGTCCTCCAAACAGTCCCCGTCCCTTTGTTTAATCTCCTTTACAATAAA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 AGTAGTTAACAAGTCCTCCAAACAGTCTCCATCTCTTTGTTTAATCTCCTTTACAATAAA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 AGAAGTTAACAAGTCCTCTAAACTTTCCTGCCTCCTTTGTTGCA-CTAAGG-ACACAAAA
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 GCCAAGGGGAGAGAATTAAGAATCTTTGAAGTAGACCAAGGCTCAAAGGAATCAG-CCAA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 GCCAAGGGGAGAGAATTAAGAATCTTTGAAGTAGACCAAGGCTCAAAGGAATCAG-CCAA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 GCCAAGGGGAGAGAATTAAAAATCTTTGAAGTAGATCAAGGCTCAAAGGAATCAG-CCAA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 GG--AGGAGGGAAAATAAA-AAGCTTTGAAGTGCATTAAAGC-CAAAGAATGCTGGCCAA
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 GTAGACTTAAAGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGTGAACAATAAAAGGAA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 GTAGACTTAAAGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGTGAACAATAAAAGGAA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 GTAGACTTAAAGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGGGAACAATAAAAGGAA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 GTAGATTTAAAGTAGCCACCGATTTCCAAAACCTTGCTTGAAAGGGAGGAATTGGAGAGG
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 GTAAA-ATTTATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCGTAACTGTCTC
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 GTAAA-ATTTATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCGTAACTGTCTC
265692_ENSG00000112238_HUMAN_9283_9748/1-473 GTAAA-ATTTATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCGTAACTGTCTG
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 GCGAGGATTTATGGATAAATTATACAGTGGATTTGTCAGTTAAAATATCGGAACTGTCTG
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 AGGGACAATACCCCATGCTGAGGATTAATGGTCCCGCCGGACCTTTGATTCACCAGCGCC
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 AGGGACAATACCCCATGCTGAGGATTAATGGTCCCGCCGGACCTTTGATTCACCAGCGCC
265692_ENSG00000112238_HUMAN_9283_9748/1-473 GAGGACAATACCCCATGCTGAGGATTAATGGTCCCGCCGGACCTTTGATTCACCAGTGCC
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 GAGGACAAAGCCACATGCTGAGGATTAATGGTTGCTCCGGACCTTTGATTCCGCACCCTC
265692_1.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 85.62
Mean single sequence MFE: -79.72
Consensus MFE: -56.51
Energy contribution: -59.45
Covariance contribution: 2.94
Mean z-score: -1.88
Structure conservation index: 0.71
SVM decision value: 0.52
SVM RNA-class probability: 0.768555
Prediction: RNA
######################################################################
>265692_ENSRNOG00000007683_RAT_9227_9692/1-473
AGAAGUUAACAAGUCCUCCAAACAGUCCCCGUCCCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGUGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAGGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGCGCC
..((((..(((((((((((...((((((((....((((((((((......))).)))))))....)))))(.(((.((((..(((((((.((........))))))))).............((((......)))).)))).))).)....)))((((......)))).......))).(((.(((((......))))).)))...)))))))).))))....................(((.......)))..((((.(((((((.(((((....))))).)))))))..))))... ( -74.10)
>265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473
AGAAGUUAACAAGUCCUCCAAACAGUCCCCGUCCCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGUGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAGGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGCGCC
..((((..(((((((((((...((((((((....((((((((((......))).)))))))....)))))(.(((.((((..(((((((.((........))))))))).............((((......)))).)))).))).)....)))((((......)))).......))).(((.(((((......))))).)))...)))))))).))))....................(((.......)))..((((.(((((((.(((((....))))).)))))))..))))... ( -74.10)
>265692_ENSG00000112238_HUMAN_9283_9748/1-473
AGUAGUUAACAAGUCCUCCAAACAGUCUCCAUCUCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAAAAUCUUUGAAGUAGAUCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGGGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUGGAGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGUGCC
............((((((((.(((((((((((..((((.(((.((.((((..((((....((((..((((((..........(((((((.((........)))))))))........((((.((((......)))))))).)))))).....))))))))..))))...)).))).))))........)))))))........(((((.....))))).............)))).))))))))..........((((.(((((((.(((((....))))).)))))))..))))... ( -82.60)
>265692_SINFRUG00000152747_FUGU_5223_5688/1-473
AGAAGUUAACAAGUCCUCUAAACUUUCCUGCCUCCUUUGUUGCACUAAGGACACAAAAGGAGGAGGGAAAAUAAAAAGCUUUGAAGUGCAUUAAAGCCAAAGAAUGCUGGCCAAGUAGAUUUAAAGUAGCCACCGAUUUCCAAAACCUUGCUUGAAAGGGAGGAAUUGGAGAGGGCGAGGAUUUAUGGAUAAAUUAUACAGUGGAUUUGUCAGUUAAAAUAUCGGAACUGUCUGGAGGACAAAGCCACAUGCUGAGGAUUAAUGGUUGCUCCGGACCUUUGAUUCCGCACCCUC
.....(((((((((((((.....((((((.((((((((..(((......).))..)))))))).)))))).......((((((((.(((......((((........))))...)))..)))))))).(((.....(((((((..((((.((....)).))))..))))))).)))))))))))...((((((((........)))))))).))))).....((((((.((((((((.....(((((....(....).....))))).))))))))....).)))))....... ( -88.10)
>consensus
AGAAGUUAACAAGUCCUCCAAACAGUCCCCGUCCCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAG_CCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGGGAACAAUAAAAGGAAGUAAA_AUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAAGGACAAUACCCCAUGCUGAGGAUUAAUGGUCCCGCCGGACCUUUGAUUCACCAGCGCC
............((((((((.(((((..(..(((.((((((...(((((...((((...((.....((((((...........((((((.((........))))))))..........((((.((((......)))))))).))))))....))...)))).)))))..)))))).)))........((((..((.(((((........))))).))...)))).....)..))))).))))))))..........((((.(((((((.(((((....))))).)))))))..))))... (-56.51 = -59.45 + 2.94)
265692_1.rnaz
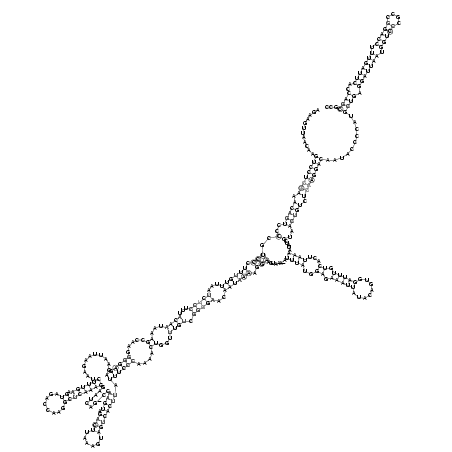
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004