Index >
Results for CNB 264053
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
264053_ENSG00000185045_HUMAN_8824_8931/1-109 -CCTCAGTTTCACTGCCATGGTTTCCACTCCTGAGAACCACCAGACTTTCATCAACTCAG
264053_ENSMUSG00000043873_MOUSE_5451_5559/1-109 CCCTCAGGTTCAGTGCCATGGTCTCCACTCCACACAACCAACAGACTTTCATTAACTCAG
264053_ENSRNOG00000016972_RAT_9893_10000/1-109 CCCCTAGTTTCACCACCATGGTTTCCACTTCTCAGAACCGCCAGACCTTTATTACCTCAG
264053_SINFRUG00000153198_FUGU_1686_1791/1-109 CC---AGATTCACCACAATGGTGTCAACACAAACCAGCAGAAGCAAATTCATCCAGTCCT
* ** **** * ***** ** ** * * * ** ** **
264053_ENSG00000185045_HUMAN_8824_8931/1-109 TCATCAAATTCCTGCGCCAGTATGAGTTTGACGGGCTGGACTTTGACTG
264053_ENSMUSG00000043873_MOUSE_5451_5559/1-109 CCATCAAATTCCTGCGCCAGTATGGGTTTGATGGACTGAACCTGGACTG
264053_ENSRNOG00000016972_RAT_9893_10000/1-109 TCATCAAATTCCTGCGACAGTATGGGTTTGATGGACTGGACCTGGACT-
264053_SINFRUG00000153198_FUGU_1686_1791/1-109 CCATCAAACAGCTGAGAAAATACGGATTTGATGGCCTGGATCTGGACTG
******* *** * * ** * ***** ** *** * * ****
//
264053.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 109
Mean pairwise identity: 72.63
Mean single sequence MFE: -29.50
Consensus MFE: -18.00
Energy contribution: -20.25
Covariance contribution: 2.25
Mean z-score: -2.36
Structure conservation index: 0.61
SVM decision value: 2.78
SVM RNA-class probability: 0.996975
Prediction: RNA
######################################################################
>264053_ENSG00000185045_HUMAN_8824_8931/1-109
CCUCAGUUUCACUGCCAUGGUUUCCACUCCUGAGAACCACCAGACUUUCAUCAACUCAGUCAUCAAAUUCCUGCGCCAGUAUGAGUUUGACGGGCUGGACUUUGACUG
...(((((...(((...(((((..((....))..))))).)))............((((((.((((((((.(((....))).))))))))..)))))).....))))) ( -29.00)
>264053_ENSMUSG00000043873_MOUSE_5451_5559/1-109
CCCUCAGGUUCAGUGCCAUGGUCUCCACUCCACACAACCAACAGACUUUCAUUAACUCAGCCAUCAAAUUCCUGCGCCAGUAUGGGUUUGAUGGACUGAACCUGGACUG
...(((((((((((....(((........)))............................(((((((((((.(((....))).)))))))))))))))))))))).... ( -35.40)
>264053_ENSRNOG00000016972_RAT_9893_10000/1-109
CCCCUAGUUUCACCACCAUGGUUUCCACUUCUCAGAACCGCCAGACCUUUAUUACCUCAGUCAUCAAAUUCCUGCGACAGUAUGGGUUUGAUGGACUGGACCUGGACU
...((((............(((((..........))))).................(((((((((((((((.(((....))).))))))))).))))))..))))... ( -24.80)
>264053_SINFRUG00000153198_FUGU_1686_1791/1-109
CCAGAUUCACCACAAUGGUGUCAACACAAACCAGCAGAAGCAAAUUCAUCCAGUCCUCCAUCAAACAGCUGAGAAAAUACGGAUUUGAUGGCCUGGAUCUGGACUG
.(((...((((.....))))..........((((..(((.....)))((((((....((((((((...(((........))).)))))))).)))))))))).))) ( -28.80)
>consensus
CCCUCAGUUUCACCACCAUGGUUUCCACUCCACACAACCAACAGACUUUCAUCAACUCAGCCAUCAAAUUCCUGCGACAGUAUGGGUUUGAUGGACUGGACCUGGACUG
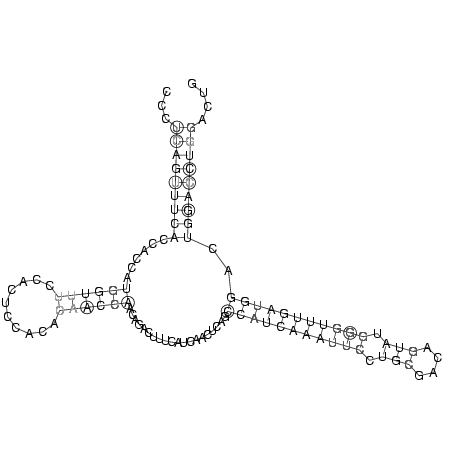
...(((((((((......((((((..........))))))....................(((((((((((.(((....))).)))))))))))..))))))))).... (-18.00 = -20.25 + 2.25)
264053.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004