Index >
Results for CNB 251196-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
251196_ENSG00000082898_HUMAN_5544_5647/1-104 CCTCCTCCTAGTGCCTCAAACTCGGAACCGGTTGAAACCGGTTCAGACTGGGAGATTCCA
251196_ENSMUSG00000020290_MOUSE_3674_3777/1-104 CCTCCTCCTAGTGCCTCAAACTCGGAACCGGTTGAAACCGGTTCAGACTGGGAGATTCCA
251196_ENSDARG00000005555_ZEBRAFISH_6682_6782/1-104 -CTTCTCACA-AGCAACAAACTCGTAACCGGTTGAAACCGGTTCAGACTGGGAGATTCCA
251196_ENSRNOG00000009935_RAT_4112_4214/1-104 CCTCCTCCTAGTGCCTCAAACTCGGAACCGGTTGAAACCGGTTCAGACTGGGAGATTCCA
** *** * ** ******** ***********************************
251196_ENSG00000082898_HUMAN_5544_5647/1-104 TCTCGGTTGAGTTTTCAGCCCATGGCGCCGCGGAGCGTTTGGAA
251196_ENSMUSG00000020290_MOUSE_3674_3777/1-104 TCTCGGTTGAGTTTTCAGCCCATGGCGCCGCGGAGCGTTTGGAA
251196_ENSDARG00000005555_ZEBRAFISH_6682_6782/1-104 TCTCGGTTTAGTTTTCAGCCGATGGCGCCGCGAAAAGTTTGGA-
251196_ENSRNOG00000009935_RAT_4112_4214/1-104 TCTCGGTTGAGTTTTCAGCCCATGGCGCCGCGGAGCGTTTGGA-
******** *********** *********** * *******
251196-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 104
Mean pairwise identity: 92.62
Mean single sequence MFE: -37.43
Consensus MFE: -31.35
Energy contribution: -31.72
Covariance contribution: 0.37
Mean z-score: -1.71
Structure conservation index: 0.84
SVM decision value: 0.41
SVM RNA-class probability: 0.724313
Prediction: RNA
######################################################################
>251196_ENSG00000082898_HUMAN_5544_5647/1-104
CCUCCUCCUAGUGCCUCAAACUCGGAACCGGUUGAAACCGGUUCAGACUGGGAGAUUCCAUCUCGGUUGAGUUUUCAGCCCAUGGCGCCGCGGAGCGUUUGGAA
((.(((((..(((((..((((((.((((((((....)))))))).((((((((.......)))))))))))))).........)))))...)))).)...)).. ( -40.50)
>251196_ENSMUSG00000020290_MOUSE_3674_3777/1-104
CCUCCUCCUAGUGCCUCAAACUCGGAACCGGUUGAAACCGGUUCAGACUGGGAGAUUCCAUCUCGGUUGAGUUUUCAGCCCAUGGCGCCGCGGAGCGUUUGGAA
((.(((((..(((((..((((((.((((((((....)))))))).((((((((.......)))))))))))))).........)))))...)))).)...)).. ( -40.50)
>251196_ENSDARG00000005555_ZEBRAFISH_6682_6782/1-104
CUUCUCACAAGCAACAAACUCGUAACCGGUUGAAACCGGUUCAGACUGGGAGAUUCCAUCUCGGUUUAGUUUUCAGCCGAUGGCGCCGCGAAAAGUUUGGA
.......(((((...(((((.(.(((((((....))))))))(((((((((.......))))))))))))))...(((...)))..........))))).. ( -28.20)
>251196_ENSRNOG00000009935_RAT_4112_4214/1-104
CCUCCUCCUAGUGCCUCAAACUCGGAACCGGUUGAAACCGGUUCAGACUGGGAGAUUCCAUCUCGGUUGAGUUUUCAGCCCAUGGCGCCGCGGAGCGUUUGGA
((.(((((..(((((..((((((.((((((((....)))))))).((((((((.......)))))))))))))).........)))))...)))).)...)). ( -40.50)
>consensus
CCUCCUCCUAGUGCCUCAAACUCGGAACCGGUUGAAACCGGUUCAGACUGGGAGAUUCCAUCUCGGUUGAGUUUUCAGCCCAUGGCGCCGCGGAGCGUUUGGA_
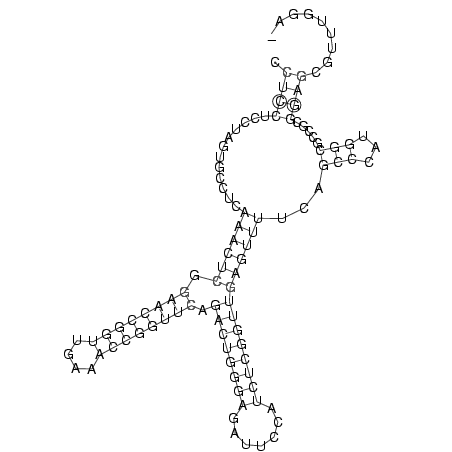
.((((............((((((.((((((((....)))))))).((((((((.......))))))))))))))...(((...))).....))))......... (-31.35 = -31.72 + 0.37)
251196-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004