Index >
Results for CNB 244427_5
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 TGACCAGGGTTTTATTCACAGGCCTGTGATGAACGCCAAGCTGATCAGGCGGAATGAAGA
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 TGACCAGGGTTTTATTCACAGGCCTGTGATGAACGCCAAGCTGATCAGGCGGAATGAAGA
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 TGACC-GGCTTTTATTCACTCGCTAGTGATGAACGCCAAGCTGATCAGGCGGAATGGGCG
244427_ENSG00000163064_HUMAN_6937_7444/1-510 TGACCAGGGTTTTATTCACAGGCCTGTGATGAACGCCAAGCTGATCAGGCGGAATGAAGA
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 GTAATTAAAACCTATAAATTATCTTCCGCAGCCCTTCTCACGGCGTCTCCTGCAGGCGAG
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 GTAATTAAAACCTATAAATTATCTTCCGCAGCCCTTCTCACGGCGTCTCCTGCAGGCGAG
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 CTAATTAAAACCTATAAATTATCTTTTGCAGTCCCTCTCAAGACGTCGCATGCAGCCGAG
244427_ENSG00000163064_HUMAN_6937_7444/1-510 GTAATTAAAACCTATAAATTATCTTCCGCAGCCCTTCTCGAGGCGTCTCCTGCAGGCGAG
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 ATAAGGCGTCGAGACCCTATTTACGGCGCTGAAAGGGACCGCGGCGCACAAAAGCTACGC
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 ATAAGGCGTCGAGACCCTATTTACGGCGCTGAAAGGGACCGCGGTGCACAAAAGCTACGC
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 ATAAGAGCTGGAGTGCTAATTTACCGC-CTGAAAAGGAGCGTGGCTCACAATGGCTACTT
244427_ENSG00000163064_HUMAN_6937_7444/1-510 ATAAGGCGTCGAGACCCTATTTACCGCGCTGAAAGGGACCGCGGCGCACAAAAGCTACGC
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 GGCT-AAATAGGATATTGATAGTGTCCTAATTAGAATTTAATTAAGTACCCACGGTCGCT
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 GGCT-AAATAGGATATTGATAGTGTCCTAATTAGAATTTAATTAAGTACCCACGGTCGCT
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 GGCTCAAATAGCATATTGATAGTGTCCTAATTAGAAATTTATTAAGTACCCACTCGAGCT
244427_ENSG00000163064_HUMAN_6937_7444/1-510 AGCT-AAATAGGATATTGATAGTGTCCTAATTAGAATTTAATTAAGTACCCACGCTTGCT
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 TGTGGGATAAAGATTTCAAT
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 TGCGGGATAAAGATTTCAAT
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 TTTCGAATAAAGATTTCAA-
244427_ENSG00000163064_HUMAN_6937_7444/1-510 TGCGGGATAAAGATTTCAAT
244427_5.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 260
Mean pairwise identity: 89.21
Mean single sequence MFE: -79.48
Consensus MFE: -55.84
Energy contribution: -59.53
Covariance contribution: 3.69
Mean z-score: -1.75
Structure conservation index: 0.70
SVM decision value: 0.11
SVM RNA-class probability: 0.587784
Prediction: RNA
######################################################################
>244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510
UGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCACGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACGGCGCUGAAAGGGACCGCGGCGCACAAAAGCUACGCGGCUAAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGGUCGCUUGUGGGAUAAAGAUUUCAAU
.(((..((((((...(((((....))))).)))).))..((((....(((.(...(((((.(((((..........))))))))))..).))).......)))))))(((..(((((((.....((((((((((.....))).)))))))....(((.(((((..((......))..)))))((((.((((((((.....)))))))).))))..............)))....)))))))..)))............. ( -84.00)
>244427_ENSRNOG00000002457_RAT_6069_6575/1-510
UGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCACGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACGGCGCUGAAAGGGACCGCGGUGCACAAAAGCUACGCGGCUAAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGGUCGCUUGCGGGAUAAAGAUUUCAAU
.(((..((((((...(((((....))))).)))).))..((((....(((.(...(((((.(((((..........))))))))))..).))).......)))))))((((((((((((.....((((((((((.....))).)))))))....(((.(((((..((......))..)))))((((.((((((((.....)))))))).))))..............)))....))))))))))))............. ( -86.40)
>244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510
UGACCGGCUUUUAUUCACUCGCUAGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGGGCGCUAAUUAAAACCUAUAAAUUAUCUUUUGCAGUCCCUCUCAAGACGUCGCAUGCAGCCGAGAUAAGAGCUGGAGUGCUAAUUUACCGCCUGAAAAGGAGCGUGGCUCACAAUGGCUACUUGGCUCAAAUAGCAUAUUGAUAGUGUCCUAAUUAGAAAUUUAUUAAGUACCCACUCGAGCUUUUCGAAUAAAGAUUUCAA
.(((((((.(((((.((((....))))))))).)))..((((.((((((((((..((((((........((......((((((((((((((..(((....)).)..)).)))))..))))))).....))))))))...)).))))))))....((((((((((......))))))...))))...)))).........).))).......(((((((.....((....))(((((...)))))....))))))).. ( -70.00)
>244427_ENSG00000163064_HUMAN_6937_7444/1-510
UGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCGAGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACCGCGCUGAAAGGGACCGCGGCGCACAAAAGCUACGCAGCUAAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGCUUGCUUGCGGGAUAAAGAUUUCAAU
.(((....((((...(((((....))))).))))(((..(..((...(((.(...(((((.(((((..........))))))))))..).)))..)).)..))))))(((((((((((((....((((.((.............))))))....(((((.(((..((......))..)))..((((.((((((((.....)))))))).))))...........)).)))...)))))))))))))............. ( -77.52)
>consensus
UGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCAAGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACCGCGCUGAAAGGGACCGCGGCGCACAAAAGCUACGCGGCU_AAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGCUCGCUUGCGGGAUAAAGAUUUCAAU
.(((..((((((((.(((((....)))))....((((..........))))..............)))))))).................(((........))))))(((((((((((((....(((((((............)))))))....(((.(((((..((......))..)))))......((((((((.....))))))))...................)))...)))))))))))))............. (-55.84 = -59.53 + 3.69)
244427_5.rnaz
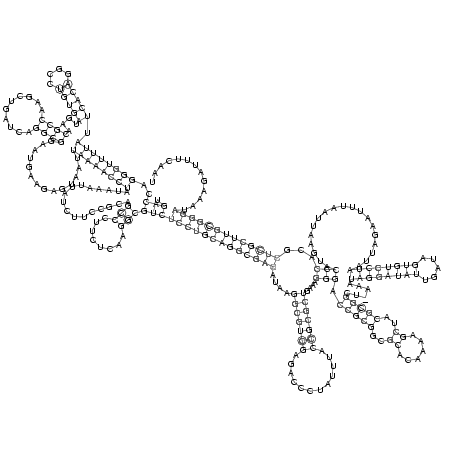
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004