Index >
Results for CNB 244427_4
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 NNNNN-TCACTTTGATTAAACGTCTTGCCAGTGCTATGTGACAGAGCTTTTGACCAGGGT
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 NNNNN-TCACTTTGATTAAACGTCTTGCCAGCGCTATGTGACAGAGCTTTTGACCAGGGT
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 CCCTGCTTACCTTGATAAAGCGCCCTGCCAGCGGGTAGTGACAGTGCTTTTGACC-GGCT
244427_ENSG00000163064_HUMAN_6937_7444/1-510 NNNNNNTCACTTTGATTAAGCGTCTTGCCAGCGCGATGTGACAGAGCTTTTGACCAGGGT
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 TTTATTCACAGGCCTGTGATGAACGCCAAGCTGATCAGGCGGAATGAAGAGTAATTAAAA
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 TTTATTCACAGGCCTGTGATGAACGCCAAGCTGATCAGGCGGAATGAAGAGTAATTAAAA
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 TTTATTCACTCGCTAGTGATGAACGCCAAGCTGATCAGGCGGAATGGGCGCTAATTAAAA
244427_ENSG00000163064_HUMAN_6937_7444/1-510 TTTATTCACAGGCCTGTGATGAACGCCAAGCTGATCAGGCGGAATGAAGAGTAATTAAAA
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 CCTATAAATTATCTTCCGCAGCCCTTCTCACGGCGTCTCCTGCAGGCGAGATAAGGCGTC
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 CCTATAAATTATCTTCCGCAGCCCTTCTCACGGCGTCTCCTGCAGGCGAGATAAGGCGTC
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 CCTATAAATTATCTTTTGCAGTCCCTCTCAAGACGTCGCATGCAGCCGAGATAAGAGCTG
244427_ENSG00000163064_HUMAN_6937_7444/1-510 CCTATAAATTATCTTCCGCAGCCCTTCTCGAGGCGTCTCCTGCAGGCGAGATAAGGCGTC
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 GAGACCCTATTTACGGCGCTGAAAGGGACCGCGGCGCACAAAAGCTACGCGGCT-AAATA
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 GAGACCCTATTTACGGCGCTGAAAGGGACCGCGGTGCACAAAAGCTACGCGGCT-AAATA
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 GAGTGCTAATTTACCGC-CTGAAAAGGAGCGTGGCTCACAATGGCTACTTGGCTCAAATA
244427_ENSG00000163064_HUMAN_6937_7444/1-510 GAGACCCTATTTACCGCGCTGAAAGGGACCGCGGCGCACAAAAGCTACGCAGCT-AAATA
244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510 GGATATTGATAGTGTCCTAATTAGAATTTAATTAAGTACCCACGGTCGCTTGTGGGATAA
244427_ENSRNOG00000002457_RAT_6069_6575/1-510 GGATATTGATAGTGTCCTAATTAGAATTTAATTAAGTACCCACGGTCGCTTGCGGGATAA
244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510 GCATATTGATAGTGTCCTAATTAGAAATTTATTAAGTACCCACTCGAGCTTTTCGAATAA
244427_ENSG00000163064_HUMAN_6937_7444/1-510 GGATATTGATAGTGTCCTAATTAGAATTTAATTAAGTACCCACGCTTGCTTGCGGGATAA
244427_4.aln
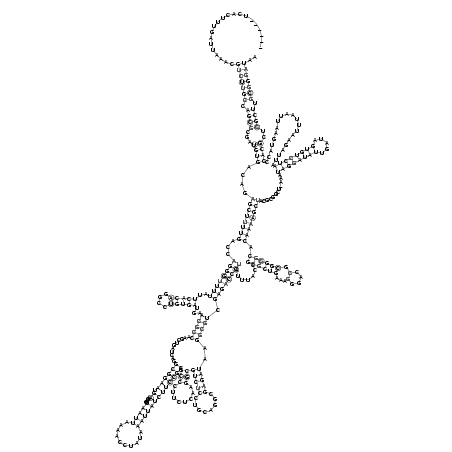
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 87.58
Mean single sequence MFE: -92.08
Consensus MFE: -67.43
Energy contribution: -68.17
Covariance contribution: 0.75
Mean z-score: -1.44
Structure conservation index: 0.73
SVM decision value: 0.00
SVM RNA-class probability: 0.534426
Prediction: RNA
######################################################################
>244427_ENSMUSG00000036888_MOUSE_6983_7489/1-510
NNNNNUCACUUUGAUUAAACGUCUUGCCAGUGCUAUGUGACAGAGCUUUUGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCACGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACGGCGCUGAAAGGGACCGCGGCGCACAAAAGCUACGCGGCUAAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGGUCGCUUGUGGGAUAA
...................((((....(((.(((.(((((.((((((((.....))))))))..))))))))))).)))).(((((.....((...(((.(...(((((.(((((..........))))))))))..).)))..))....))))).(((..(((((((.....((((((((((.....))).)))))))....(((.(((((..((......))..)))))((((.((((((((.....)))))))).))))..............)))....)))))))..)))... ( -93.30)
>244427_ENSRNOG00000002457_RAT_6069_6575/1-510
NNNNNUCACUUUGAUUAAACGUCUUGCCAGCGCUAUGUGACAGAGCUUUUGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCACGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACGGCGCUGAAAGGGACCGCGGUGCACAAAAGCUACGCGGCUAAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGGUCGCUUGCGGGAUAA
...................((((....(((.(((.(((((.((((((((.....))))))))..))))))))))).)))).(((((.....((...(((.(...(((((.(((((..........))))))))))..).)))..))....))))).((((((((((((.....((((((((((.....))).)))))))....(((.(((((..((......))..)))))((((.((((((((.....)))))))).))))..............)))....))))))))))))... ( -95.70)
>244427_ENSDARG00000015073_ZEBRAFISH_4427_4928/1-510
CCCUGCUUACCUUGAUAAAGCGCCCUGCCAGCGGGUAGUGACAGUGCUUUUGACCGGCUUUUAUUCACUCGCUAGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGGGCGCUAAUUAAAACCUAUAAAUUAUCUUUUGCAGUCCCUCUCAAGACGUCGCAUGCAGCCGAGAUAAGAGCUGGAGUGCUAAUUUACCGCCUGAAAAGGAGCGUGGCUCACAAUGGCUACUUGGCUCAAAUAGCAUAUUGAUAGUGUCCUAAUUAGAAAUUUAUUAAGUACCCACUCGAGCUUUUCGAAUAA
...........((((.(((((((.......))(((((......(((((((((((((((.(((((.((((....))))))))).)))..(((..((((((((((..((((((........((......((((((((((((((..(((....)).)..)).)))))..))))))).....))))))))...)).)))))))).....)))((((((......))))))..)).))))).))))).(((((((..((.......))...))))))).)))))......))))))))).... ( -84.30)
>244427_ENSG00000163064_HUMAN_6937_7444/1-510
NNNNNNUCACUUUGAUUAAGCGUCUUGCCAGCGCGAUGUGACAGAGCUUUUGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCGAGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACCGCGCUGAAAGGGACCGCGGCGCACAAAAGCUACGCAGCUAAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGCUUGCUUGCGGGAUAA
.....................(((((((.((((((.((((....((((((((...((((((((..(((((....))))).((.((((..........(((.(...(((((.(((((..........))))))))))..).)))..(((((..(((.....)))...)))))...)))))))))))))).......((((((...(....).)))))).)))))))).)))).((((.((((((((.....)))))))).))))..................)))..))).))))))).. ( -95.00)
>consensus
______UCACUUUGAUUAAACGUCUUGCCAGCGCGAUGUGACAGAGCUUUUGACCAGGGUUUUAUUCACAGGCCUGUGAUGAACGCCAAGCUGAUCAGGCGGAAUGAAGAGUAAUUAAAACCUAUAAAUUAUCUUCCGCAGCCCUUCUCAAGGCGUCUCCUGCAGGCGAGAUAAGGCGUCGAGACCCUAUUUACCGCGCUGAAAGGGACCGCGGCGCACAAAAGCUACGCGGCU_AAAUAGGAUAUUGAUAGUGUCCUAAUUAGAAUUUAAUUAAGUACCCACGCUCGCUUGCGGGAUAA
.....................(((((((.((((...((((....((((((((...((((((((..(((((....)))))...(((((...........((((((.((....(((((..........))))))))))))).(((........)))(((((........)))))..))))).)))))))).......((((((...(....).)))))).))))))))............((((((((.....)))))))).....................))))..)))).))))))).. (-67.43 = -68.17 + 0.75)
244427_4.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004