Index >
Results for CNB 238158
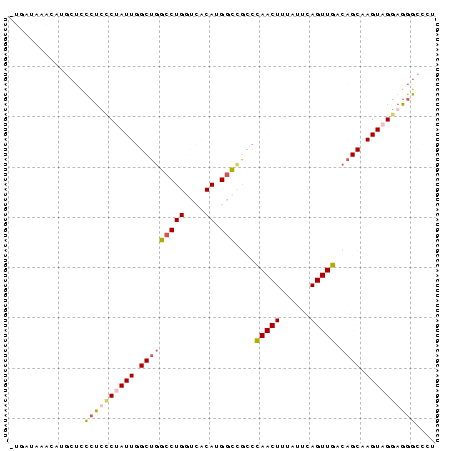
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
238158_ENSG00000170166_HUMAN_9856_9940/1-85 GTGATAAACTTGCTCCCTCGCCATTGGCTGGCCTGGTCACATGGCTGCCCAACTTTATTC
238158_ENSRNOG00000001578_RAT_9852_9936/1-85 GTGATAAACATGCTCTCTTCCTATTGGCTGGCCTGGTCACATGGCCGCCCAACTTTATTC
238158_ENSDARG00000010540_ZEBRAFISH_7426_7509/1-85 -TGATGAAGTCCCTCCACCTCGATTGGCCAAGCTGGTCACATGGTAGGCTAACTTTATTC
238158_ENSMUSG00000042464_MOUSE_9858_9940/1-85 -TGATAAACATGCTTTCTTCCTATTGGCTGGCCTGGTCACATGGCCGCCCAACTTTATTC
**** ** ** * ****** ************ * * **********
238158_ENSG00000170166_HUMAN_9856_9940/1-85 AGTTGACAGCAAGTAGGAGGGCCCT
238158_ENSRNOG00000001578_RAT_9852_9936/1-85 AGTTGACAGCAAGTAGGAGGGCCCA
238158_ENSDARG00000010540_ZEBRAFISH_7426_7509/1-85 AGTTGACAGCAAGTAGGAGGGCTTT
238158_ENSMUSG00000042464_MOUSE_9858_9940/1-85 AGTTGACAGCAAGTAGGAGGGCCC-
**********************
//
238158.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 85
Mean pairwise identity: 83.50
Mean single sequence MFE: -27.33
Consensus MFE: -17.67
Energy contribution: -18.80
Covariance contribution: 1.13
Mean z-score: -1.81
Structure conservation index: 0.65
SVM decision value: 0.25
SVM RNA-class probability: 0.654797
Prediction: RNA
######################################################################
>238158_ENSG00000170166_HUMAN_9856_9940/1-85
GUGAUAAACUUGCUCCCUCGCCAUUGGCUGGCCUGGUCACAUGGCUGCCCAACUUUAUUCAGUUGACAGCAAGUAGGAGGGCCCU
..............(((((((((((((((.....))))).)))))(((.(((((......)))))...))).....))))).... ( -25.70)
>238158_ENSRNOG00000001578_RAT_9852_9936/1-85
GUGAUAAACAUGCUCUCUUCCUAUUGGCUGGCCUGGUCACAUGGCCGCCCAACUUUAUUCAGUUGACAGCAAGUAGGAGGGCCCA
...........((((((((.((...(((.((((((....)).)))))))(((((......)))))......)).))))))))... ( -28.60)
>238158_ENSDARG00000010540_ZEBRAFISH_7426_7509/1-85
UGAUGAAGUCCCUCCACCUCGAUUGGCCAAGCUGGUCACAUGGUAGGCUAACUUUAUUCAGUUGACAGCAAGUAGGAGGGCUUU
....((((.(((((((((..(((..((...))..)))....)))..(((((((......))))...))).....)))))))))) ( -26.90)
>238158_ENSMUSG00000042464_MOUSE_9858_9940/1-85
UGAUAAACAUGCUUUCUUCCUAUUGGCUGGCCUGGUCACAUGGCCGCCCAACUUUAUUCAGUUGACAGCAAGUAGGAGGGCCC
..............((((((((((.(((((((((....)).))))...(((((......)))))..))).))))))))))... ( -28.10)
>consensus
_UGAUAAACAUGCUCCCUCCCUAUUGGCUGGCCUGGUCACAUGGCCGCCCAACUUUAUUCAGUUGACAGCAAGUAGGAGGGCCCU
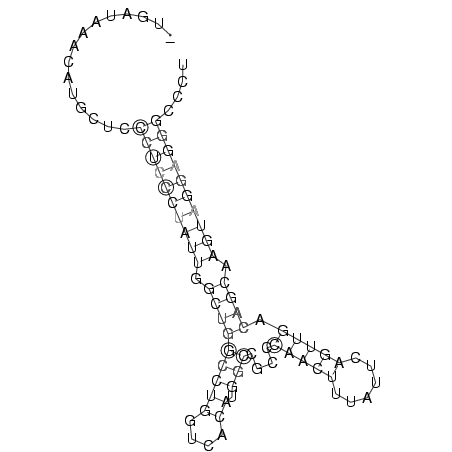
...............((((((((((.(((((((((....)).)))....(((((......))))).)))).)))))))))).... (-17.67 = -18.80 + 1.13)
238158.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004