Index >
Results for CNB 226470-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
226470_ENSG00000182742_HUMAN_8379_8474/1-100 AGTGTTTATGTCAACTACATATTCCCCTAGATACGAATTTGTGA---CCACAAAATTCCT
226470_ENSMUSG00000038692_MOUSE_8402_8497/1-100 AGTGTTTATGTCAACTACATATTCCCCTAGATACGAATTTGTGA---CCACAAAATTCCT
226470_SINFRUG00000136631_FUGU_7997_8094/1-100 -GTGTATAAATCCACTATATACTCCCCTAGAAGCGAATTTGTGATTGCTTTAATGATTTT
226470_ENSDARG00000013533_ZEBRAFISH_378_474/1-100 AGCCTATACATCAACTACATACTCCCCTAGATACGAATTTGTGA---CTGTGCGTCTGTT
226470_ENSRNOG00000008191_RAT_8416_8511/1-100 AGTGTTTATGTCAACTACATATTCCCCTAGATACGAATTTGTGA---CCACAAAATTCCT
* * ** ** **** *** ********* *********** * * *
226470_ENSG00000182742_HUMAN_8379_8474/1-100 TACACAAATTCGGATCTACAGGGTATATACAGAAGACAG-
226470_ENSMUSG00000038692_MOUSE_8402_8497/1-100 TACACAAATTCGGATCTACAGGGTATATACAGAAGACAG-
226470_SINFRUG00000136631_FUGU_7997_8094/1-100 TACACAAATCCGGATCTACAGGGTAGATATAGAAGACAG-
226470_ENSDARG00000013533_ZEBRAFISH_378_474/1-100 TACACAAATCCGGATCTACAGGGTATATATAGATGACAGG
226470_ENSRNOG00000008191_RAT_8416_8511/1-100 TACACAAATTCGGATCTACAGGGTATATACAGAAGACAG-
********* *************** *** *** *****
226470-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 100
Mean pairwise identity: 84.63
Mean single sequence MFE: -24.58
Consensus MFE: -20.27
Energy contribution: -22.27
Covariance contribution: 2.00
Mean z-score: -3.17
Structure conservation index: 0.82
SVM decision value: 3.08
SVM RNA-class probability: 0.998369
Prediction: RNA
######################################################################
>226470_ENSG00000182742_HUMAN_8379_8474/1-100
AGUGUUUAUGUCAACUACAUAUUCCCCUAGAUACGAAUUUGUGACCACAAAAUUCCUUACACAAAUUCGGAUCUACAGGGUAUAUACAGAAGACAG
........((((..((..((((.(((.(((((.((((((((((................)))))))))).)))))..))).))))..))..)))). ( -26.99)
>226470_ENSMUSG00000038692_MOUSE_8402_8497/1-100
AGUGUUUAUGUCAACUACAUAUUCCCCUAGAUACGAAUUUGUGACCACAAAAUUCCUUACACAAAUUCGGAUCUACAGGGUAUAUACAGAAGACAG
........((((..((..((((.(((.(((((.((((((((((................)))))))))).)))))..))).))))..))..)))). ( -26.99)
>226470_SINFRUG00000136631_FUGU_7997_8094/1-100
GUGUAUAAAUCCACUAUAUACUCCCCUAGAAGCGAAUUUGUGAUUGCUUUAAUGAUUUUUACACAAAUCCGGAUCUACAGGGUAGAUAUAGAAGACAG
.............((((((.((.(((((((..((.(((((((...................))))))).))..)))..)))).))))))))....... ( -20.21)
>226470_ENSDARG00000013533_ZEBRAFISH_378_474/1-100
AGCCUAUACAUCAACUACAUACUCCCCUAGAUACGAAUUUGUGACUGUGCGUCUGUUUACACAAAUCCGGAUCUACAGGGUAUAUAUAGAUGACAGG
..(((...((((...((.((((.((..(((((.((.(((((((.....((....))...))))))).)).)))))..)))))).))..))))..))) ( -21.70)
>226470_ENSRNOG00000008191_RAT_8416_8511/1-100
AGUGUUUAUGUCAACUACAUAUUCCCCUAGAUACGAAUUUGUGACCACAAAAUUCCUUACACAAAUUCGGAUCUACAGGGUAUAUACAGAAGACAG
........((((..((..((((.(((.(((((.((((((((((................)))))))))).)))))..))).))))..))..)))). ( -26.99)
>consensus
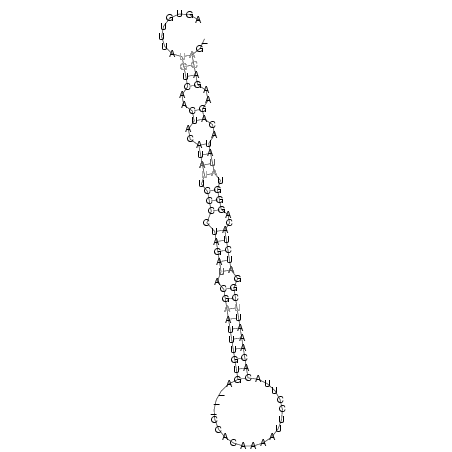
AGUGUUUAUGUCAACUACAUAUUCCCCUAGAUACGAAUUUGUGA___CCACAAAAUUCCUUACACAAAUUCGGAUCUACAGGGUAUAUACAGAAGACAG_
........((((..((..((((.(((.(((((.((((((((((...................)))))))))).)))))..))).))))..))..)))).. (-20.27 = -22.27 + 2.00)
226470-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004