Index >
Results for CNB 223876_5
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
223876_ENSG00000109132_HUMAN_9426_9917/1-521 CAGGA-ATCAATGATAGGGAGGTTGGACAGCTCAGTTCCCCAGTGCCAGCCCAATAGACG
223876_ENSRNOG00000002299_RAT_9393_9879/1-521 CAGGA-ATCAATGATAGGGAGGTTGGACAGCTCAGTTCCCCAGTGCCAGCCCAATAGACG
223876_SINFRUG00000134872_FUGU_9301_9810/1-521 CAGGGGATCAATGATAGGGAGGTTGGACAGCCCGGGCCCCCAGTGCCAGCCCAATAGACT
223876_ENSMUSG00000012520_MOUSE_9442_9928/1-521 CAGGA-ATCAATGATAGGGAGGTTGGACAGCTCAGTTCCCCAGTGCCAGCCCAATAGACG
223876_ENSG00000109132_HUMAN_9426_9917/1-521 GATGAGTTATTGTCATGTAAAAA-GCGCCAGCAATAAGACCAACCGCTTTGCTATTGTCC
223876_ENSRNOG00000002299_RAT_9393_9879/1-521 GATGAGTTATTGTCATGTAAAAA-GCGCCAGCAATGAGACCAACCGCTTTGCTATTGTCC
223876_SINFRUG00000134872_FUGU_9301_9810/1-521 GATGAGTTATTGTCATGTAAAAAAGCGCTAGCAATAAGACCAAACGCTTTGCTATTGTCC
223876_ENSMUSG00000012520_MOUSE_9442_9928/1-521 GATGAGTTATTGTCATGTAAAAA-GCGCCAGCAATAAGACCAACCGCTTTGCTATTGTCC
223876_ENSG00000109132_HUMAN_9426_9917/1-521 AAGTGGAAAGAGCCAAGTTTATTATGAGGACTATATGCTCTAGAGACCTCAGACAAGGCA
223876_ENSRNOG00000002299_RAT_9393_9879/1-521 AAGTGGAAAGAGCCAAGTTTATTATGAGGACTATATACTCTAGAGACCTCAGACAAGGCA
223876_SINFRUG00000134872_FUGU_9301_9810/1-521 AAGCGGAAAGAGCCAAGTTTATTATGAGGACTATATGCTCTAGAGACCTCAGGCTAGGCG
223876_ENSMUSG00000012520_MOUSE_9442_9928/1-521 AAGTGGAAAGAGCCAAGTTTATTATGAGGACTATATACTCTAGAGACCTCAGACAAGGCA
223876_ENSG00000109132_HUMAN_9426_9917/1-521 TCTCATAGGAGGCTTTTTCATAAAACTAGGCTCTGCTGGTAGTAAGGAGGCCAGTTTGGA
223876_ENSRNOG00000002299_RAT_9393_9879/1-521 TCTCACAGGAGGCTTTTTCATAAAACTAGGCTCTGCTGGTAGTAAGGAGGCCAGTTTGGA
223876_SINFRUG00000134872_FUGU_9301_9810/1-521 GCTCTTAGGGGGCTTTT-CATAAAACAGGGCTAGGTTCCTATAAAGGAGGCCAGTTTTGG
223876_ENSMUSG00000012520_MOUSE_9442_9928/1-521 TCTCACAGGAGGCTTTTTCATAAAACTAGGCTCGGCTGGTAGTAAGGAGGCCAGTGTGGA
223876_ENSG00000109132_HUMAN_9426_9917/1-521 GGCAGGCGTTGAGCTGTGCACATCTCCCCAC
223876_ENSRNOG00000002299_RAT_9393_9879/1-521 GGCAGGCGTTGAGCTGTGCACATCTTCCCAC
223876_SINFRUG00000134872_FUGU_9301_9810/1-521 AGCAGGCGCTGAGCAGTGAACATCTACCCA-
223876_ENSMUSG00000012520_MOUSE_9442_9928/1-521 GGCAGGCGTTGAGCAGTGCACATCTCCCCAC
223876_5.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 271
Mean pairwise identity: 92.41
Mean single sequence MFE: -89.57
Consensus MFE: -73.44
Energy contribution: -76.88
Covariance contribution: 3.44
Mean z-score: -1.88
Structure conservation index: 0.82
SVM decision value: 0.56
SVM RNA-class probability: 0.783487
Prediction: RNA
######################################################################
>223876_ENSG00000109132_HUMAN_9426_9917/1-521
CAGGAAUCAAUGAUAGGGAGGUUGGACAGCUCAGUUCCCCAGUGCCAGCCCAAUAGACGGAUGAGUUAUUGUCAUGUAAAAAGCGCCAGCAAUAAGACCAACCGCUUUGCUAUUGUCCAAGUGGAAAGAGCCAAGUUUAUUAUGAGGACUAUAUGCUCUAGAGACCUCAGACAAGGCAUCUCAUAGGAGGCUUUUUCAUAAAACUAGGCUCUGCUGGUAGUAAGGAGGCCAGUUUGGAGGCAGGCGUUGAGCUGUGCACAUCUCCCCAC
...............(((((((((.(((((((((((((..(.(((((((.((((((.(((.((.((((((((..(((.....)))...)))))..))))).))).....))))))(((....))).((((((.(((((....(((((.((.((.....)).)).)))))((.(((((.(((....))).))))).))...))))).))))))))))))).)..))))(((.(((....))).))).))))))))).)).)))))))... ( -92.60)
>223876_ENSRNOG00000002299_RAT_9393_9879/1-521
CAGGAAUCAAUGAUAGGGAGGUUGGACAGCUCAGUUCCCCAGUGCCAGCCCAAUAGACGGAUGAGUUAUUGUCAUGUAAAAAGCGCCAGCAAUGAGACCAACCGCUUUGCUAUUGUCCAAGUGGAAAGAGCCAAGUUUAUUAUGAGGACUAUAUACUCUAGAGACCUCAGACAAGGCAUCUCACAGGAGGCUUUUUCAUAAAACUAGGCUCUGCUGGUAGUAAGGAGGCCAGUUUGGAGGCAGGCGUUGAGCUGUGCACAUCUUCCCAC
...............((((((.((.(((((((((((((..(.(((((((.((((((.(((.((..(((((((..(((.....)))...)))))))...)).))).....))))))(((....))).((((((.(((((....(((((.((.((.....)).)).)))))((.(((((.(((....))).))))).))...))))).))))))))))))).)..))))(((.(((....))).))).))))))))).))...)))))).. ( -91.50)
>223876_SINFRUG00000134872_FUGU_9301_9810/1-521
CAGGGGAUCAAUGAUAGGGAGGUUGGACAGCCCGGGCCCCCAGUGCCAGCCCAAUAGACUGAUGAGUUAUUGUCAUGUAAAAAAGCGCUAGCAAUAAGACCAAACGCUUUGCUAUUGUCCAAGCGGAAAGAGCCAAGUUUAUUAUGAGGACUAUAUGCUCUAGAGACCUCAGGCUAGGCGGCUCUUAGGGGGCUUUUCAUAAAACAGGGCUAGGUUCCUAUAAAGGAGGCCAGUUUUGGAGCAGGCGCUGAGCAGUGAACAUCUACCCA
..(((.....(((...(((.(.((((.....)))).))))((((((((((((.....(((((..(....)..))).))......((....)).......((...(((((.((....))..)))))..(((((((..(((((...(((((.((.((.....)).)).)))))...)))))))))))).)))))))..........(((((((.(((((((....)))).)))))))))).....))))))).........)))...))). ( -84.60)
>223876_ENSMUSG00000012520_MOUSE_9442_9928/1-521
CAGGAAUCAAUGAUAGGGAGGUUGGACAGCUCAGUUCCCCAGUGCCAGCCCAAUAGACGGAUGAGUUAUUGUCAUGUAAAAAGCGCCAGCAAUAAGACCAACCGCUUUGCUAUUGUCCAAGUGGAAAGAGCCAAGUUUAUUAUGAGGACUAUAUACUCUAGAGACCUCAGACAAGGCAUCUCACAGGAGGCUUUUUCAUAAAACUAGGCUCGGCUGGUAGUAAGGAGGCCAGUGUGGAGGCAGGCGUUGAGCAGUGCACAUCUCCCCAC
...............(((((((((.((.((((((((((..(.((((((((((((((.(((.((.((((((((..(((.....)))...)))))..))))).))).....))))))(((....)))..(((((.(((((....(((((.((.((.....)).)).)))))((.(((((.(((....))).))))).))...))))).))))))))))))).)..))))(((.((......)).))).)))))).)).)).)))))))... ( -89.60)
>consensus
CAGGA_AUCAAUGAUAGGGAGGUUGGACAGCUCAGUUCCCCAGUGCCAGCCCAAUAGACGGAUGAGUUAUUGUCAUGUAAAAA_GCGCCAGCAAUAAGACCAACCGCUUUGCUAUUGUCCAAGUGGAAAGAGCCAAGUUUAUUAUGAGGACUAUAUACUCUAGAGACCUCAGACAAGGCAUCUCACAGGAGGCUUUUUCAUAAAACUAGGCUCGGCUGGUAGUAAGGAGGCCAGUUUGGAGGCAGGCGUUGAGCAGUGCACAUCUCCCCAC
................(((((((((.(((((((((((((..(.((((((((((((((.(((.((..(((((((..(((......)))...)))))))...)).))).....))))))(((....)))..(((((.(((((....(((((.((.((.....)).)).)))))((.(((((..(((....)))))))).))...))))).))))))))))))).)..))))(((.((......)).))).))))))))).)).)))))))... (-73.44 = -76.88 + 3.44)
223876_5.rnaz
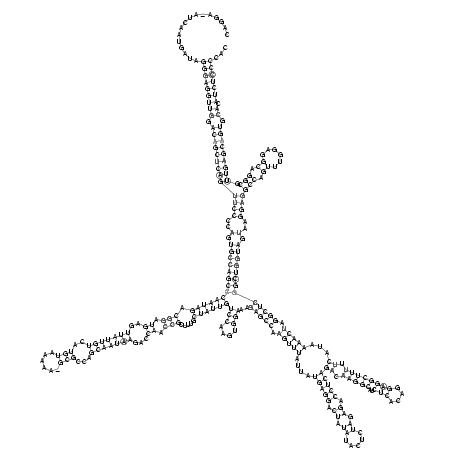
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004