Index >
Results for CNB 204490_3-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 CCCAAACGCCCGCATAGGTGCT---CGGCGGCAAATCCAATGTGGCTCTCAGGAGAAATT
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 ACCATATGCCTGCAGAGGTGGCGGGGGGCAGCAAGCTCTCTGCAGCCCTCCAGAGAAATT
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 ACCATATGCCTGCAGA---NNNNNNNNNCAGCAAGCTCTCTGCAGCCCTCCAGAGAAATT
204490_ENSG00000152120_HUMAN_8890_9516/1-700 ACCATATGCCTGCATAGGTGTTGTGGAACAGCAAATATTCTGCAGCCCTCCAGAGAAATT
*** * *** *** * * **** ** ** *** ********
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 CCTTAATTGTAAATAATTTTACCACATGAGACAATCAAGTCACCTTGAACGCCAACCACC
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 CCTTAATTGTAAATAATTTCACCATGCGACACAATCAAGTCACCTTGAATGCGAA--TCC
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 CCTTAATTGTAAATAATTTCACCATGCGACACAATCAAGTCACCTTGAATGCGAA--TCC
204490_ENSG00000152120_HUMAN_8890_9516/1-700 CCTTAATTGTAAATAATTTCACCATGCGACACAATCAAGTCACCTTGAATGC-AA--ACC
******************* **** ** ******************* ** ** **
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 CCTCCTTACTTCACCGCCGGCCAGCTNNNNNNNNNNNNNNNNNNNNNNNTCCGCTCTTTC
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 CC--------------------------------------------------------TC
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 CC--------------------------------------------------------TC
204490_ENSG00000152120_HUMAN_8890_9516/1-700 CC--------------------------------------------------------TC
** **
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 GGTCTGCAGAGGCAAAGTGTTATTTAAGCTTTACTGGTCTGCGTTAAATTCCGCAATTTG
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 AGCCCGAGCTGGCAAAGTGTTATTTAAGCTTTACTGGGCTGCGTTAAATTCTGCAATTTG
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 GGCCCGCGCAGGCAAAGTGTTATTTAAGCTTTACTGGGCTGCGTTAAATTCTGCAATTTG
204490_ENSG00000152120_HUMAN_8890_9516/1-700 AGCCTGCGGGGGCAAAGTGTTATTTAAGCTTTACTGGGCTGCGTTAAATTCTGCAATTTG
* * * *************************** ************* ********
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 AAGGGCTGTTAAGTTTTTCAATTGAAATTTCATTTAAGATGCAGGTGCTTTTGATTTTAT
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 AAGGGCTGTTAAGTTTTTCAATTGAAATTTCATTTAAAATGCAGGTGCTTTTTATTATAT
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 AAGGGCTGTTAAGTTTTTCAATTGAAATTTCATTTAAAATGCAGGTGCTTTTTATTATAT
204490_ENSG00000152120_HUMAN_8890_9516/1-700 AAGGGCTGTTAAGTTTTTCAATTGAAATTTCATTTAAAATGCAGGTGCTTTTTATTATAT
************************************* ************** *** ***
204490_3-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 78.35
Mean single sequence MFE: -66.18
Consensus MFE: -48.80
Energy contribution: -48.05
Covariance contribution: -0.75
Mean z-score: -1.74
Structure conservation index: 0.74
SVM decision value: 0.68
SVM RNA-class probability: 0.821390
Prediction: RNA
######################################################################
>204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700
CCCAAACGCCCGCAUAGGUGCUCGGCGGCAAAUCCAAUGUGGCUCUCAGGAGAAAUUCCUUAAUUGUAAAUAAUUUUACCACAUGAGACAAUCAAGUCACCUUGAACGCCAACCACCCCUCCUUACUUCACCGCCGGCCAGCUNNNNNNNNNNNNNNNNNNNNNNNUCCGCUCUUUCGGUCUGCAGAGGCAAAGUGUUAUUUAAGCUUUACUGGUCUGCGUUAAAUUCCGCAAUUUGAAGGGCUGUUAAGUUUUUCAAUUGAAAUUUCAUUUAAGAUGCAGGUGCUUUUGAUUUUAU
..((((.((((((((.((((...((((.....((..(((((......(((((...))))).....(((((....))))))))))..))...(((((....))))).))))...))))....((((......(((.((((((............................((.((((........))))))(((((.........))))).)))))).)))..........(((((.((((((((....)))))))))))))..........)))))))).)).)).))))....... ( -68.00)
>204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700
ACCAUAUGCCUGCAGAGGUGGCGGGGGGCAGCAAGCUCUCUGCAGCCCUCCAGAGAAAUUCCUUAAUUGUAAAUAAUUUCACCAUGCGACACAAUCAAGUCACCUUGAAUGCGAAUCCCCUCAGCCCGAGCUGGCAAAGUGUUAUUUAAGCUUUACUGGGCUGCGUUAAAUUCUGCAAUUUGAAGGGCUGUUAAGUUUUUCAAUUGAAAUUUCAUUUAAAAUGCAGGUGCUUUUUAUUAUAU
.......((((((((((((((((((((((.(((.......))).)))))))...((((((..(((....)))..))))))..................))))))))((((((((((...(.(((((((((((...(((......))).)))))....)))))).)....)))).)))..((((((((((....)))))))))).......)))........))))))).............. ( -73.80)
>204490_ENSRNOG00000022194_RAT_7638_8269/1-700
ACCAUAUGCCUGCAGANNNNNNNNNCAGCAAGCUCUCUGCAGCCCUCCAGAGAAAUUCCUUAAUUGUAAAUAAUUUCACCAUGCGACACAAUCAAGUCACCUUGAAUGCGAAUCCCCUCGGCCCGCGCAGGCAAAGUGUUAUUUAAGCUUUACUGGGCUGCGUUAAAUUCUGCAAUUUGAAGGGCUGUUAAGUUUUUCAAUUGAAAUUUCAUUUAAAAUGCAGGUGCUUUUUAUUAUAU
.......(((((((...............(((((....((((((((.((((((((((..(((....)))..))))))....((((...((.(((((....))))).)).((((...(.((((((((....))(((((.........)))))...)))))).)....)))))))).)))).))))))))..)))))(((....))).............))))))).............. ( -57.90)
>204490_ENSG00000152120_HUMAN_8890_9516/1-700
ACCAUAUGCCUGCAUAGGUGUUGUGGAACAGCAAAUAUUCUGCAGCCCUCCAGAGAAAUUCCUUAAUUGUAAAUAAUUUCACCAUGCGACACAAUCAAGUCACCUUGAAUGCAAACCCCUCAGCCUGCGGGGGCAAAGUGUUAUUUAAGCUUUACUGGGCUGCGUUAAAUUCUGCAAUUUGAAGGGCUGUUAAGUUUUUCAAUUGAAAUUUCAUUUAAAAUGCAGGUGCUUUUUAUUAUAU
.((((((((((....))))).)))))...((((........(((((((......((((((..(((....)))..)))))).......(((((..(((((....))))).(((...((((.(.....).)))))))..)))))..............))))))).......((((((..((((((((((....))))))))))(((((......)))))..))))))))))........... ( -65.00)
>consensus
ACCAUAUGCCUGCAGAGGUG_U___GGGCAGCAAACUCUCUGCAGCCCUCCAGAGAAAUUCCUUAAUUGUAAAUAAUUUCACCAUGCGACACAAUCAAGUCACCUUGAAUGCGAA__ACCCC________________________________________________________UCAGCCCGCGCAGGCAAAGUGUUAUUUAAGCUUUACUGGGCUGCGUUAAAUUCUGCAAUUUGAAGGGCUGUUAAGUUUUUCAAUUGAAAUUUCAUUUAAAAUGCAGGUGCUUUUUAUUAUAU
......((((((((..............(((.((......((((((((......((((((..(((....)))..)))))).......(((((..(((((....))))).........................................................................(((......)))...)))))..............)))))))).....)))))(((((.((((((((....)))))))))))))...............))))))))............. (-48.80 = -48.05 + -0.75)
204490_3-rev.rnaz
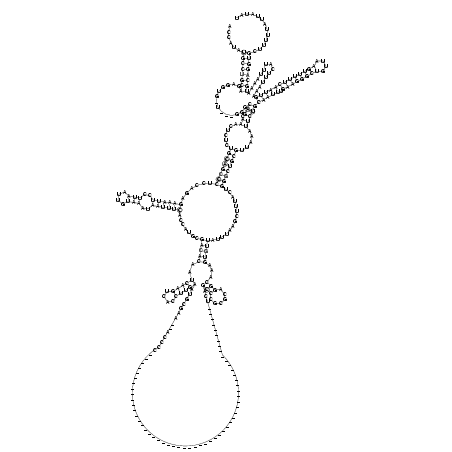
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004