Index >
Results for CNB 200349
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
200349_ENSG00000136630_HUMAN_6634_6848/1-238 -ACATAAGGGCGGATTTGCGTCACCCGAGCAACTTGCCGGTGGAGATAAAGTTGCACAAA
200349_ENSMUSG00000039377_MOUSE_6614_6829/1-238 TACAGAAGAGCGGATTTGCGTCACCACAGCAACTTGCCGGTGGAGATAAAGTTGCACAAA
200349_ENSRNOG00000002309_RAT_6084_6299/1-238 TACAGAAGAGCGGATTTGCGTCACCGCAGCAACTTGCCGGTGGAGATAAAGTTGCACAAA
200349_SINFRUG00000148545_FUGU_4281_4508/1-238 -------GGACGGATTTGCGTCACCGT-GCAACTTGCCGGTCGAGATAAAGGTGCACAAA
* *************** ************* ********* ********
200349_ENSG00000136630_HUMAN_6634_6848/1-238 TATTGAAAG-GGGAAGTGCTAGGAGTCATTATAGAGTTTTTC------------------
200349_ENSMUSG00000039377_MOUSE_6614_6829/1-238 TATTGAAAG-GGGAAGTGCTAGGAGTCATTATAGAGTTTTTC------------------
200349_ENSRNOG00000002309_RAT_6084_6299/1-238 TATTGAAAG-GGGAAGTGCTAGGAGTCATTATAGAGTTTTTC------------------
200349_SINFRUG00000148545_FUGU_4281_4508/1-238 TATTGAAAAAGGGAAGTTCTAACAGTCATTATAAAGTCCACCCCCCACCAGCAGCCCCGC
******** ******* *** ********** *** *
200349_ENSG00000136630_HUMAN_6634_6848/1-238 TCCGGAAGAAATAAGGATTTCTGCAGTATCCTAAAATACTAAGGCCGCTTCTATTTTGAG
200349_ENSMUSG00000039377_MOUSE_6614_6829/1-238 TCCGGAAGAAATAAGGATTTCTGCAGTATCCTAAAATACTAGGACCGCTTCTATTTTGAG
200349_ENSRNOG00000002309_RAT_6084_6299/1-238 ACCGGAAGAAATAAGGATTTCTGCAGTATCCTAAAATACTAGGACCGCTTCTATTTTGAG
200349_SINFRUG00000148545_FUGU_4281_4508/1-238 TCCGGAAGAAATAAGGATTTCTTCTGTATCCTAAAATACTAATACAGGTTCTATTTTGGG
********************* * **************** * * ********** *
200349_ENSG00000136630_HUMAN_6634_6848/1-238 A--CCAATCTCGCAGGCACATCCGCTCATTTAGTCCCGAGTTTGAGCCCATCAAAAA-
200349_ENSMUSG00000039377_MOUSE_6614_6829/1-238 A--CCAATCTCGCAGGCATATCCGCTCATTTAG-CCCAAGTTTGAGCCCATCAAAAAA
200349_ENSRNOG00000002309_RAT_6084_6299/1-238 A--CCAATCTCGCAGGCATATCCGCTCATTTAG-TCCAAGTTTGAGCCCATCAAAAAA
200349_SINFRUG00000148545_FUGU_4281_4508/1-238 GGGCAAATCTAGCAGGAATATCCGCTCATTTAA-CACGAACCCCAGCCCATCAAAAA-
* ***** ***** * ************* * * *************
//
200349.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 238
Mean pairwise identity: 83.97
Mean single sequence MFE: -60.65
Consensus MFE: -41.25
Energy contribution: -42.00
Covariance contribution: 0.75
Mean z-score: -1.54
Structure conservation index: 0.68
SVM decision value: 0.01
SVM RNA-class probability: 0.540494
Prediction: RNA
######################################################################
>200349_ENSG00000136630_HUMAN_6634_6848/1-238
ACAUAAGGGCGGAUUUGCGUCACCCGAGCAACUUGCCGGUGGAGAUAAAGUUGCACAAAUAUUGAAAGGGGAAGUGCUAGGAGUCAUUAUAGAGUUUUUCUCCGGAAGAAAUAAGGAUUUCUGCAGUAUCCUAAAAUACUAAGGCCGCUUCUAUUUUGAGACCAAUCUCGCAGGCACAUCCGCUCAUUUAGUCCCGAGUUUGAGCCCAUCAAAAA
......((((((((.(((..((((.(.((.....)))))))((((((((((.((.....((((.....(((((.(((.(((((((.((((.(((.....)))(....)..)))).)))))))))).).))))..)))).....)).)))).))))))((((....))))....))).)))))))).............(((((.....))))).. ( -59.00)
>200349_ENSMUSG00000039377_MOUSE_6614_6829/1-238
UACAGAAGAGCGGAUUUGCGUCACCACAGCAACUUGCCGGUGGAGAUAAAGUUGCACAAAUAUUGAAAGGGGAAGUGCUAGGAGUCAUUAUAGAGUUUUUCUCCGGAAGAAAUAAGGAUUUCUGCAGUAUCCUAAAAUACUAGGACCGCUUCUAUUUUGAGACCAAUCUCGCAGGCAUAUCCGCUCAUUUAGCCCAAGUUUGAGCCCAUCAAAAAA
.......((((((((.((((((.((((.((.....))..)))).)))....((((.........((((..((((((((.(((((((.((((.(((.....)))(....)..)))).)))))))))....(((((......)))))..)))))).))))((((....))))))))))).))))))))............(((((.....)))))... ( -64.20)
>200349_ENSRNOG00000002309_RAT_6084_6299/1-238
UACAGAAGAGCGGAUUUGCGUCACCGCAGCAACUUGCCGGUGGAGAUAAAGUUGCACAAAUAUUGAAAGGGGAAGUGCUAGGAGUCAUUAUAGAGUUUUUCACCGGAAGAAAUAAGGAUUUCUGCAGUAUCCUAAAAUACUAGGACCGCUUCUAUUUUGAGACCAAUCUCGCAGGCAUAUCCGCUCAUUUAGUCCAAGUUUGAGCCCAUCAAAAAA
.......((((((((.((((((.((((.((.....))..)))).)))....((((.........((((..((((((((.(((((((.((((....((((((....)))))))))).)))))))))....(((((......)))))..)))))).))))((((....))))))))))).))))))))............(((((.....)))))... ( -62.80)
>200349_SINFRUG00000148545_FUGU_4281_4508/1-238
GGACGGAUUUGCGUCACCGUGCAACUUGCCGGUCGAGAUAAAGGUGCACAAAUAUUGAAAAAGGGAAGUUCUAACAGUCAUUAUAAAGUCCACCCCCCACCAGCAGCCCCGCUCCGGAAGAAAUAAGGAUUUCUUCUGUAUCCUAAAAUACUAAUACAGGUUCUAUUUUGGGGGGCAAAUCUAGCAGGAAUAUCCGCUCAUUUAACACGAACCCCAGCCCAUCAAAAA
((((((..((((.((((((.((.....)))))).))......((((.((..(((.(((.....((.....)).....))).)))...)).))))........))))..))).)))(((((((((....)))))))))....(((((((((((......)))...))))))))((((......(((.((.....)))))..........(.....).))))........ ( -56.60)
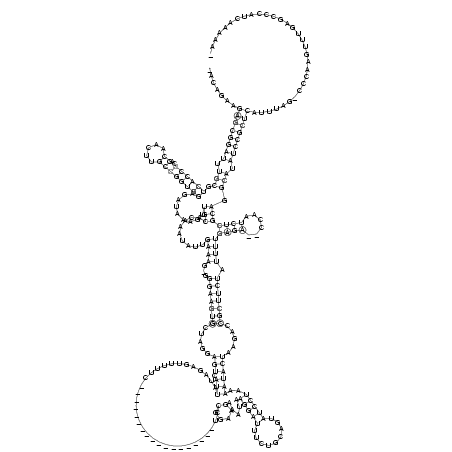
>consensus
_ACAGAAGAGCGGAUUUGCGUCACCGCAGCAACUUGCCGGUGGAGAUAAAGUUGCACAAAUAUUGAAAG_GGGAAGUGCUAGGAGUCAUUAUAGAGUUUUUC__________________UCCGGAAGAAAUAAGGAUUUCUGCAGUAUCCUAAAAUACUAAGACCGCUUCUAUUUUGAGA__CCAAUCUCGCAGGCAUAUCCGCUCAUUUAG_CCCAAGUUUGAGCCCAUCAAAAA_
.......((((((((.(((.((((((..((.....)))))))).........(((.........((((...(((((((.....(((.(((................................(....).....(((((.........)))))..)))))).....))))))).))))((((......))))))).))).))))))))............................... (-41.25 = -42.00 + 0.75)
200349.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004