Index >
Results for CNB 149120-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
149120_ENSG00000103449_HUMAN_9515_9693/1-203 GCAGCGCGCATGTGTCCTGCTATAATTATGATTA----TCAATAATGCAT--TGCGATTA
149120_ENSMUSG00000031665_MOUSE_9663_9841/1-203 ACAGCGCGCATGTGTCCTGCTATAATTATGATTA----TCAATAATGCAT--TGCGATTA
149120_SINFRUG00000130323_FUGU_4948_5148/1-203 -CACCGCGCATGTGCCCCGTTATGATTATCAATAATGCTCTGTGATTAATCATACGAGGG
149120_ENSRNOG00000013907_RAT_9650_9827/1-203 -CAGCGCGCATGTGTCCTGCTATAATTATGATTA----TCAATAATGCAT--TGCGATTA
** ********** ** * *** ***** * ** ** * ** ** * ***
149120_ENSG00000103449_HUMAN_9515_9693/1-203 ATCA----TAGAGGGGCTCTTTGAAAGGCGATTGGCACCGGGCCAGC------GCTATTC
149120_ENSMUSG00000031665_MOUSE_9663_9841/1-203 ATCA----TAGAGGGGCTCTTTGAAAGGCGATTGGCACCGGGCCAGC------GCTATTC
149120_SINFRUG00000130323_FUGU_4948_5148/1-203 AGGAGGAGGAGGGGGGTTCTTTGAAAGGCGATTGGCAATTCGCCAGCCCCCCTCCTATTC
149120_ENSRNOG00000013907_RAT_9650_9827/1-203 ATCA----TAGAGGGGCTCTTTGAAAGGCGATTGGCATCGGGCCAGC------GCTATTC
* * ** **** ******************** ****** ******
149120_ENSG00000103449_HUMAN_9515_9693/1-203 AAACCCGCTCGCCTTAATCAATTAGTTCGTGATTTGCTGCAGACCCCTGTCTCTC-CGC-
149120_ENSMUSG00000031665_MOUSE_9663_9841/1-203 AAACCCGCTCGCCTTAATCAATTAGTTCGTGATTTGCTGCAGACCCCTGTCTCTC-CGC-
149120_SINFRUG00000130323_FUGU_4948_5148/1-203 AAACGAAGTCTCTTTAATCAATTAGCTCCTGATTTGCTGGGGACTCTTGTCTCTCTCTCA
149120_ENSRNOG00000013907_RAT_9650_9827/1-203 AAACCCGCTCGCCTTAATCAATTAGTTCGTGATTTGCTGCAGACCCCTGTCTCTC-CGC-
**** ** * ************ ** ********** *** * ******** * *
149120_ENSG00000103449_HUMAN_9515_9693/1-203 GCGCTGGCCCAATAAG------C
149120_ENSMUSG00000031665_MOUSE_9663_9841/1-203 GCGCTGGCCCAATAAG------C
149120_SINFRUG00000130323_FUGU_4948_5148/1-203 GCTCCTACCCAATGAGTTGATC-
149120_ENSRNOG00000013907_RAT_9650_9827/1-203 GCGCTGGCCCAATAAG------C
** * ****** **
149120-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 203
Mean pairwise identity: 81.31
Mean single sequence MFE: -61.32
Consensus MFE: -36.45
Energy contribution: -37.26
Covariance contribution: 0.81
Mean z-score: -2.30
Structure conservation index: 0.59
SVM decision value: 0.80
SVM RNA-class probability: 0.854045
Prediction: RNA
######################################################################
>149120_ENSG00000103449_HUMAN_9515_9693/1-203
GCAGCGCGCAUGUGUCCUGCUAUAAUUAUGAUUAUCAAUAAUGCAUUGCGAUUAAUCAUAGAGGGGCUCUUUGAAAGGCGAUUGGCACCGGGCCAGCGCUAUUCAAACCCGCUCGCCUUAAUCAAUUAGUUCGUGAUUUGCUGCAGACCCCUGUCUCUCCGCGCGCUGGCCCAAUAAGC
.((((((((........(((...((((((((...........((...))((((((.....((((((...((((((.((((.(((((.....))))))))).))))))))).)))...)))))).......))))))))....)))(((....))).....))))))))........... ( -57.70)
>149120_ENSMUSG00000031665_MOUSE_9663_9841/1-203
ACAGCGCGCAUGUGUCCUGCUAUAAUUAUGAUUAUCAAUAAUGCAUUGCGAUUAAUCAUAGAGGGGCUCUUUGAAAGGCGAUUGGCACCGGGCCAGCGCUAUUCAAACCCGCUCGCCUUAAUCAAUUAGUUCGUGAUUUGCUGCAGACCCCUGUCUCUCCGCGCGCUGGCCCAAUAAGC
.((((((((........(((...((((((((...........((...))((((((.....((((((...((((((.((((.(((((.....))))))))).))))))))).)))...)))))).......))))))))....)))(((....))).....))))))))........... ( -58.00)
>149120_SINFRUG00000130323_FUGU_4948_5148/1-203
CACCGCGCAUGUGCCCCGUUAUGAUUAUCAAUAAUGCUCUGUGAUUAAUCAUACGAGGGAGGAGGAGGAGGGGGGUUCUUUGAAAGGCGAUUGGCAAUUCGCCAGCCCCCCUCCUAUUCAAACGAAGUCUCUUUAAUCAAUUAGCUCCUGAUUUGCUGGGGACUCUUGUCUCUCUCUCAGCUCCUACCCAAUGAGUUGAUC
....(((....)))...((.(((((((...(((......)))...)))))))))((((((((((.((((((((((((........(((((........))))))))))))))))).)))..(((((((((((..(((((.........)))))....))))))).)))))))))))(((((((.........))))))).. ( -72.10)
>149120_ENSRNOG00000013907_RAT_9650_9827/1-203
CAGCGCGCAUGUGUCCUGCUAUAAUUAUGAUUAUCAAUAAUGCAUUGCGAUUAAUCAUAGAGGGGCUCUUUGAAAGGCGAUUGGCAUCGGGCCAGCGCUAUUCAAACCCGCUCGCCUUAAUCAAUUAGUUCGUGAUUUGCUGCAGACCCCUGUCUCUCCGCGCGCUGGCCCAAUAAGC
((((((((........(((...((((((((...........((...))((((((.....((((((...((((((.((((.(((((.....))))))))).))))))))).)))...)))))).......))))))))....)))(((....))).....))))))))........... ( -57.50)
>consensus
_CAGCGCGCAUGUGUCCUGCUAUAAUUAUGAUUA____UCAAUAAUGCAU__UGCGAUUAAUCA____UAGAGGGGCUCUUUGAAAGGCGAUUGGCACCGGGCCAGC______GCUAUUCAAACCCGCUCGCCUUAAUCAAUUAGUUCGUGAUUUGCUGCAGACCCCUGUCUCUC_CGC_GCGCUGGCCCAAUAAG______C
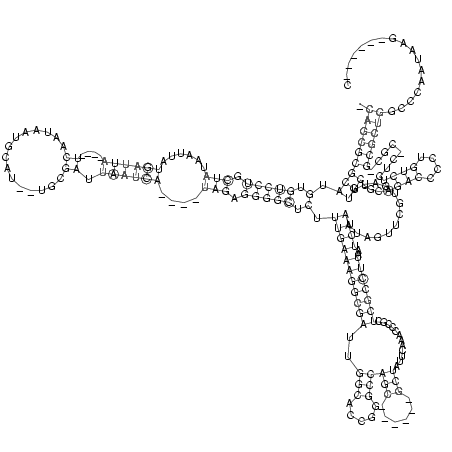
.(((((((((.(.(((((.(((......((((((....((...............)).))))))....))).))))).).(((((((((((..(((.....)))(((......)))............)))))))..)))).............)))...((((....))))........))))))................. (-36.45 = -37.26 + 0.81)
149120-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004