Index >
Results for CNB 136553
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
136553_ENSG00000126773_HUMAN_3649_3745/1-97 ACACTACTCTCTTCTCCCTTACTCCCTCTTTTCACCCTTCCTGTGTTCTTGGTGGGGTTT
136553_ENSDARG00000008933_ZEBRAFISH_6049_6143/1-97 -CTCTTCTCTCTGCCCCTCTCCTTCCACTATTCACACTACCGGTGTTCCTGGTGGGATTT
136553_ENSMUSG00000034501_MOUSE_3154_3250/1-97 ACACTACTGTCATCTCCTCTACTGCCCCTCTTCACACTTCCTGTTTTCTTGGTGGGGTTT
136553_ENSRNOG00000005568_RAT_1822_1918/1-97 ACAGTGCTGTCCTCTCCTCTACTGCCTCTCTTCACACTGCCTGTTTTCTTGGTGGGGTTC
* * ** ** * ** * ** ** ** ***** ** ** ** *** ******* **
136553_ENSG00000126773_HUMAN_3649_3745/1-97 CCCCGACCTATTCAGAGTTGGCCAGGAGCAGCAGGCA
136553_ENSDARG00000008933_ZEBRAFISH_6049_6143/1-97 CCCAGGCCGCTGCGCTCATGGCCAGGCACTCCTGGC-
136553_ENSMUSG00000034501_MOUSE_3154_3250/1-97 CCTCGTCCAGTTCAGAGCTGGCCAGGAGCAGTGGGCA
136553_ENSRNOG00000005568_RAT_1822_1918/1-97 CCTCGTCCAGTTCAGAGCTGGCCAGGAGCAGTGGGCA
** * ** * * ******** * ***
//
136553.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 97
Mean pairwise identity: 75.43
Mean single sequence MFE: -27.75
Consensus MFE: -18.73
Energy contribution: -18.72
Covariance contribution: -0.00
Mean z-score: -1.60
Structure conservation index: 0.67
SVM decision value: 0.36
SVM RNA-class probability: 0.704306
Prediction: RNA
######################################################################
>136553_ENSG00000126773_HUMAN_3649_3745/1-97
ACACUACUCUCUUCUCCCUUACUCCCUCUUUUCACCCUUCCUGUGUUCUUGGUGGGGUUUCCCCGACCUAUUCAGAGUUGGCCAGGAGCAGCAGGCA
.......................................((((((((((((((((((...))))(((((....)).))).)))))))))).)))).. ( -26.80)
>136553_ENSDARG00000008933_ZEBRAFISH_6049_6143/1-97
CUCUUCUCUCUGCCCCUCUCCUUCCACUAUUCACACUACCGGUGUUCCUGGUGGGAUUUCCCAGGCCGCUGCGCUCAUGGCCAGGCACUCCUGGC
..........((((.......((((((((...((((.....))))...)))))))).......(((((.((....))))))).))))........ ( -26.20)
>136553_ENSMUSG00000034501_MOUSE_3154_3250/1-97
ACACUACUGUCAUCUCCUCUACUGCCCCUCUUCACACUUCCUGUUUUCUUGGUGGGGUUUCCUCGUCCAGUUCAGAGCUGGCCAGGAGCAGUGGGCA
...(((((((.............(((((.((....((.....))......)).))))).((((.(.(((((.....)))))).)))))))))))... ( -29.90)
>136553_ENSRNOG00000005568_RAT_1822_1918/1-97
ACAGUGCUGUCCUCUCCUCUACUGCCUCUCUUCACACUGCCUGUUUUCUUGGUGGGGUUCCCUCGUCCAGUUCAGAGCUGGCCAGGAGCAGUGGGCA
........((((.((.((((...(((.((((..((((.....)).....((((((((...))))).)))))..))))..)))..)))).)).)))). ( -28.10)
>consensus
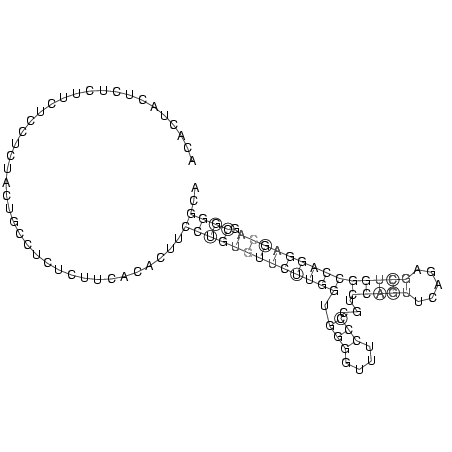
ACACUACUCUCUUCUCCUCUACUGCCUCUCUUCACACUUCCUGUGUUCUUGGUGGGGUUUCCCCGUCCAGUUCAGAGCUGGCCAGGAGCAGCGGGCA
.......................................(((((((((((((.(((....)))...(((((.....)))))))))))))).)))).. (-18.73 = -18.72 + -0.00)
136553.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004