Index >
Results for CNB 134297_9
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 -AGGGACGTTTTAATTGTCCGGATTAAGAAGGATACAATTTTCTTAGGATCAGAGGGGAG
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 -AGGGAAGTTTTAATTGTCCGGATTAAGAAGGATACAATTTTCCGAGGATCAGAGGGACG
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 GAGGAAACTTTTAATTGTG-GGATTA-GAAGGAGAGAGAATTTGTGGGATCAGAGGAAAA
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 CGGGAAAGTTTTAATTGCA-TGATTA-GCTG-AGCGGGA-TTAGAGGGA-CATTTGCCTG
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 -AGGGACGTTTTAATTGTCCGGATTAAGAAGGATACAATTTTCTTAGGATCAGAGGGGAG
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 CTTTGAAAAG-TAGTTTCCT-CAAAAAATCAAATCAAAGGTTTCGGTTCTGGAAAACATC
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 TTTTGAAAAG-TAGTTTCCT-CATAAAATCAAATCAAAGGTTCCAGTTGTTGAAAACATC
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 TTTCCAGGCA-GAGTTTCTTGCTTAAAATCGAATCAAAGGATTTGCGAGCTTAAAATGGC
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 GTGGGATAGCCTAGATTTCC-ACTAAAA-CAC--CAAACTATGTGCTTGAGTGAAACACA
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 CTTTGAAAAG-TAGTTTCCT-CAAAAAATCAAATCAAAGGTTTTGGTTCTGGAAAACATC
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 AAC----------AAAATCTCTTGGCTGGTTAATTGGAATTGCATCCAGTTCAGCAGCGG
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 AAC----------AAAATCTCTTTGCTGGTTAATTGGAATCTCATCCAGTTCAGCAGCGG
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 AACTGGCTTTTTCAACCCCCCCTCAATGTTTAATTGGAACTGCATCTAGTTCACC-----
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 AACACTGAAAGG-AAAATCATTTTACACTTTAATTGGTTCCGTTTGTGGATCA-------
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 AAC----------AAAATCTCTTGGCTGGTTAATTGGAATTGCATCCAGTTCAGCAGCGG
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 ATCAAATCACCTCCAACAATTAATTTAGATTTAATTCTGTAATTATCAGCAAATCGAGTA
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 ATCAAATCACCTCCAACAATTAATTTAGATTTAATTCTGTAATTATCAGCAAATCGAGTA
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 --CAAATCATCCAAAACAATTAATTTTGATTTAATTCTATAATTATCATCAAATCGAATA
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 ----AATGACTTTCAACAATTAATTTACGTTTCACACTCTAATTATCTGTACATCGACTA
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 ATCAAATCACCTCCAACAATTAATTTAGATTTAATTCTGTAATTATCAGCAAATCGAGTA
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 ATGTGCAAGTTAATTTAGATAGTTAAAAATTAACTTGCGTGAAGTTAATTGAGTAATTAA
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 ATGCGCAAGTTAATTTAGATAGTTAAAAATTAACTTGCGTGAAGTTAATTGAGTAATTAA
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 ATGTGCAACCTAATTTAGATAGTTAAAAATTAACGTGCGTGAAGTTAATTGAGTAATTAA
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 ATGTGCAGCTCAGTTATGATAGCTACAAATGAATTCACGTTACGTTAATTCAGCAATTAA
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 ATGTGCAAGTTAATTTAGATAGTTAAAAATTAACTTGCGTGAAGTTAATTGAGTAATTAA
134297_9.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 73.60
Mean single sequence MFE: -69.16
Consensus MFE: -25.33
Energy contribution: -26.09
Covariance contribution: 0.76
Mean z-score: -1.91
Structure conservation index: 0.37
SVM decision value: 0.68
SVM RNA-class probability: 0.822028
Prediction: RNA
######################################################################
>134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004
AGGGACGUUUUAAUUGUCCGGAUUAAGAAGGAUACAAUUUUCUUAGGAUCAGAGGGGAGCUUUGAAAAGUAGUUUCCUCAAAAAAUCAAAUCAAAGGUUUCGGUUCUGGAAAACAUCAACAAAAUCUCUUGGCUGGUUAAUUGGAAUUGCAUCCAGUUCAGCAGCGGAUCAAAUCACCUCCAACAAUUAAUUUAGAUUUAAUUCUGUAAUUAUCAGCAAAUCGAGUAAUGUGCAAGUUAAUUUAGAUAGUUAAAAAUUAACUUGCGUGAAGUUAAUUGAGUAAUUAA
..(((((.......)))))((((((((((((......)))))))))((((.((((..(((((....))))..)..))))............(((((((((..(((.((.....))..))).)))))).)))((((.(.(((((((......))))))).).)))).))))........)))..((((((((((.(((((....(((.......))).))))).........((((((((((((..........))))))))))))...))))))))))......... ( -69.50)
>134297_ENSG00000164853_HUMAN_5679_7549/1-2004
AGGGAAGUUUUAAUUGUCCGGAUUAAGAAGGAUACAAUUUUCCGAGGAUCAGAGGGACGUUUUGAAAAGUAGUUUCCUCAUAAAAUCAAAUCAAAGGUUCCAGUUGUUGAAAACAUCAACAAAAUCUCUUUGCUGGUUAAUUGGAAUCUCAUCCAGUUCAGCAGCGGAUCAAAUCACCUCCAACAAUUAAUUUAGAUUUAAUUCUGUAAUUAUCAGCAAAUCGAGUAAUGCGCAAGUUAAUUUAGAUAGUUAAAAAUUAACUUGCGUGAAGUUAAUUGAGUAAUUAA
.((((((((.....(((((..........))))).))))))))((((....(((((((...((....))..).))))))................(((((...(((((((.....))))))).........((((.(.(((((((......))))))).).)))))))))......))))...((((((((((.(((((....(((.......))).))))).......((((((((((((((..........)))))))))))))).))))))))))......... ( -74.50)
>134297_SINFRUG00000149320_FUGU_3658_5564/1-2004
GAGGAAACUUUUAAUUGUGGGAUUAGAAGGAGAGAGAAUUUGUGGGAUCAGAGGAAAAUUUCCAGGCAGAGUUUCUUGCUUAAAAUCGAAUCAAAGGAUUUGCGAGCUUAAAAUGGCAACUGGCUUUUUCAACCCCCCCUCAAUGUUUAAUUGGAACUGCAUCUAGUUCACCCAAAUCAUCCAAAACAAUUAAUUUUGAUUUAAUUCUAUAAUUAUCAUCAAAUCGAAUAAUGUGCAACCUAAUUUAGAUAGUUAAAAAUUAACGUGCGUGAAGUUAAUUGAGUAAUUAA
(((....))).((((((((((((((((.(((((..(((((((.(((.......(((((...((((((...((((...((((.(((((.........)))))..))))...)))).))..))))..)))))......))).))).)))).....((((((....)))))).......)).)))....(((......))).))))))))))))))))........((((.((((.((((...((((((..........))))))...))))....)))).))))........ ( -58.72)
>134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004
CGGGAAAGUUUUAAUUGCAUGAUUAGCUGAGCGGGAUUAGAGGGACAUUUGCCUGGUGGGAUAGCCUAGAUUUCCACUAAAACACCAAACUAUGUGCUUGAGUGAAACACAAACACUGAAAGGAAAAUCAUUUUACACUUUAAUUGGUUCCGUUUGUGGAUCAAAUGACUUUCAACAAUUAAUUUACGUUUCACACUCUAAUUAUCUGUACAUCGACUAAUGUGCAGCUCAGUUAUGAUAGCUACAAAUGAAUUCACGUUACGUUAAUUCAGCAAUUAA
..........((((((((.....(((((((((..(((((((((.((((.....(((((((....))(((.......)))...)))))....)))).)....(((((((........((((((...(((((.((........)).))))).((((((.....)))))).))))))..((.....))..))))))).))))))))...(((((((......)))))))))))))))).............((((((.(((...))).)))))))))))))) ( -69.80)
>134297_ENSRNOG00000001284_RAT_5363_7190/1-2004
AGGGACGUUUUAAUUGUCCGGAUUAAGAAGGAUACAAUUUUCUUAGGAUCAGAGGGGAGCUUUGAAAAGUAGUUUCCUCAAAAAAUCAAAUCAAAGGUUUUGGUUCUGGAAAACAUCAACAAAAUCUCUUGGCUGGUUAAUUGGAAUUGCAUCCAGUUCAGCAGCGGAUCAAAUCACCUCCAACAAUUAAUUUAGAUUUAAUUCUGUAAUUAUCAGCAAAUCGAGUAAUGUGCAAGUUAAUUUAGAUAGUUAAAAAUUAACUUGCGUGAAGUUAAUUGAGUAAUUAA
..(((((.......)))))((((((((((((......)))))))))((((.((((..(((((....))))..)..))))............(((((((((((....((.....))....)))))))).)))((((.(.(((((((......))))))).).)))).))))........)))..((((((((((.(((((....(((.......))).))))).........((((((((((((..........))))))))))))...))))))))))......... ( -73.30)
>consensus
_AGGGAAGUUUUAAUUGUCCGGAUUAAGAAGGAUACAAUUUUCGUAGGAUCAGAGGGAAGCUUUGAAAAG_UAGUUUCCU_CAUAAAAUCAAAUCAAAGGUUUUGGUUGUGGAAAACAUCAAC__________AAAAUCUCUUGGCUGGUUAAUUGGAAUUGCAUCCAGUUCAGCAGCGGAUCAAAUCACCUCCAACAAUUAAUUUAGAUUUAAUUCUGUAAUUAUCAGCAAAUCGAGUAAUGUGCAAGUUAAUUUAGAUAGUUAAAAAUUAACUUGCGUGAAGUUAAUUGAGUAAUUAA
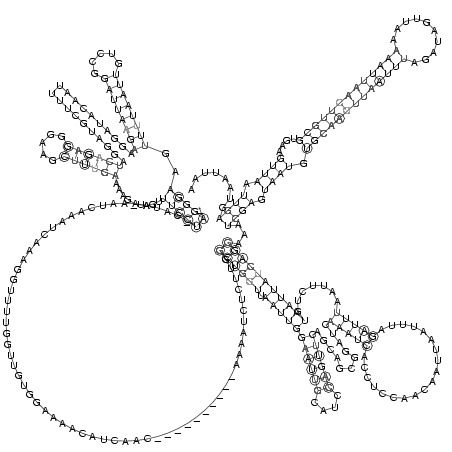
.(((((..((((((((.....))))))))....................((((((.....)))))).........)))))................................................................((((((.(((((((((((....))))))...........(((((...................))))).......)))))))))))...((((.((((.(((((((((((((..........)))))))))))))....)))).))))........ (-25.33 = -26.09 + 0.76)
134297_9.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004