Index >
Results for CNB 134297_9-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 TTAATTACTCAATTAACTTCACGCAAGTTAATTTTTAACTATCTAAATTAACTTGCACAT
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 TTAATTACTCAATTAACTTCACGCAAGTTAATTTTTAACTATCTAAATTAACTTGCGCAT
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 TTAATTACTCAATTAACTTCACGCACGTTAATTTTTAACTATCTAAATTAGGTTGCACAT
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 TTAATTGCTGAATTAACGTAACGTGAATTCATTTGTAGCTATCATAACTGAGCTGCACAT
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 TTAATTACTCAATTAACTTCACGCAAGTTAATTTTTAACTATCTAAATTAACTTGCACAT
****** ** ******* * *** ** **** ** ***** ** * *** ***
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 TACTCGATTTGCTGATAATTACAGAATTAAATCTAAATTAATTGTTGGAGGTGATTTGAT
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 TACTCGATTTGCTGATAATTACAGAATTAAATCTAAATTAATTGTTGGAGGTGATTTGAT
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 TATTCGATTTGATGATAATTATAGAATTAAATCAAAATTAATTGTTTTGGATGATTTG--
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 TAGTCGATGTACAGATAATTAGAGTGTGAAACGTAAATTAATTGTTGAAAGTCATT----
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 TACTCGATTTGCTGATAATTACAGAATTAAATCTAAATTAATTGTTGGAGGTGATTTGAT
** ***** * ******** ** * *** ************ * ***
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 CCGCTGCTGAACTGGATGCAATTCCAATTAACCAGCCAAGAGATTTT----------GTT
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 CCGCTGCTGAACTGGATGAGATTCCAATTAACCAGCAAAGAGATTTT----------GTT
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 -----GGTGAACTAGATGCAGTTCCAATTAAACATTGAGGGGGGGTTGAAAAAGCCAGTT
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 -------TGATCCACAAACGGAACCAATTAAAGTGTAAAATGATTTT-CCTTTCAGTGTT
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 CCGCTGCTGAACTGGATGCAATTCCAATTAACCAGCCAAGAGATTTT----------GTT
*** * * ******** * * ** ***
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 GATGTTTTCCAGAACCGAAACCTTTGATTTGATTTTTTG-AGGAAACTA-CTTTTCAAAG
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 GATGTTTTCAACAACTGGAACCTTTGATTTGATTTTATG-AGGAAACTA-CTTTTCAAAA
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 GCCATTTTAAGCTCGCAAATCCTTTGATTCGATTTTAAGCAAGAAACTC-TGCCTGGAAA
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 TGTGTTTCACTCAAGCACATAGTTTG--GTG-TTTTAGT-GGAAATCTAGGCTATCCCAC
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 GATGTTTTCCAGAACCAAAACCTTTGATTTGATTTTTTG-AGGAAACTA-CTTTTCAAAG
*** * **** * **** ** ** * *
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 CTCCCCTCTGATCCTAAGAAAATTGTATCCTTCTTAATCCGGACAATTAAAACGTCCCT-
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 CGTCCCTCTGATCCTCGGAAAATTGTATCCTTCTTAATCCGGACAATTAAAACTTCCCT-
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 TTTTCCTCTGATCCCACAAATTCTCTCTCCTTC-TAATCC-CACAATTAAAAGTTTCCTC
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 CAGGCAAATG-TCCCTCTAA-TCCCGCT-CAGC-TAATCA-TGCAATTAAAACTTTCCCG
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 CTCCCCTCTGATCCTAAGAAAATTGTATCCTTCTTAATCCGGACAATTAAAACGTCCCT-
* ** *** ** * * * ***** ********* * **
134297_9-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 73.60
Mean single sequence MFE: -61.90
Consensus MFE: -23.12
Energy contribution: -25.08
Covariance contribution: 1.96
Mean z-score: -1.76
Structure conservation index: 0.37
SVM decision value: 0.15
SVM RNA-class probability: 0.605648
Prediction: RNA
######################################################################
>134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004
UUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUAACCAGCCAAGAGAUUUUGUUGAUGUUUUCCAGAACCGAAACCUUUGAUUUGAUUUUUUGAGGAAACUACUUUUCAAAGCUCCCCUCUGAUCCUAAGAAAAUUGUAUCCUUCUUAAUCCGGACAAUUAAAACGUCCCU
.........(((((((......((((((((((((..........)))))))))))).........(((((((((.......)))...))))))....)))))))(..((((.(((((((...((((...((.((((......)))).))...))))(((((((((..((..(.(((((........)))))..)..))..)))))))))((....)).....)))))..)).))))..)...((((((..........))))))....((((.........)))).. ( -61.30)
>134297_ENSG00000164853_HUMAN_5679_7549/1-2004
UUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCGCAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGAGAUUCCAAUUAACCAGCAAAGAGAUUUUGUUGAUGUUUUCAACAACUGGAACCUUUGAUUUGAUUUUAUGAGGAAACUACUUUUCAAAACGUCCCUCUGAUCCUCGGAAAAUUGUAUCCUUCUUAAUCCGGACAAUUAAAACUUCCCU
.........(((((((.....(((((((((((((..........)))))))))))))........(((((((((.......)))...))))))....)))))))..((((((...(((.(((((((...((.((((......)))).))...)))).(((((...(((((((.....)))))))..(((........(((((.......((....)).....)))))(((....((((.....))))....))).)))))))).....)))))).....)))))).. ( -65.60)
>134297_SINFRUG00000149320_FUGU_3658_5564/1-2004
UUAAUUACUCAAUUAACUUCACGCACGUUAAUUUUUAACUAUCUAAAUUAGGUUGCACAUUAUUCGAUUUGAUGAUAAUUAUAGAAUUAAAUCAAAAUUAAUUGUUUUGGAUGAUUUGGGUGAACUAGAUGCAGUUCCAAUUAAACAUUGAGGGGGGGUUGAAAAAGCCAGUUGCCAUUUUAAGCUCGCAAAUCCUUUGAUUCGAUUUUAAGCAAGAAACUCUGCCUGGAAAUUUUCCUCUGAUCCCACAAAUUCUCUCUCCUUCUAAUCCCACAAUUAAAAGUUUCCUC
..........(((((((.........)))))))................(((..((.........((((((((..((....))..))))))))....((((((((...((((.....(((.(((((......))))))...........((((((((((((((((..((((........(((((.(((..((((....))))))).)))))(((........)))))))...)))))....)))))).......))))).)).....)))).))))))))..))..))). ( -52.31)
>134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004
UUAAUUGCUGAAUUAACGUAACGUGAAUUCAUUUGUAGCUAUCAUAACUGAGCUGCACAUUAGUCGAUGUACAGAUAAUUAGAGUGUGAAACGUAAAUUAAUUGUUGAAAGUCAUUUGAUCCACAAACGGAACCAAUUAAAGUGUAAAAUGAUUUUCCUUUCAGUGUUUGUGUUUCACUCAAGCACAUAGUUUGGUGUUUUAGUGGAAAUCUAGGCUAUCCCACCAGGCAAAUGUCCCUCUAAUCCCGCUCAGCUAAUCAUGCAAUUAAAACUUUCCCG
((((((((((((((.(((...))).))))))................((((((((.(((((....))))).))....(((((((.(((((((((((((..((((..(((((((((((..(((......)))((........))...)))))))))))....)))))))))))))))))......((((.((((((((...((((((....))..))))...))))))))..))))..)))))))...))))))........)))))))).......... ( -69.40)
>134297_ENSRNOG00000001284_RAT_5363_7190/1-2004
UUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUAACCAGCCAAGAGAUUUUGUUGAUGUUUUCCAGAACCAAAACCUUUGAUUUGAUUUUUUGAGGAAACUACUUUUCAAAGCUCCCCUCUGAUCCUAAGAAAAUUGUAUCCUUCUUAAUCCGGACAAUUAAAACGUCCCU
.........(((((((......((((((((((((..........)))))))))))).........(((((((((.......)))...))))))....)))))))(..((((.(((((((...((((...((.((((......)))).))...))))(((((((((..((..(.(((((........)))))..)..))..)))))))))((....)).....)))))..)).))))..)...((((((..........))))))....((((.........)))).. ( -60.90)
>consensus
UUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUAACCAGCAAAGAGAUUUU__________GUUGAUGUUUUCCACAACCAAAACCUUUGAUUUGAUUUUAUG_AGGAAACUA_CUUUUCAAAACUCCCCUCUGAUCCUAAGAAAAUUGUAUCCUUCUUAAUCCGGACAAUUAAAACUUCCCU_
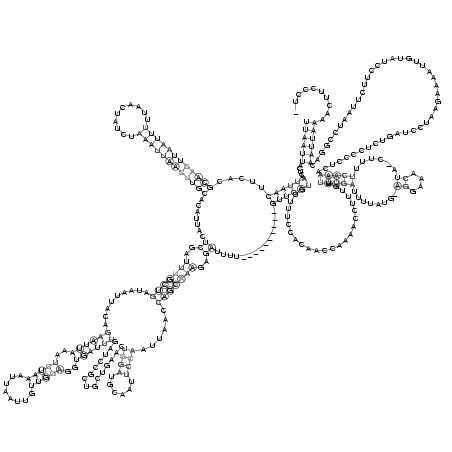
((((((.....((((((.....((((((((((((..........)))))))))))).......((..(((((((.........((((((..(((((........)))))..))))))...............((((......))))......)))))))..))..............))))))........................(((((........((....))......))))).............................................)))))).......... (-23.12 = -25.08 + 1.96)
134297_9-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004